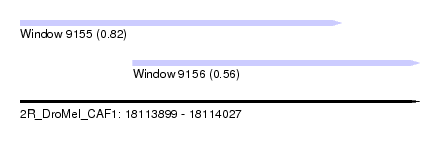
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 18,113,899 – 18,114,027 |
| Length | 128 |
| Max. P | 0.815507 |

| Location | 18,113,899 – 18,114,002 |
|---|---|
| Length | 103 |
| Sequences | 6 |
| Columns | 108 |
| Reading direction | forward |
| Mean pairwise identity | 79.22 |
| Mean single sequence MFE | -33.91 |
| Consensus MFE | -23.12 |
| Energy contribution | -25.82 |
| Covariance contribution | 2.70 |
| Combinations/Pair | 1.13 |
| Mean z-score | -1.63 |
| Structure conservation index | 0.68 |
| SVM decision value | 0.66 |
| SVM RNA-class probability | 0.815507 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
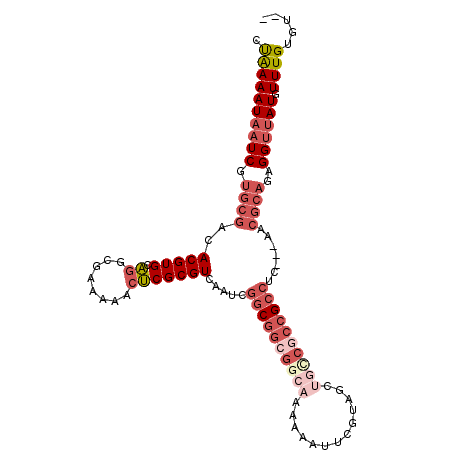
>2R_DroMel_CAF1 18113899 103 + 20766785 CUAAAAUAAUCGUGCGACACGUGCAGGCGAAAAACUCGCGUCAAUCGGCGGCGGCAAAAAAUUCGUAGCUGCCGCCGCCUC---AACGCAGAGGUUAUGUUUGUGU-- .....((((((.((((..(((((.((........))))))).....((((((((((.............))))))))))..---..))))..))))))........-- ( -38.12) >DroSec_CAF1 65225 97 + 1 CUAAAAUAAUCGUGCGACACGUGCAGGCGAAAAACUCGCGUCAAUCGGCGGCGGCAAAAAAUUCGUAG------CCGCCUA---AACGCAGAGGUUAUGUUUGUGU-- .....((((((.((((..(((((.((........))))))).....((((((.((.........)).)------)))))..---..))))..))))))........-- ( -29.70) >DroSim_CAF1 66827 103 + 1 CUAAAAUAAUCGUGCGACACGUGCAGGCGAAAAACUCGCGUCAAUCGGCGGCGGCAAAAAAUUCGUAGCUGCCGCCGCCUC---AACGCAGAGGUUAUGUUUGUGU-- .....((((((.((((..(((((.((........))))))).....((((((((((.............))))))))))..---..))))..))))))........-- ( -38.12) >DroEre_CAF1 64153 102 + 1 GUAAAAUAAUCGUGCGACACGUGCAGACAAAAAACUCGCGUCAAUCGACGGCGGCAAAAA-UUCGCUGAUGCCGCCGCCUC---AUCGCAGAGGUUAUGUUUGUGU-- ..........((((...)))).(((((((..........(((....)))(((((((....-........)))))))(((((---......)))))..)))))))..-- ( -30.80) >DroYak_CAF1 66001 107 + 1 CUAAAAUAAUCGUGCGACACGUGCAGGCGAAAAACUCGCGUCAAACGGCGGCGGCAAAAA-AUCGCUGCUGUCGCCGCCUCAUCAUCGCAGAGGUUAUGUUUGUGUGC .....((((((.(((((...(((.((((((.....)).........((((((((((....-.....)))))))))))))))))..)))))..)))))).......... ( -37.50) >DroPer_CAF1 67169 97 + 1 ACGAAAUUUUCG-----CACGUGUCGCACAGAAAAGCGCGUCAAAUCGCCCAGUCGACGCGCUC-UGCCGACCGCCGCUUC---GGUGCUGAGGUUAUGUUUGGGU-- (((.((((((.(-----(.((.((((..((((...(((((((.............)))))))))-)).))))))..((...---.)))).)))))).)))......-- ( -29.22) >consensus CUAAAAUAAUCGUGCGACACGUGCAGGCGAAAAACUCGCGUCAAUCGGCGGCGGCAAAAAAUUCGUAGCUGCCGCCGCCUC___AACGCAGAGGUUAUGUUUGUGU__ .((((((((((.((((..(((((.((........))))))).....((((((((((.............)))))))))).......))))..)))))).))))..... (-23.12 = -25.82 + 2.70)

| Location | 18,113,935 – 18,114,027 |
|---|---|
| Length | 92 |
| Sequences | 6 |
| Columns | 97 |
| Reading direction | forward |
| Mean pairwise identity | 77.78 |
| Mean single sequence MFE | -33.03 |
| Consensus MFE | -21.01 |
| Energy contribution | -23.87 |
| Covariance contribution | 2.86 |
| Combinations/Pair | 1.19 |
| Mean z-score | -1.29 |
| Structure conservation index | 0.64 |
| SVM decision value | 0.05 |
| SVM RNA-class probability | 0.556251 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
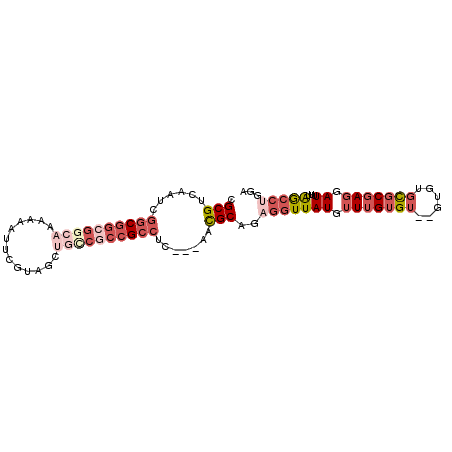
>2R_DroMel_CAF1 18113935 92 + 20766785 CGCGUCAAUCGGCGGCGGCAAAAAAUUCGUAGCUGCCGCCGCCUC---AACGCAGAGGUUAUGUUUGUGU--GUGUGCGCUAGGAUUUUAGCCUGGA ((((.((...((((((((((.............))))))))))..---.(((((((.......)))))))--.))))))(((((.......))))). ( -36.62) >DroSec_CAF1 65261 86 + 1 CGCGUCAAUCGGCGGCGGCAAAAAAUUCGUAG------CCGCCUA---AACGCAGAGGUUAUGUUUGUGU--GUGUGCGCGAGGAUUUUAGCCUGGA .((((.....((((((.((.........)).)------)))))..---.))))..((((((.(((..(((--(....))))..)))..))))))... ( -30.10) >DroSim_CAF1 66863 92 + 1 CGCGUCAAUCGGCGGCGGCAAAAAAUUCGUAGCUGCCGCCGCCUC---AACGCAGAGGUUAUGUUUGUGU--GUGUGCGCGAGGAUUUUAGCCUGGA .((((.....((((((((((.............))))))))))..---.))))..((((((.(((..(((--(....))))..)))..))))))... ( -38.52) >DroEre_CAF1 64189 91 + 1 CGCGUCAAUCGACGGCGGCAAAAA-UUCGCUGAUGCCGCCGCCUC---AUCGCAGAGGUUAUGUUUGUGU--GUGUGCGCGAGGAUUUAAGCCUGGG .(((......(.((((((((....-........)))))))))...---..)))..(((((((.(((((((--....))))))).))...)))))... ( -30.10) >DroYak_CAF1 66037 96 + 1 CGCGUCAAACGGCGGCGGCAAAAA-AUCGCUGCUGUCGCCGCCUCAUCAUCGCAGAGGUUAUGUUUGUGUGCGUGUGCGCGAGGAUUUUAGCCAGGA .(((......((((((((((....-........)))))))))).......)))...(((((.(((..(((((....)))))..)))..))))).... ( -36.62) >DroPer_CAF1 67200 81 + 1 CGCGUCAAAUCGCCCAGUCGACGCGCUC-UGCCGACCGCCGCUUC---GGUGCUGAGGUUAUGUUUGGGU--G----UGCGAUGAUUUUGA------ .(((......)))...((((.(((((((-.((.((((..(((...---.)))....))))..))..))))--)----))))))........------ ( -26.20) >consensus CGCGUCAAUCGGCGGCGGCAAAAAAUUCGUAGCUGCCGCCGCCUC___AACGCAGAGGUUAUGUUUGUGU__GUGUGCGCGAGGAUUUUAGCCUGGA .(((......((((((((((.............)))))))))).......)))..(((((((.(((((((......))))))).))...)))))... (-21.01 = -23.87 + 2.86)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:52:35 2006