
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 18,072,091 – 18,072,211 |
| Length | 120 |
| Max. P | 0.882050 |

| Location | 18,072,091 – 18,072,211 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 89.22 |
| Mean single sequence MFE | -49.54 |
| Consensus MFE | -41.68 |
| Energy contribution | -41.10 |
| Covariance contribution | -0.58 |
| Combinations/Pair | 1.25 |
| Mean z-score | -2.29 |
| Structure conservation index | 0.84 |
| SVM decision value | 0.92 |
| SVM RNA-class probability | 0.882050 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 18072091 120 - 20766785 GUGAGGCGGCGAUUGAGCGUUGCGUGGACACGCGCAUCUCGGAGGCCAUGCAGUGCAAGAUCUGCAUGGACCGCGCCAUCAACACAGUGUUCAAUCCGUGCUGUCACGUCAUCGCCUGCG ((.((((((.(((((((((.((((((....)))))).)..((.((((((((((........)))))))).))...))...........))))))))((((....))))...)))))))). ( -51.40) >DroVir_CAF1 44088 120 - 1 GUGAGGCGGCGAUUGAGCGCUGCGUGGACAAUCGCAUCCACGAGGCCAUGCAGUGCAAGAUCUGCAUGGAUCGUGCGAUCAACACGGUGUUCAAUCCCUGCUGUCACGUGAUUGCCUGUG (((((((((.((((((((((((.((.((...((((....((((..((((((((........)))))))).)))))))))).)).)))))))))))).))))).))))............. ( -54.80) >DroGri_CAF1 37920 120 - 1 GUGAAGCGGCGAUUGAGCGCUGCGUGGAUACGCGCAUCCUCGAGGCGAUGCAGUGCAAGAUCUGCAUGGAUCGUGCGAUCAACACAGUGUUCAAUCCAUGCUGUCACGUCAUCGCCUGUG ((((((((..((((((((((((.((.(((.((((((((((((...)))(((((........))))).)))).)))))))).)).))))))))))))..)))).))))............. ( -50.80) >DroWil_CAF1 117669 120 - 1 GUGAAGCAGCGAUUGAGCGCUGCGUGGACACGCGCAUAUCCGAGGCAAUGCAAUGUAAAAUCUGUAUGGAUCGCGCCAUCAACACAGUGUUCAAUCCAUGCUGUCACGUUAUCGCUUGUG ((((((((..((((((((((((.((.((...((((..(((((..(((...............))).))))).))))..)).)).))))))))))))..)))).))))............. ( -41.56) >DroMoj_CAF1 52038 120 - 1 GUGAGGCGGCGAUUGAGCGCUGCGUGGACAGCCGCAUCAACGAGGCUAUGCAGUGCAAGAUCUGCAUGGAUCGGCCGAUCAACACAGUGUUCAAUCCGUGCUGUCACGUCAUUGCCUGUG ...((((((.((((((((((((.((((....))))(((..(((..((((((((........)))))))).)))...))).....))))))))))))((((....))))...))))))... ( -47.50) >DroAna_CAF1 29750 120 - 1 GUGAGGCGGCGAUUGAGCGCUGCGUGGACACGCGGAUAUCCGAGGCUAUGCAGUGCAAGAUCUGCAUGGACCGCGCCAUCAACACAGUGUUCAAUCCGUGCUGUCACGUAAUUGCCUGUG ...((((((.((((((((((((.((.((..(((((....))..((((((((((........)))))))).)))))...)).)).))))))))))))((((....))))...))))))... ( -51.20) >consensus GUGAGGCGGCGAUUGAGCGCUGCGUGGACACGCGCAUCUCCGAGGCCAUGCAGUGCAAGAUCUGCAUGGAUCGCGCCAUCAACACAGUGUUCAAUCCGUGCUGUCACGUCAUCGCCUGUG ((((((((..((((((((((((.((.((..((((...........((((((((........))))))))..))))...)).)).))))))))))))..)))).))))............. (-41.68 = -41.10 + -0.58)

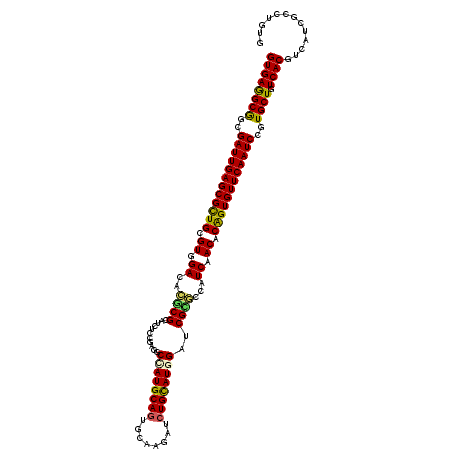
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:52:23 2006