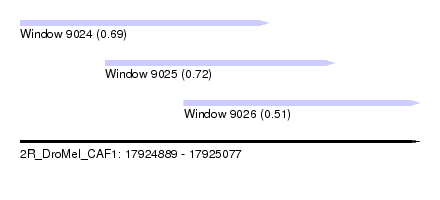
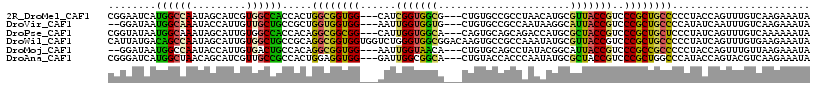
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 17,924,889 – 17,925,077 |
| Length | 188 |
| Max. P | 0.718454 |

| Location | 17,924,889 – 17,925,006 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 76.53 |
| Mean single sequence MFE | -51.20 |
| Consensus MFE | -23.38 |
| Energy contribution | -23.34 |
| Covariance contribution | -0.05 |
| Combinations/Pair | 1.45 |
| Mean z-score | -2.05 |
| Structure conservation index | 0.46 |
| SVM decision value | 0.33 |
| SVM RNA-class probability | 0.691064 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
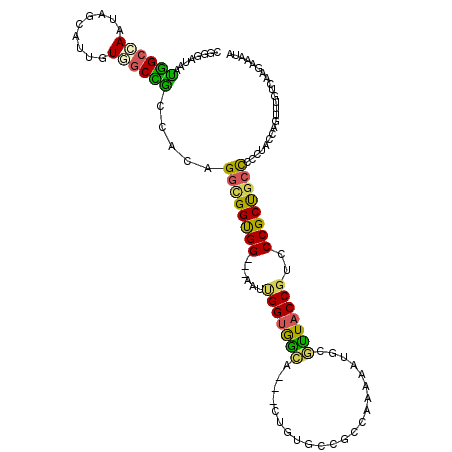
>2R_DroMel_CAF1 17924889 117 + 20766785 GCCUCACCGCCCGGAAUGGUCGUCAGUGCGGCCAGCUUCAAUGGCCGAAAUG---CCGGACUGGGUAAUGCCGGCGGUGGCGGAAUCAUGGCCAAUAGCAUCGUGGCCACCACUGGCGGU .....(((((((((..(((.(((.....((((((.......))))))..)))---)))..))))))...(((((.((((((.(.......((.....))....).)))))).)))))))) ( -52.20) >DroVir_CAF1 1229 111 + 1 GACUCAC---CUUGCAUGGUGGUCAGCUCGGCCAGUUUUAAUGGGCGCAACG---CUGGCCUGGGCAAUGCUGGCGGCG---GGAUAAUGGCAAAUACCAUUGUUGCUGCCGCUGGUGGU (((.(((---(......))))))).(((((((((((...............)---)))))).))))...((..((((((---(((((((((......)))))))).)))))))..))... ( -54.96) >DroPse_CAF1 1313 117 + 1 GCCUCGCCGCCCGGAAUGGUGGUCAGUGCGGCCAGUUUCAAUGGUCGGAAUG---CAGGCCUGGGCAACGCCGGCGGUGGCGGUAUAAUGGCAAAUAGCAUUGUGGCCACCACAGGCGGC .....((((((.(...(((((((((...((((((.......)))))).((((---(..(((.......(((((....))))).......))).....))))).)))))))))).)))))) ( -51.84) >DroWil_CAF1 2265 120 + 1 GCUUCGCCACCUGGAAUGGUUGUCAGCGCGGCUAGUUUCAAUGGGCGUAAUGCUGCCGGAAUGGGUAAUGCCGGCGGUGGCAUUAUGACAGCCAAUAGCAUUGUGGCUGCCGCAGGCGGU ...((((((((......))).......(((((.(((..(((((((((((((((..(((...(((......))).)))..)))))))....))).....)))))..)))))))).))))). ( -51.00) >DroMoj_CAF1 1286 111 + 1 GAAUCAC---CUUGCAUGGUGGUUAGCUCAGCCAAUUUUAAUGGACGCAACG---CUGGCAUGGGCAAUGCCGGCGGUG---GGAUAAUGGCCAAUACCAUUGUGACUGCCACAGGCGGU .((((((---(......)))))))......(((..................(---(((((((.....))))))))((..---(.(((((((......)))))))..)..))...)))... ( -42.70) >DroAna_CAF1 1179 117 + 1 GCCUCACCACCGGGCAUGGUUGUCAGCGCAGCCAGUUUCAAUGGCCGGAAUG---CCGGGCUCGGCAACGCCGGCGGCGGCGGGAUCAUGGCUAACAGCAUCGUUGCCGCCACUGGAGGU (((((....((((((.(((((((....)))))))..........((((....---)))))))))).......(((((((((((.....((.....))...)))))))))))....))))) ( -54.50) >consensus GCCUCACC_CCUGGAAUGGUGGUCAGCGCGGCCAGUUUCAAUGGCCGCAAUG___CCGGCCUGGGCAAUGCCGGCGGUGGCGGGAUAAUGGCCAAUAGCAUUGUGGCCGCCACAGGCGGU ((((.((.(((......))).)).)).)).(((........(((...........))).....)))..((((.(.((((((..(((((((........))))))))))))).).)))).. (-23.38 = -23.34 + -0.05)
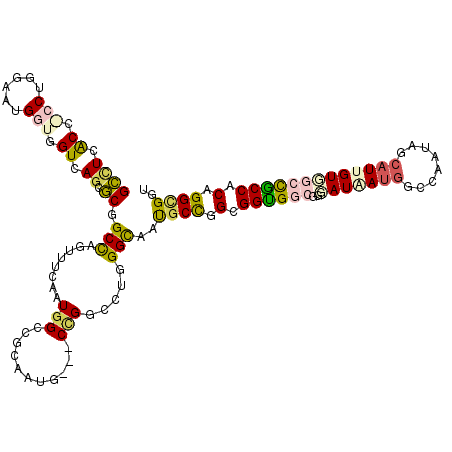

| Location | 17,924,929 – 17,925,037 |
|---|---|
| Length | 108 |
| Sequences | 6 |
| Columns | 108 |
| Reading direction | forward |
| Mean pairwise identity | 78.11 |
| Mean single sequence MFE | -46.85 |
| Consensus MFE | -23.50 |
| Energy contribution | -23.78 |
| Covariance contribution | 0.29 |
| Combinations/Pair | 1.48 |
| Mean z-score | -2.07 |
| Structure conservation index | 0.50 |
| SVM decision value | 0.39 |
| SVM RNA-class probability | 0.718454 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 17924929 108 + 20766785 AUGGCCGAAAUGCCGGACUGGGUAAUGCCGGCGGUGGCGGAAUCAUGGCCAAUAGCAUCGUGGCCACCACUGGCGGUGGCAUCGGUGGCGCUGUGCCGCCUAACAUGC .((((((....(((((.(........))))))(((......))).))))))...((((.((.((((((......))))))...((((((.....))))))..)))))) ( -47.30) >DroVir_CAF1 1266 105 + 1 AUGGGCGCAACGCUGGCCUGGGCAAUGCUGGCGGCG---GGAUAAUGGCAAAUACCAUUGUUGCUGCCGCUGGUGGUGGAAUUGGUGGUGCUGUGCCGCCAAUAAGGC ...((((((.(((((.((....(..(((..((((((---(((((((((......)))))))).)))))))..)))...)....))))))).))))))(((.....))) ( -49.50) >DroPse_CAF1 1353 108 + 1 AUGGUCGGAAUGCAGGCCUGGGCAACGCCGGCGGUGGCGGUAUAAUGGCAAAUAGCAUUGUGGCCACCACAGGCGGCGGCAUUGGUGGCACAGUGCAGCAGACCAUGC ((((((.........((((((((...)))...((((((..(((((((........))))))))))))).))))).((.((((((......)))))).)).)))))).. ( -48.70) >DroMoj_CAF1 1323 105 + 1 AUGGACGCAACGCUGGCAUGGGCAAUGCCGGCGGUG---GGAUAAUGGCCAAUACCAUUGUGACUGCCACAGGCGGUGGAAUUGGUAACACUGUGCAGCCUAUACGGC ......(((.(((((((((.....)))))))))(((---((.......))..(((((...(.((((((...)))))).)...))))).)))..))).(((.....))) ( -41.00) >DroAna_CAF1 1219 108 + 1 AUGGCCGGAAUGCCGGGCUCGGCAACGCCGGCGGCGGCGGGAUCAUGGCUAACAGCAUCGUUGCCGCCACUGGAGGUGGGAUUGGCGGCACUGUACCACCCAAUAUGC ...(((((..(((((....)))))...)))))(((((((((((..((.....))..))).))))))))....(.(((((.....(((....))).))))))....... ( -45.90) >DroPer_CAF1 1212 108 + 1 AUGGUCGGAAUGCAGGCCUGGGCAACGCCGGCGGUGGCGGUAUAAUGGCAAAUAGCAUUGUGGCCACCACAGGCGGCGGCAUUGGUGGCACAGUGCAGCAGACCAUGC ((((((.........((((((((...)))...((((((..(((((((........))))))))))))).))))).((.((((((......)))))).)).)))))).. ( -48.70) >consensus AUGGCCGGAAUGCAGGCCUGGGCAACGCCGGCGGUGGCGGGAUAAUGGCAAAUAGCAUUGUGGCCACCACAGGCGGUGGCAUUGGUGGCACUGUGCAGCCGAACAUGC ...((((....(((......)))..((((.(.((((((..(((((((........))))))))))))).).))))..........))))................... (-23.50 = -23.78 + 0.29)


| Location | 17,924,966 – 17,925,077 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 76.55 |
| Mean single sequence MFE | -38.65 |
| Consensus MFE | -21.87 |
| Energy contribution | -20.93 |
| Covariance contribution | -0.94 |
| Combinations/Pair | 1.67 |
| Mean z-score | -1.50 |
| Structure conservation index | 0.57 |
| SVM decision value | -0.05 |
| SVM RNA-class probability | 0.510093 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 17924966 111 + 20766785 CGGAAUCAUGGCCAAUAGCAUCGUGGCCACCACUGGCGGUGG---CAUCGGUGGCG---CUGUGCCGCCUAACAUGCGUUACCGUCCCGCUGCCCCCUACCAGUUUGUCAAGAAAUA .((.....((((((.........)))))).....((((((((---...((((((((---((((........))).)))))))))..)))))))).....))................ ( -43.40) >DroVir_CAF1 1302 109 + 1 --GGAUAAUGGCAAAUACCAUUGUUGCUGCCGCUGGUGGUGG---AAUUGGUGGUG---CUGUGCCGCCAAUAAGGCAUUACCGUCCCGCUGCCCCAUAUCAAUUUGUCAAGAAAUA --.(((((.((((...((....))...))))...((..((((---.(((((((((.---....)))))))))..(((......)))))))..))..........)))))........ ( -34.60) >DroPse_CAF1 1390 111 + 1 CGGUAUAAUGGCAAAUAGCAUUGUGGCCACCACAGGCGGCGG---CAUUGGUGGCA---CAGUGCAGCAGACCAUGCGCUACCGUCCCGCUGCUCCCUAUCAGUUUGUCAAAAAAUA ........((((((((.((((((((.((((((...((....)---)..))))))))---))))))(((((.....(((....)))....)))))........))))))))....... ( -40.90) >DroWil_CAF1 2345 117 + 1 CAUUAUGACAGCCAAUAGCAUUGUGGCUGCCGCAGGCGGUGGUGGUCUGGGUGGCGGACAAGUGCCGCCAAAUAUGCGUUACCGUCCCGCUGCCCCCUAUCAGUUUGUGAAGAAAUA ........((((((.........)))))).(((((((..((((((...(((..((((((..(((.(((.......))).))).)).))))..))).)))))))))))))........ ( -42.30) >DroMoj_CAF1 1359 109 + 1 --GGAUAAUGGCCAAUACCAUUGUGACUGCCACAGGCGGUGG---AAUUGGUAACA---CUGUGCAGCCUAUACGGCAUUACCGUCCCGCCGCCCCCUACCAGUUUGUUAAGAAAUA --..(((((((......)))))))(((((.....((((((((---...((((((..---(((((.......)))))..))))))..))))))))......)))))............ ( -36.20) >DroAna_CAF1 1256 111 + 1 CGGGAUCAUGGCUAACAGCAUCGUUGCCGCCACUGGAGGUGG---GAUUGGCGGCA---CUGUACCACCCAAUAUGCGCUACCGUCCCGCUGGCCCAUACCAGUACGUCAAGAAAUA .....((.((((.....(((....)))....(((((..((((---(.(..((((.(---(.((((((.......)).).))).)).))))..)))))).)))))..)))).)).... ( -34.50) >consensus CGGGAUAAUGGCCAAUAGCAUUGUGGCCGCCACAGGCGGUGG___AAUUGGUGGCA___CUGUGCCGCCAAAAAUGCGUUACCGUCCCGCUGCCCCCUACCAGUUUGUCAAGAAAUA ........((((((.........)))))).....((((((((......(((((((......................)))))))..))))))))....................... (-21.87 = -20.93 + -0.94)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:50:30 2006