
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 17,898,981 – 17,899,139 |
| Length | 158 |
| Max. P | 0.855319 |

| Location | 17,898,981 – 17,899,099 |
|---|---|
| Length | 118 |
| Sequences | 6 |
| Columns | 118 |
| Reading direction | reverse |
| Mean pairwise identity | 78.47 |
| Mean single sequence MFE | -43.09 |
| Consensus MFE | -26.73 |
| Energy contribution | -27.45 |
| Covariance contribution | 0.72 |
| Combinations/Pair | 1.24 |
| Mean z-score | -1.37 |
| Structure conservation index | 0.62 |
| SVM decision value | 0.00 |
| SVM RNA-class probability | 0.534315 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
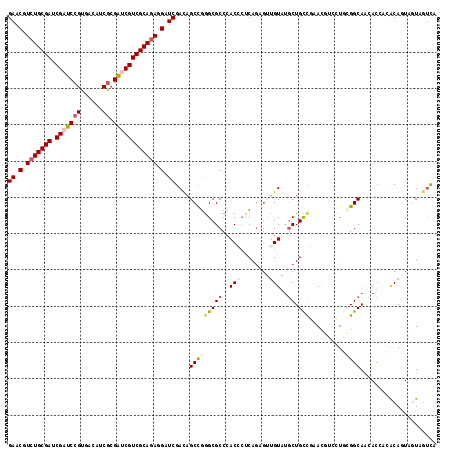
>2R_DroMel_CAF1 17898981 118 - 20766785 GAACGUCUGCGAUCGAUCCGUGAUGUCGCGAUCGUCGCAGAGGAUCGACAGCCGGGCGCCCACCCGCAGUGCGGUGUGCUGUCGAACCCUCUGGGGCAGGAUUUCGCAGUAGUAGUCA ..((..((((((..((((((((....)))....(((.((((((.((((((((...((((((((.....))).)))))))))))))..)))))).))).)))))))))))..))..... ( -57.10) >DroVir_CAF1 21841 118 - 1 GAACGUCUGCGAGCGAUCCGUUACAUCGCGAUCGUCGCAGAGGAUCGACAGCCGGGCGCCCACGCUCAGUUUUGUAUGCUGCCUCACGUCCUGCGGCAACACCACACAGUAAUAGUCA ((.(.(((((((.(((((((......)).)))))))))))).).))(((....(((((....))))).((..(((.((((((..........)))))))))..)).........))). ( -44.00) >DroGri_CAF1 22237 118 - 1 GAACGUCUGCGAACGAUCCGUAACAUCGCGAUCGUCGCAGAGGAUCGAUAGCCGGGCGCCAACACUCAGCGUUGUAUACUGCUCCACUUUGUCCGGCAAUAUCUGACAUUUGUAGACA ((.(.(((((((.(((((((......)).)))))))))))).).))((((((((((((.........((((........))))......))))))))..))))............... ( -40.06) >DroWil_CAF1 26630 118 - 1 GAACGUCUGCGAACGAUCCGUUAUGUCGAGACCGUCGCAAAGGAUCGACAGCCGAGCGCCCACCAUCAACGAUGUAUGCUGUCGAAUUUGUUCUGGCAAUACCAAACAAUAAUACUCA (((((..(((((.((.((((......)).)).))))))).....((((((((.(......)..((((...))))...))))))))...)))))(((.....))).............. ( -28.60) >DroMoj_CAF1 22456 118 - 1 GAACGUCUGCGAUCGAUCCGUGACAUCGCGACCGUCGCAGAGGAUCGACAGCUUGGCGCCCACACUCAGAGCUGUGUGCUGCCUCACGCCCUGCGGCAACACCUCGCAGUAGUAGUCG ((.(.(((((((.((...((((....))))..))))))))).).))(((.(((.((((..(((((........))))).)))).......((((((.......)))))).))).))). ( -43.10) >DroAna_CAF1 45253 118 - 1 GAACGUCUGCGAUCGAUCAGUGAUGUCACGGUCGUCGCAGAGGAUCGACAGCCGGGCGCCCACCCUCAGAGUUGUGCGCUGUCGAACGUCCUGCGGCAGGAUAACACAGUAGUAGUUC ((((.(((((((.(((((.(((....)))))))))))))))...((((((((.(((......))).....(.....)))))))))..((((((...))))))............)))) ( -45.70) >consensus GAACGUCUGCGAUCGAUCCGUGACAUCGCGAUCGUCGCAGAGGAUCGACAGCCGGGCGCCCACCCUCAGAGUUGUAUGCUGCCGAACGUCCUGCGGCAACACCACACAGUAGUAGUCA ((.(.(((((((.(((((((......)).)))))))))))).).))....(((((((((..(((........)))..)).)))..........))))..................... (-26.73 = -27.45 + 0.72)

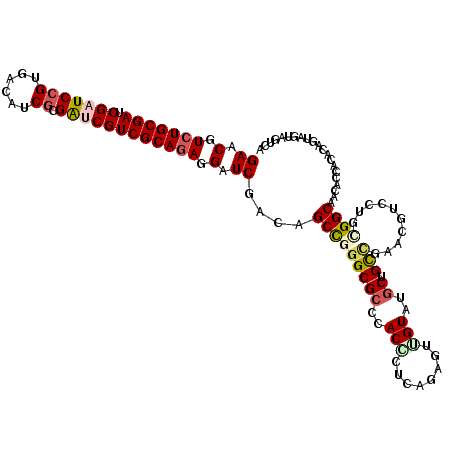
| Location | 17,899,021 – 17,899,139 |
|---|---|
| Length | 118 |
| Sequences | 6 |
| Columns | 118 |
| Reading direction | forward |
| Mean pairwise identity | 83.05 |
| Mean single sequence MFE | -48.07 |
| Consensus MFE | -38.34 |
| Energy contribution | -37.07 |
| Covariance contribution | -1.27 |
| Combinations/Pair | 1.32 |
| Mean z-score | -2.25 |
| Structure conservation index | 0.80 |
| SVM decision value | 0.81 |
| SVM RNA-class probability | 0.855319 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 17899021 118 + 20766785 CACACCGCACUGCGGGUGGGCGCCCGGCUGUCGAUCCUCUGCGACGAUCGCGACAUCACGGAUCGAUCGCAGACGUUCAGGCAGGGCGACCACCACAAGCUCGAGUGUCGAACCUAUC .((((((..((...(((((.(((((.(((...((.(.((((((((((((.((......))))))).))))))).).)).))).))))).)))))...))..)).)))).......... ( -54.00) >DroVir_CAF1 21881 118 + 1 CAUACAAAACUGAGCGUGGGCGCCCGGCUGUCGAUCCUCUGCGACGAUCGCGAUGUAACGGAUCGCUCGCAGACGUUCAAGCAGGGCGACCGUCACAAGCUCGUGUGCCGCACGUACC ..(((......((((((((((((((.(((...((.(.((((((((((((.((......))))))).))))))).).)).))).))))).))..)))..))))(((.....)))))).. ( -49.90) >DroGri_CAF1 22277 118 + 1 UAUACAACGCUGAGUGUUGGCGCCCGGCUAUCGAUCCUCUGCGACGAUCGCGAUGUUACGGAUCGUUCGCAGACGUUCAAGCAGGGCGACAAUCACAAGCUCGAGUGCCGCACCUAUC .......((((((((((((.(((((.(((...((.(.((((((((((((.((......))))))).))))))).).)).))).))))).)))).....)))).))))........... ( -44.70) >DroWil_CAF1 26670 118 + 1 CAUACAUCGUUGAUGGUGGGCGCUCGGCUGUCGAUCCUUUGCGACGGUCUCGACAUAACGGAUCGUUCGCAGACGUUCAAGCAGGGUGACCACCAUAAGUUAGAAUGUCGAACAUAUA ...(((((....(((((((.(((((.(((...((.(.((((((((((((.((......))))))).))))))).).)).))).))))).)))))))......).)))).......... ( -42.00) >DroMoj_CAF1 22496 118 + 1 CACACAGCUCUGAGUGUGGGCGCCAAGCUGUCGAUCCUCUGCGACGGUCGCGAUGUCACGGAUCGAUCGCAGACGUUCAAGCAAGGCGACCACCACAGGCUCGUCUGCCAAACGUUCC ((.(((((.(((.(.((((.((((..(((...((.(.((((((((((((.((......))))))).))))))).).)).)))..)))).))))).)))))).)).))........... ( -46.60) >DroAna_CAF1 45293 118 + 1 CGCACAACUCUGAGGGUGGGCGCCCGGCUGUCGAUCCUCUGCGACGACCGUGACAUCACUGAUCGAUCGCAGACGUUCAGGCAGGGCGACCACCACAAGCUCGAGUGUCGAACCUAUC ((.(((.(((.((((((((.(((((.(((...((.(.(((((((((((.(((....))).).))).))))))).).)).))).))))).))))).....)))))))))))........ ( -51.20) >consensus CACACAACACUGAGGGUGGGCGCCCGGCUGUCGAUCCUCUGCGACGAUCGCGACAUCACGGAUCGAUCGCAGACGUUCAAGCAGGGCGACCACCACAAGCUCGAGUGCCGAACCUAUC .........((....((((.(((((.(((...((.(.((((((((((((.((......))))))).))))))).).)).))).))))).))))....))................... (-38.34 = -37.07 + -1.27)


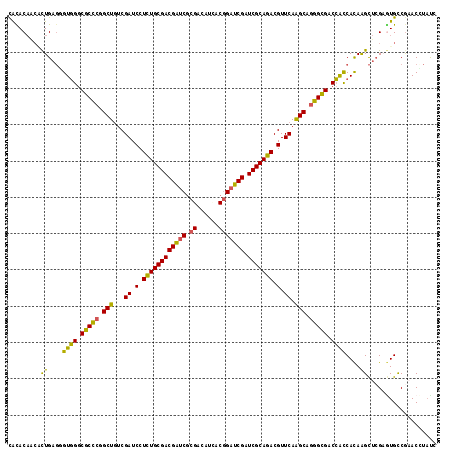
| Location | 17,899,021 – 17,899,139 |
|---|---|
| Length | 118 |
| Sequences | 6 |
| Columns | 118 |
| Reading direction | reverse |
| Mean pairwise identity | 83.05 |
| Mean single sequence MFE | -50.17 |
| Consensus MFE | -38.99 |
| Energy contribution | -40.72 |
| Covariance contribution | 1.73 |
| Combinations/Pair | 1.16 |
| Mean z-score | -1.95 |
| Structure conservation index | 0.78 |
| SVM decision value | 0.50 |
| SVM RNA-class probability | 0.758555 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 17899021 118 - 20766785 GAUAGGUUCGACACUCGAGCUUGUGGUGGUCGCCCUGCCUGAACGUCUGCGAUCGAUCCGUGAUGUCGCGAUCGUCGCAGAGGAUCGACAGCCGGGCGCCCACCCGCAGUGCGGUGUG .(((((((((.....)))))))))(((((.(((((.((.(((.(.(((((((.(((((.(((....))))))))))))))).).)))...)).))))).)))))(((...)))..... ( -59.30) >DroVir_CAF1 21881 118 - 1 GGUACGUGCGGCACACGAGCUUGUGACGGUCGCCCUGCUUGAACGUCUGCGAGCGAUCCGUUACAUCGCGAUCGUCGCAGAGGAUCGACAGCCGGGCGCCCACGCUCAGUUUUGUAUG ....(((((((.((..((((..(((..((.(((((.((((((.(.(((((((.(((((((......)).)))))))))))).).)))..))).)))))))))))))).)).))))))) ( -53.40) >DroGri_CAF1 22277 118 - 1 GAUAGGUGCGGCACUCGAGCUUGUGAUUGUCGCCCUGCUUGAACGUCUGCGAACGAUCCGUAACAUCGCGAUCGUCGCAGAGGAUCGAUAGCCGGGCGCCAACACUCAGCGUUGUAUA .....(((((((.((.((....(((.(((.(((((.((((((.(.(((((((.(((((((......)).)))))))))))).).)))..))).))))).)))))))))).))))))). ( -51.20) >DroWil_CAF1 26670 118 - 1 UAUAUGUUCGACAUUCUAACUUAUGGUGGUCACCCUGCUUGAACGUCUGCGAACGAUCCGUUAUGUCGAGACCGUCGCAAAGGAUCGACAGCCGAGCGCCCACCAUCAACGAUGUAUG ..........(((((.......(((((((.(.(.(.((((((.(.(.(((((.((.((((......)).)).))))))).).).)))..))).).).).)))))))....)))))... ( -29.30) >DroMoj_CAF1 22496 118 - 1 GGAACGUUUGGCAGACGAGCCUGUGGUGGUCGCCUUGCUUGAACGUCUGCGAUCGAUCCGUGACAUCGCGACCGUCGCAGAGGAUCGACAGCUUGGCGCCCACACUCAGAGCUGUGUG ...........((.((.((((((.(((((.((((..((((((.(.(((((((.((...((((....))))..))))))))).).)))..)))..)))).)))).).))).))))).)) ( -50.40) >DroAna_CAF1 45293 118 - 1 GAUAGGUUCGACACUCGAGCUUGUGGUGGUCGCCCUGCCUGAACGUCUGCGAUCGAUCAGUGAUGUCACGGUCGUCGCAGAGGAUCGACAGCCGGGCGCCCACCCUCAGAGUUGUGCG .(((((((((.....)))))))))(((((.(((((.((.(((.(.(((((((.(((((.(((....))))))))))))))).).)))...)).))))).))))).............. ( -57.40) >consensus GAUAGGUUCGACACUCGAGCUUGUGGUGGUCGCCCUGCUUGAACGUCUGCGAUCGAUCCGUGACAUCGCGAUCGUCGCAGAGGAUCGACAGCCGGGCGCCCACCCUCAGAGUUGUAUG .....(((((.....)))))(((..((((.(((((.((((((.(.(((((((.(((((((......)).)))))))))))).).)))..))).))))).))))...)))......... (-38.99 = -40.72 + 1.73)

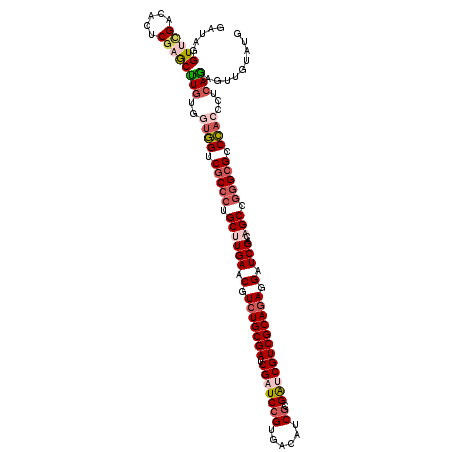

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:50:21 2006