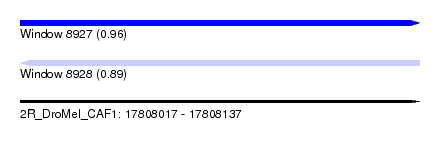
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 17,808,017 – 17,808,137 |
| Length | 120 |
| Max. P | 0.963647 |

| Location | 17,808,017 – 17,808,137 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 77.72 |
| Mean single sequence MFE | -51.30 |
| Consensus MFE | -25.71 |
| Energy contribution | -26.68 |
| Covariance contribution | 0.98 |
| Combinations/Pair | 1.33 |
| Mean z-score | -2.66 |
| Structure conservation index | 0.50 |
| SVM decision value | 1.56 |
| SVM RNA-class probability | 0.963647 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
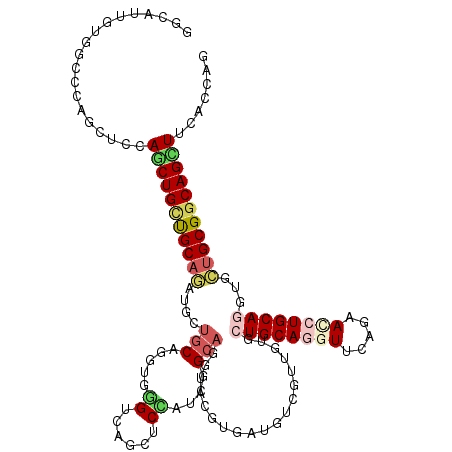
>2R_DroMel_CAF1 17808017 120 + 20766785 GGCGUUGUGGCUCAGCUCCAACUGGUGCAACUGGUGCAUGUGGGUCAGCUCCAGCGGCAGGUGGGUGAUGUUGUUGUGCUGCAGGUUCAGCACCUGCAGAUGCUGCGGCAGUUUCCUCAG (.(((((..(((..((.(((....(((((.....))))).))))).)))..))))).)..(.(((...((((((.(((((((((((.....)))))))).))).))))))...))).).. ( -48.10) >DroVir_CAF1 109568 120 + 1 GGCAUUGUGGCCCAGCUCCAGCUGCUGCAAGUGCUGCAGAUGGGUCAACUCCAUGGGCAGGGACGUGAUAUCGUUGUGCUGCAAAUUGAGAACCUGCAAAUGUUGCGGCAGCUUCACCAG (((......))).......(((((((((((....((((((((((.....)))))..((((.((((......))))...))))...........)))))....)))))))))))....... ( -40.40) >DroGri_CAF1 101094 120 + 1 GUUAUUGUGUCCCAGUUCCAGCUGCUGCAGAUGAUGCAGAUGUGUUAGCUCAAUCGGCAGAUACGUGAUGUCAUUGUGCUGCAGAUUGAGGACCUGCAAGUGCUGCGGCAGUUUAAGCAG ..............(((..(((((((((((....(((((....(((..(((((((.((((.((((((....)).)))))))).)))))))))))))))....)))))))))))..))).. ( -46.60) >DroMoj_CAF1 118080 120 + 1 AUGGUUGUGACCCAGUUCCAGCUGCUGCAGAUGCUGCAAGUGUGUCAGCUCGAUUGGCAGCGUUGUGAUGUCAUUGUGCUGCAAGUUGAGCACCUGCAGCUGCUGCGGCAGCUUCAACAA ..(((....)))..(((..(((((((((((..((((((.((((.((((((......((((((..(((....)))..)))))).)))))))))).))))))..)))))))))))..))).. ( -58.30) >DroAna_CAF1 82865 120 + 1 GGCAUUGUGGCUCAGCUCCACCUGUCGCAGCUGACUCAUGUGGGUCAGCUCCAGUGGCAGGUGGGUGAUGUUGUUGUGCUGCAGGUUCAGGACCUGCAGGUGCUGCGGCAGCUUCCUUAG (((((.(..((...((.((((((((((((((((((((....)))))))))...)))))))))))))...))..).)))))(((((((...)))))))(((.((((...))))..)))... ( -62.90) >DroPer_CAF1 84070 120 + 1 GUCGUUGUGACCGAUCUCCAGCUGCUGCAGGUGCUGCAGGUGGGUGAUUUCGAUGGGCAGGUUCGUGAUGCUGUUGUGCUGCAGGUUCAGAAUCUGCAGGUUCAGCGGCAGCUUCACCAG (((((((.((((((((((((.((((.((....)).)))).)))).)))).......((((((((.(((..((((......))))..))))))))))).)))))))))))........... ( -51.50) >consensus GGCAUUGUGGCCCAGCUCCAGCUGCUGCAGAUGCUGCAGGUGGGUCAGCUCCAUCGGCAGGUACGUGAUGUCGUUGUGCUGCAGGUUCAGAACCUGCAGGUGCUGCGGCAGCUUCACCAG ...................(((((((((((....(((.....((......))....)))...................((((((((.....))))))))...)))))))))))....... (-25.71 = -26.68 + 0.98)

| Location | 17,808,017 – 17,808,137 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 77.72 |
| Mean single sequence MFE | -40.27 |
| Consensus MFE | -20.84 |
| Energy contribution | -20.90 |
| Covariance contribution | 0.06 |
| Combinations/Pair | 1.35 |
| Mean z-score | -2.26 |
| Structure conservation index | 0.52 |
| SVM decision value | 0.98 |
| SVM RNA-class probability | 0.894156 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 17808017 120 - 20766785 CUGAGGAAACUGCCGCAGCAUCUGCAGGUGCUGAACCUGCAGCACAACAACAUCACCCACCUGCCGCUGGAGCUGACCCACAUGCACCAGUUGCACCAGUUGGAGCUGAGCCACAACGCC ...((....))((..((((..((((((((.....)))))))).............((.((.(((.(((((.((((.....)).)).))))).)))...)).)).)))).))......... ( -39.50) >DroVir_CAF1 109568 120 - 1 CUGGUGAAGCUGCCGCAACAUUUGCAGGUUCUCAAUUUGCAGCACAACGAUAUCACGUCCCUGCCCAUGGAGUUGACCCAUCUGCAGCACUUGCAGCAGCUGGAGCUGGGCCACAAUGCC (..((..((((((.((((...((((((((..(((((((((((....(((......)))..)))).....)))))))...))))))))...)))).))))))...))..)........... ( -40.10) >DroGri_CAF1 101094 120 - 1 CUGCUUAAACUGCCGCAGCACUUGCAGGUCCUCAAUCUGCAGCACAAUGACAUCACGUAUCUGCCGAUUGAGCUAACACAUCUGCAUCAUCUGCAGCAGCUGGAACUGGGACACAAUAAC .........((((.((((....(((((((.(((((((.((((.....((....)).....)))).))))))).......)))))))....)))).))))..................... ( -33.60) >DroMoj_CAF1 118080 120 - 1 UUGUUGAAGCUGCCGCAGCAGCUGCAGGUGCUCAACUUGCAGCACAAUGACAUCACAACGCUGCCAAUCGAGCUGACACACUUGCAGCAUCUGCAGCAGCUGGAACUGGGUCACAACCAU ..(((..((((((.((((..((((((((((.(((.((((((((....((....))....))))).....))).)))..))))))))))..)))).))))))..)))(((.......))). ( -49.80) >DroAna_CAF1 82865 120 - 1 CUAAGGAAGCUGCCGCAGCACCUGCAGGUCCUGAACCUGCAGCACAACAACAUCACCCACCUGCCACUGGAGCUGACCCACAUGAGUCAGCUGCGACAGGUGGAGCUGAGCCACAAUGCC ....((.((((((....))..((((((((.....))))))))..............(((((((....((.(((((((.(....).))))))).)).)))))))))))...))........ ( -45.40) >DroPer_CAF1 84070 120 - 1 CUGGUGAAGCUGCCGCUGAACCUGCAGAUUCUGAACCUGCAGCACAACAGCAUCACGAACCUGCCCAUCGAAAUCACCCACCUGCAGCACCUGCAGCAGCUGGAGAUCGGUCACAACGAC ...((((.((((..(.((...((((((.........)))))))))..)))).))))......(((.((((....)..(((.((((.((....)).)))).))).))).)))......... ( -33.20) >consensus CUGAUGAAGCUGCCGCAGCACCUGCAGGUCCUCAACCUGCAGCACAACAACAUCACGAACCUGCCAAUGGAGCUGACCCACCUGCAGCACCUGCAGCAGCUGGAGCUGGGCCACAACGAC .......((((((.((((...((((((((.....))))))))...................(((....((......)).....)))....)))).))))))................... (-20.84 = -20.90 + 0.06)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:48:56 2006