
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 2,858,435 – 2,858,546 |
| Length | 111 |
| Max. P | 0.758385 |

| Location | 2,858,435 – 2,858,546 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 112 |
| Reading direction | reverse |
| Mean pairwise identity | 80.23 |
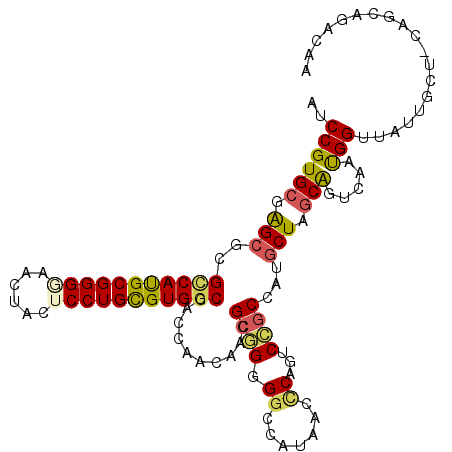
| Mean single sequence MFE | -40.32 |
| Consensus MFE | -28.07 |
| Energy contribution | -28.02 |
| Covariance contribution | -0.05 |
| Combinations/Pair | 1.35 |
| Mean z-score | -1.59 |
| Structure conservation index | 0.70 |
| SVM decision value | 0.49 |
| SVM RNA-class probability | 0.758385 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
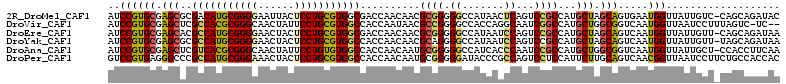
>2R_DroMel_CAF1 2858435 111 - 20766785 AUCCGUGCGAGCGCGACAUGCGGGGAAUUACUCCUGCGUGGCGACCAACAACGCGGGGGCCAUAACUCAGUCCGCCAUGCUAGCAGUGAAUGGUUAUUGUC-CAGCAGAUAC (((.....(.(((.(((..((((((......))))))(((((..((........))..)))))......))))))).((((.(((((((....))))))).-.))))))).. ( -36.70) >DroVir_CAF1 10204 109 - 1 AUCCGUGCGAGCUCGCCACGCGGGCAACUAUUCCUGCGUGGCCACCAAUAACGCCGGGGCCACCAGGCAAUCGGCCAUGCUGGCGGUCAAUGGUUAAUCCUUUAGUC-UC-- ..((((..((.(.(((((.((((((....(((((((.((((((.((.........)))))))))))).)))..))).))))))))))).))))..............-..-- ( -42.20) >DroEre_CAF1 9938 111 - 1 AUCCGUGCGAGCACGCCAUGCGGGGAACUACUCCUGCGUGGCGACCAACAACGCGGGGGCCAUAAUCCAGUCCGCCAUGCUAGCAGUCAAUGGUUAUUGUU-CAGCAGAUAA .....((((((((((((((((((((......))))))))))))((((...((..((((((.........)))).)).((....))))...))))...))))-).)))..... ( -40.10) >DroYak_CAF1 11928 111 - 1 AUCCGUGCGAGCGCGCCAUGCGGGGAACUACUCCUGCGUGGCCACCAACAACGCAGGGGCCAUAAUCCAGUCCGCCAUGCUAGCAGUCAAUGGUUAUUGUU-UAGCAGAUAA (((.(.(((..((.(((((((((((......))))))))))))............(((.......))).)..)))).((((((((((........)))).)-)))))))).. ( -35.60) >DroAna_CAF1 9918 111 - 1 AUCCGUGCGAGCUCGUCACGCGGGCAACUAUUCCUGUGUGGCCACCAACAAUGCGGGGGCCAUCACCCAAUCCGCCAUGCUGGCGGUCAAUGGUUAUUGCU-CCACCUUCAA ....(((.((((..((((((((((........)))))))))).((((.......(((........)))...(((((.....)))))....))))....)))-))))...... ( -42.60) >DroPer_CAF1 10054 112 - 1 GUCCGUGAGGGCCCGCCAUGCGGGAAACUACUCCUGCGUGGCCACCAACAAUGCGGGGGAUACCCGCCAGUCCUCCAUUCUUGCAGUCAACGGUUAAUCCUUCUGCCACCAC ....(.((((((..(((((((((((......)))))))))))..........(((((.....)))))..)))))))......((((.....((.....))..))))...... ( -44.70) >consensus AUCCGUGCGAGCGCGCCAUGCGGGGAACUACUCCUGCGUGGCCACCAACAACGCGGGGGCCAUAACCCAGUCCGCCAUGCUAGCAGUCAAUGGUUAUUGCU_CAGCAGACAA ..((((((.(((..(((((((((((......)))))))))))..........((((.((.......))...))))...))).))).....)))................... (-28.07 = -28.02 + -0.05)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:41:18 2006