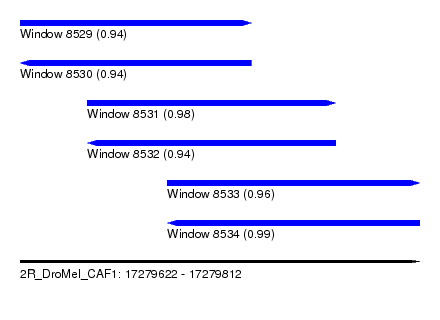
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 17,279,622 – 17,279,812 |
| Length | 190 |
| Max. P | 0.993118 |

| Location | 17,279,622 – 17,279,732 |
|---|---|
| Length | 110 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 77.35 |
| Mean single sequence MFE | -34.65 |
| Consensus MFE | -19.27 |
| Energy contribution | -20.63 |
| Covariance contribution | 1.36 |
| Combinations/Pair | 1.14 |
| Mean z-score | -2.24 |
| Structure conservation index | 0.56 |
| SVM decision value | 1.26 |
| SVM RNA-class probability | 0.937589 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
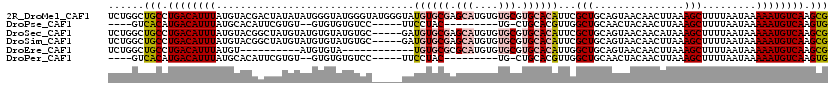
>2R_DroMel_CAF1 17279622 110 + 20766785 UCCG--UUUGU------GUGUGUGUGUCCUGCAGCCAAUAAGUUGCCAGCAGCCGUUGCCAUUGCAGC--GAAUAUCCUUUCUGGCUGCCUGACAUUUAUGUACGACUAUAUAUGGGUAU ((((--(((((------(..((.(((((..(((((((....((((....))))((((((....)))))--)...........)))))))..))))).))..)))))......)))))... ( -39.10) >DroPse_CAF1 4736 99 + 1 ---------AU------GCGUGUGCGUCCUGCAGCCAAUAAGUUGCUAGCAGCCGUUGCCACUGCAGCACAAAUAUCCUU----GUCACAUGACAUUUAUGCACAUUCGUGU--GUGUGU ---------((------(.((((((((.((((((.((((..((((....)))).))))...)))))).)).........(----(((....)))).....)))))).)))..--...... ( -28.90) >DroSec_CAF1 2660 118 + 1 UCCGCUUGUGUGUUUGUGUGUGUGUAUCCUGCAACCAAUAAGUUGCCAGCAGCCGUUGCCAUUGCAGC--GAAUACCCUUUCUGGCUGCCUGACAUUUAUGUACGGCUAUGUAUGUGUAU ..(((.((..((.(((((..(((((.....(((((......)))))(((((((((((((....)))))--(((......))).))))).)))))))..)..))))).))..)).)))... ( -35.70) >DroSim_CAF1 4629 110 + 1 --CGCUUGUGU------GUGUGUGUGUCCUGCAGCCAAUAAGUUGCCAGCAGCCGUUGCCAUUGCAGC--GAAUAUCCUUUCUGGCUGCCUGACAUUUAUGUACGGCUAUGUAUGUGUAU --.(((.(((.------.((...(((((..(((((((....((((....))))((((((....)))))--)...........)))))))..))))).))..))))))............. ( -37.70) >DroEre_CAF1 1014 95 + 1 UGC------GU------GUGUGUGUGUCCUGCAGCCAAUAAGUUGCCAGCAGCCGUUGCCAUUGCAGC--GAAUAUCCUUUCUGGCUGCCUGACAUUUAUGU----------AUGUGUA- .((------((------(..((.(((((..(((((((....((((....))))((((((....)))))--)...........)))))))..))))).))..)----------))))...- ( -37.60) >DroPer_CAF1 4782 99 + 1 ---------AU------GCGUGUGCGUCCUGCAGCCAAUAAGUUGCUAGCAGCCGUUGCCACUGCAGCACAAAUAUCCUU----GUCACAUGACAUUUAUGCACAUUCGUGU--GUGUGU ---------((------(.((((((((.((((((.((((..((((....)))).))))...)))))).)).........(----(((....)))).....)))))).)))..--...... ( -28.90) >consensus __C______GU______GUGUGUGUGUCCUGCAGCCAAUAAGUUGCCAGCAGCCGUUGCCAUUGCAGC__GAAUAUCCUUUCUGGCUGCCUGACAUUUAUGUACGGCUAUGUAUGUGUAU .................(((((.(((((..(((((......)))))..(((((((((((....)))))..(((......))).))))))..))))).))))).................. (-19.27 = -20.63 + 1.36)

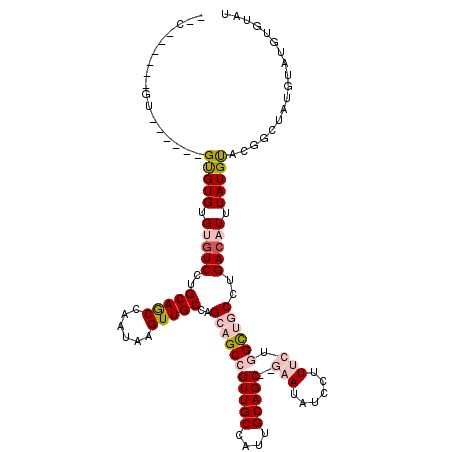
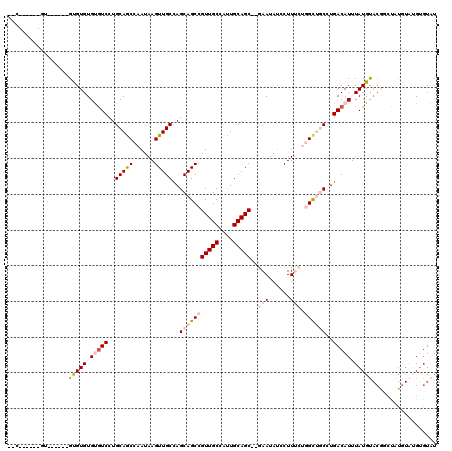
| Location | 17,279,622 – 17,279,732 |
|---|---|
| Length | 110 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 77.35 |
| Mean single sequence MFE | -30.22 |
| Consensus MFE | -20.79 |
| Energy contribution | -21.02 |
| Covariance contribution | 0.23 |
| Combinations/Pair | 1.26 |
| Mean z-score | -1.87 |
| Structure conservation index | 0.69 |
| SVM decision value | 1.32 |
| SVM RNA-class probability | 0.942065 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 17279622 110 - 20766785 AUACCCAUAUAUAGUCGUACAUAAAUGUCAGGCAGCCAGAAAGGAUAUUC--GCUGCAAUGGCAACGGCUGCUGGCAACUUAUUGGCUGCAGGACACACACAC------ACAAA--CGGA ....((.......(.....).....((((..((((((((.((((.....)--((((((.((....))..))).)))..))).))))))))..)))).......------.....--.)). ( -30.90) >DroPse_CAF1 4736 99 - 1 ACACAC--ACACGAAUGUGCAUAAAUGUCAUGUGAC----AAGGAUAUUUGUGCUGCAGUGGCAACGGCUGCUAGCAACUUAUUGGCUGCAGGACGCACACGC------AU--------- ......--....(..(((((.....((((....)))----).........((.((((((((....).((.....)).........))))))).)))))))..)------..--------- ( -29.60) >DroSec_CAF1 2660 118 - 1 AUACACAUACAUAGCCGUACAUAAAUGUCAGGCAGCCAGAAAGGGUAUUC--GCUGCAAUGGCAACGGCUGCUGGCAACUUAUUGGUUGCAGGAUACACACACACAAACACACAAGCGGA .........(((((((((.......(((((.(((((..(((......)))--)))))..)))))))))))).))((((((....)))))).............................. ( -30.01) >DroSim_CAF1 4629 110 - 1 AUACACAUACAUAGCCGUACAUAAAUGUCAGGCAGCCAGAAAGGAUAUUC--GCUGCAAUGGCAACGGCUGCUGGCAACUUAUUGGCUGCAGGACACACACAC------ACACAAGCG-- .........................((((..((((((((.((((.....)--((((((.((....))..))).)))..))).))))))))..)))).......------.........-- ( -30.60) >DroEre_CAF1 1014 95 - 1 -UACACAU----------ACAUAAAUGUCAGGCAGCCAGAAAGGAUAUUC--GCUGCAAUGGCAACGGCUGCUGGCAACUUAUUGGCUGCAGGACACACACAC------AC------GCA -.......----------.......((((..((((((((.((((.....)--((((((.((....))..))).)))..))).))))))))..)))).......------..------... ( -30.60) >DroPer_CAF1 4782 99 - 1 ACACAC--ACACGAAUGUGCAUAAAUGUCAUGUGAC----AAGGAUAUUUGUGCUGCAGUGGCAACGGCUGCUAGCAACUUAUUGGCUGCAGGACGCACACGC------AU--------- ......--....(..(((((.....((((....)))----).........((.((((((((....).((.....)).........))))))).)))))))..)------..--------- ( -29.60) >consensus AUACACAUACAUAGACGUACAUAAAUGUCAGGCAGCCAGAAAGGAUAUUC__GCUGCAAUGGCAACGGCUGCUGGCAACUUAUUGGCUGCAGGACACACACAC______AC______G__ .........................((((..((((((((.(((.........((((((.((....))..))).)))..))).))))))))..))))........................ (-20.79 = -21.02 + 0.23)
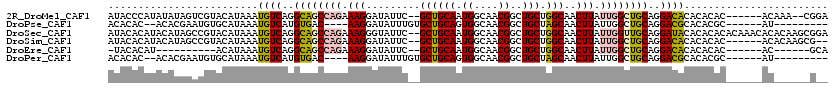

| Location | 17,279,654 – 17,279,772 |
|---|---|
| Length | 118 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 73.87 |
| Mean single sequence MFE | -38.28 |
| Consensus MFE | -15.63 |
| Energy contribution | -19.57 |
| Covariance contribution | 3.93 |
| Combinations/Pair | 1.14 |
| Mean z-score | -2.57 |
| Structure conservation index | 0.41 |
| SVM decision value | 1.84 |
| SVM RNA-class probability | 0.979354 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 17279654 118 + 20766785 AGUUGCCAGCAGCCGUUGCCAUUGCAGC--GAAUAUCCUUUCUGGCUGCCUGACAUUUAUGUACGACUAUAUAUGGGUAUGGGUAUGGGUAUGUGCGAGCAUGUGUGCGUGCACAUUCGC ...((((.(((((((((((....)))))--(((......))).)))))).....(((((((((......)))))))))...))))..((.(((((((.(((....))).))))))))).. ( -38.70) >DroPse_CAF1 4761 99 + 1 AGUUGCUAGCAGCCGUUGCCACUGCAGCACAAAUAUCCUU----GUCACAUGACAUUUAUGCACAUUCGUGU--GUGUGUGUCC-----UUCCUAC---------UG-CUGCACGUUGGC .((((....))))....(((((((((((((((......))----)).....(((((.(((((((....))))--))).))))).-----.......---------.)-))))).).)))) ( -30.90) >DroSec_CAF1 2700 113 + 1 AGUUGCCAGCAGCCGUUGCCAUUGCAGC--GAAUACCCUUUCUGGCUGCCUGACAUUUAUGUACGGCUAUGUAUGUGUAUGUGC-----GAUGUGCGAGCAUGUGUGCGUGCACAUUCGC ......(((((((((((((....)))))--(((......))).))))).)))((((.((((((((....)))))))).))))((-----((((((((.(((....))).)))))).)))) ( -43.60) >DroSim_CAF1 4661 113 + 1 AGUUGCCAGCAGCCGUUGCCAUUGCAGC--GAAUAUCCUUUCUGGCUGCCUGACAUUUAUGUACGGCUAUGUAUGUGUAUGUGC-----GAUGUGCGAGCAUGUGUGCGUGCACAUUCGC ......(((((((((((((....)))))--(((......))).))))).)))((((.((((((((....)))))))).))))((-----((((((((.(((....))).)))))).)))) ( -43.60) >DroEre_CAF1 1042 96 + 1 AGUUGCCAGCAGCCGUUGCCAUUGCAGC--GAAUAUCCUUUCUGGCUGCCUGACAUUUAUGU----------AUGUGUA------------UGUGCGCGCAUGUGUGCGUGCACAUUGGC ....(((((((((((((((....)))))--(((......))).))))))...((((......----------..))))(------------((((((((((....))))))))))))))) ( -42.00) >DroPer_CAF1 4807 99 + 1 AGUUGCUAGCAGCCGUUGCCACUGCAGCACAAAUAUCCUU----GUCACAUGACAUUUAUGCACAUUCGUGU--GUGUGUGUCC-----UUCCUAC---------UG-CUGCACGUUGGC .((((....))))....(((((((((((((((......))----)).....(((((.(((((((....))))--))).))))).-----.......---------.)-))))).).)))) ( -30.90) >consensus AGUUGCCAGCAGCCGUUGCCAUUGCAGC__GAAUAUCCUUUCUGGCUGCCUGACAUUUAUGUACGGCUAUGUAUGUGUAUGUGC_____UAUGUGCGAGCAUGUGUGCGUGCACAUUCGC ......(((((((((((((....)))))..(((......))).)))))).))((((.((((((((....)))))))).)))).........((((((.(((....))).))))))..... (-15.63 = -19.57 + 3.93)

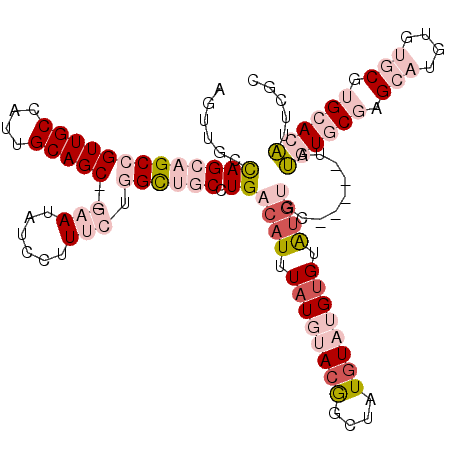
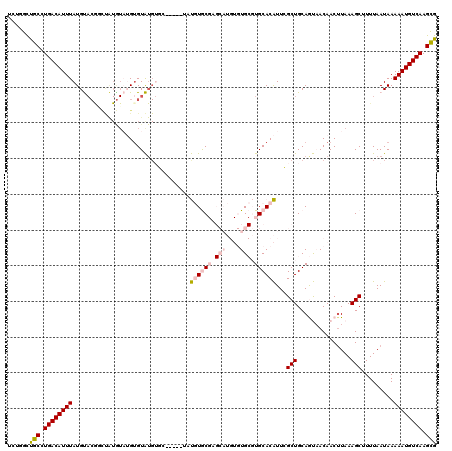
| Location | 17,279,654 – 17,279,772 |
|---|---|
| Length | 118 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 73.87 |
| Mean single sequence MFE | -33.19 |
| Consensus MFE | -14.94 |
| Energy contribution | -16.25 |
| Covariance contribution | 1.31 |
| Combinations/Pair | 1.17 |
| Mean z-score | -2.23 |
| Structure conservation index | 0.45 |
| SVM decision value | 1.28 |
| SVM RNA-class probability | 0.939893 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 17279654 118 - 20766785 GCGAAUGUGCACGCACACAUGCUCGCACAUACCCAUACCCAUACCCAUAUAUAGUCGUACAUAAAUGUCAGGCAGCCAGAAAGGAUAUUC--GCUGCAAUGGCAACGGCUGCUGGCAACU ((((((((((..(((....)))..))))))..........(((......)))..)))).......((((((.(((((.(((......)))--(((.....)))...)))))))))))... ( -32.20) >DroPse_CAF1 4761 99 - 1 GCCAACGUGCAG-CA---------GUAGGAA-----GGACACACAC--ACACGAAUGUGCAUAAAUGUCAUGUGAC----AAGGAUAUUUGUGCUGCAGUGGCAACGGCUGCUAGCAACU .......(((((-((---------((.(...-----.(...(((.(--((((((((((.(.....((((....)))----).).))))))))).))..))).)..).)))))).)))... ( -27.40) >DroSec_CAF1 2700 113 - 1 GCGAAUGUGCACGCACACAUGCUCGCACAUC-----GCACAUACACAUACAUAGCCGUACAUAAAUGUCAGGCAGCCAGAAAGGGUAUUC--GCUGCAAUGGCAACGGCUGCUGGCAACU ((((.(((((..(((....)))..)))))))-----))...........(((((((((.......(((((.(((((..(((......)))--)))))..)))))))))))).))...... ( -37.71) >DroSim_CAF1 4661 113 - 1 GCGAAUGUGCACGCACACAUGCUCGCACAUC-----GCACAUACACAUACAUAGCCGUACAUAAAUGUCAGGCAGCCAGAAAGGAUAUUC--GCUGCAAUGGCAACGGCUGCUGGCAACU ((((.(((((..(((....)))..)))))))-----))...........(((((((((.......(((((.(((((..(((......)))--)))))..)))))))))))).))...... ( -37.71) >DroEre_CAF1 1042 96 - 1 GCCAAUGUGCACGCACACAUGCGCGCACA------------UACACAU----------ACAUAAAUGUCAGGCAGCCAGAAAGGAUAUUC--GCUGCAAUGGCAACGGCUGCUGGCAACU (((.((((((.((((....)))).)))))------------)......----------............(((((((.(((......)))--(((.....)))...)))))))))).... ( -36.70) >DroPer_CAF1 4807 99 - 1 GCCAACGUGCAG-CA---------GUAGGAA-----GGACACACAC--ACACGAAUGUGCAUAAAUGUCAUGUGAC----AAGGAUAUUUGUGCUGCAGUGGCAACGGCUGCUAGCAACU .......(((((-((---------((.(...-----.(...(((.(--((((((((((.(.....((((....)))----).).))))))))).))..))).)..).)))))).)))... ( -27.40) >consensus GCCAAUGUGCACGCACACAUGCUCGCACAUA_____GCACAUACACAUACAUAGACGUACAUAAAUGUCAGGCAGCCAGAAAGGAUAUUC__GCUGCAAUGGCAACGGCUGCUGGCAACU .....(((((..((......))..)))))....................................(((((.((((((.(((......)))..(((.....)))...)))))))))))... (-14.94 = -16.25 + 1.31)

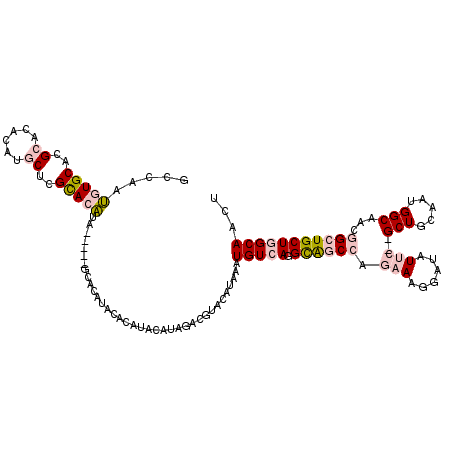
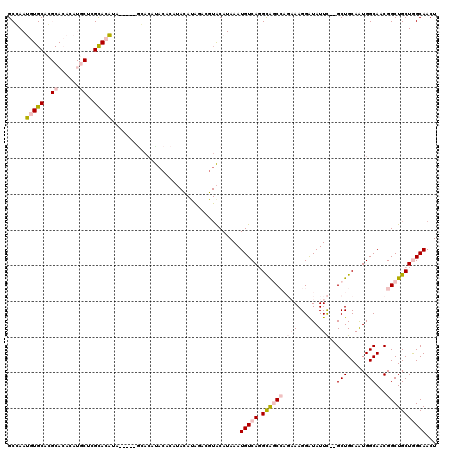
| Location | 17,279,692 – 17,279,812 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 74.51 |
| Mean single sequence MFE | -31.77 |
| Consensus MFE | -16.65 |
| Energy contribution | -17.29 |
| Covariance contribution | 0.64 |
| Combinations/Pair | 1.13 |
| Mean z-score | -2.18 |
| Structure conservation index | 0.52 |
| SVM decision value | 1.51 |
| SVM RNA-class probability | 0.960445 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 17279692 120 + 20766785 UCUGGCUGCCUGACAUUUAUGUACGACUAUAUAUGGGUAUGGGUAUGGGUAUGUGCGAGCAUGUGUGCGUGCACAUUCGCUGCAGUAACAACUUAAAGCUUUUAAUAAAAAUGUCAAGCG .......((.((((((((.(((....((((((....))))))(((.(.(.(((((((.(((....))).))))))).).)))).....................))).)))))))).)). ( -31.80) >DroPse_CAF1 4801 99 + 1 ----GUCACAUGACAUUUAUGCACAUUCGUGU--GUGUGUGUCC-----UUCCUAC---------UG-CUGCACGUUGGCUGCAACUACAACUUAAAGCUUUUAAUAAAAAUGUCAAGUG ----..(((.((((((((((((((((....))--))))))....-----.......---------.(-((....((((..........))))....))).........)))))))).))) ( -23.80) >DroSec_CAF1 2738 115 + 1 UCUGGCUGCCUGACAUUUAUGUACGGCUAUGUAUGUGUAUGUGC-----GAUGUGCGAGCAUGUGUGCGUGCACAUUCGCUGCAGUAACAACAUAAAGCUUUUAAUAAAAAUGUCAAGCG .......((.((((((((.(((..((((((((.(((...(((((-----((((((((.(((....))).)))))).)))).)))...)))))))..))))....))).)))))))).)). ( -39.70) >DroSim_CAF1 4699 115 + 1 UCUGGCUGCCUGACAUUUAUGUACGGCUAUGUAUGUGUAUGUGC-----GAUGUGCGAGCAUGUGUGCGUGCACAUUCGCUGCAGUAACAACUUAAAGCUUUUAAUAAAAAUGUCAAGCG .......((.((((((((.(((..((((.((..(((...(((((-----((((((((.(((....))).)))))).)))).)))...)))...)).))))....))).)))))))).)). ( -37.30) >DroEre_CAF1 1080 98 + 1 UCUGGCUGCCUGACAUUUAUGU----------AUGUGUA------------UGUGCGCGCAUGUGUGCGUGCACAUUGGCUGCAGUAACAACUUAAAGCUUUUAAUAAAAAUGUCAAGCG .......((.((((((((.(((----------(((((((------------((..(((....)))..))))))))).((((..(((....)))...))))....))).)))))))).)). ( -34.20) >DroPer_CAF1 4847 99 + 1 ----GUCACAUGACAUUUAUGCACAUUCGUGU--GUGUGUGUCC-----UUCCUAC---------UG-CUGCACGUUGGCUGCAACUACAACUUAAAGCUUUUAAUAAAAAUGUCAAGUG ----..(((.((((((((((((((((....))--))))))....-----.......---------.(-((....((((..........))))....))).........)))))))).))) ( -23.80) >consensus UCUGGCUGCCUGACAUUUAUGUACGGCUAUGUAUGUGUAUGUGC_____UAUGUGCGAGCAUGUGUGCGUGCACAUUCGCUGCAGUAACAACUUAAAGCUUUUAAUAAAAAUGUCAAGCG ......(((.((((((((.................................((((((.(((....))).))))))...(((...............))).........)))))))).))) (-16.65 = -17.29 + 0.64)
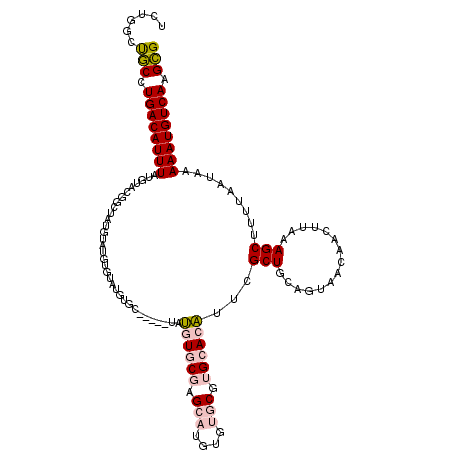
| Location | 17,279,692 – 17,279,812 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 74.51 |
| Mean single sequence MFE | -29.58 |
| Consensus MFE | -21.59 |
| Energy contribution | -21.57 |
| Covariance contribution | -0.03 |
| Combinations/Pair | 1.13 |
| Mean z-score | -2.88 |
| Structure conservation index | 0.73 |
| SVM decision value | 2.37 |
| SVM RNA-class probability | 0.993118 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 17279692 120 - 20766785 CGCUUGACAUUUUUAUUAAAAGCUUUAAGUUGUUACUGCAGCGAAUGUGCACGCACACAUGCUCGCACAUACCCAUACCCAUACCCAUAUAUAGUCGUACAUAAAUGUCAGGCAGCCAGA .(((((((((((....((...(((.((.(((((....)))))..((((((..(((....)))..))))))...................)).)))..))...)))))))))))....... ( -30.00) >DroPse_CAF1 4801 99 - 1 CACUUGACAUUUUUAUUAAAAGCUUUAAGUUGUAGUUGCAGCCAACGUGCAG-CA---------GUAGGAA-----GGACACACAC--ACACGAAUGUGCAUAAAUGUCAUGUGAC---- (((.((((((((..........((((..((((((((((....)))).)))))-).---------....)))-----)......(((--(......))))...)))))))).)))..---- ( -23.00) >DroSec_CAF1 2738 115 - 1 CGCUUGACAUUUUUAUUAAAAGCUUUAUGUUGUUACUGCAGCGAAUGUGCACGCACACAUGCUCGCACAUC-----GCACAUACACAUACAUAGCCGUACAUAAAUGUCAGGCAGCCAGA .(((((((((((....((...(((.((((((((....)))((((.(((((..(((....)))..)))))))-----))......)))))...)))..))...)))))))))))....... ( -35.70) >DroSim_CAF1 4699 115 - 1 CGCUUGACAUUUUUAUUAAAAGCUUUAAGUUGUUACUGCAGCGAAUGUGCACGCACACAUGCUCGCACAUC-----GCACAUACACAUACAUAGCCGUACAUAAAUGUCAGGCAGCCAGA .(((((((((((....((...(((......(((...((..((((.(((((..(((....)))..)))))))-----)).))...))).....)))..))...)))))))))))....... ( -32.70) >DroEre_CAF1 1080 98 - 1 CGCUUGACAUUUUUAUUAAAAGCUUUAAGUUGUUACUGCAGCCAAUGUGCACGCACACAUGCGCGCACA------------UACACAU----------ACAUAAAUGUCAGGCAGCCAGA .(((((((((((................(((((....)))))..((((((.((((....)))).)))))------------)......----------....)))))))))))....... ( -33.10) >DroPer_CAF1 4847 99 - 1 CACUUGACAUUUUUAUUAAAAGCUUUAAGUUGUAGUUGCAGCCAACGUGCAG-CA---------GUAGGAA-----GGACACACAC--ACACGAAUGUGCAUAAAUGUCAUGUGAC---- (((.((((((((..........((((..((((((((((....)))).)))))-).---------....)))-----)......(((--(......))))...)))))))).)))..---- ( -23.00) >consensus CGCUUGACAUUUUUAUUAAAAGCUUUAAGUUGUUACUGCAGCCAAUGUGCACGCACACAUGCUCGCACAUA_____GCACAUACACAUACAUAGACGUACAUAAAUGUCAGGCAGCCAGA .(((((((((((................(((((....)))))...(((((..((......))..))))).................................)))))))))))....... (-21.59 = -21.57 + -0.03)

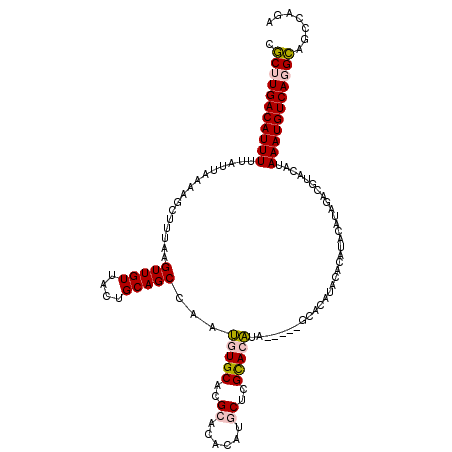
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:42:34 2006