
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 17,153,067 – 17,153,280 |
| Length | 213 |
| Max. P | 0.987024 |

| Location | 17,153,067 – 17,153,174 |
|---|---|
| Length | 107 |
| Sequences | 6 |
| Columns | 116 |
| Reading direction | forward |
| Mean pairwise identity | 79.39 |
| Mean single sequence MFE | -29.60 |
| Consensus MFE | -16.49 |
| Energy contribution | -17.50 |
| Covariance contribution | 1.01 |
| Combinations/Pair | 1.33 |
| Mean z-score | -2.43 |
| Structure conservation index | 0.56 |
| SVM decision value | 1.04 |
| SVM RNA-class probability | 0.905022 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
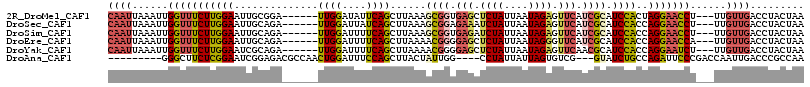
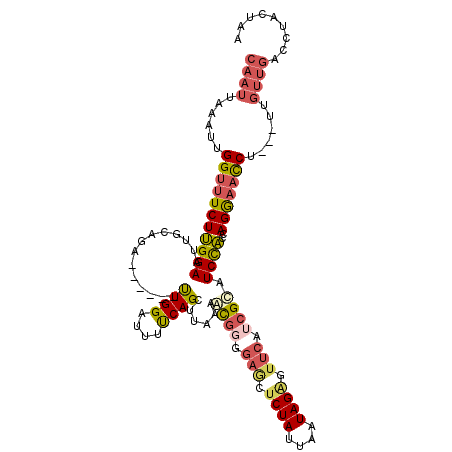
>2R_DroMel_CAF1 17153067 107 + 20766785 CAAUUAAAUUGGUUUCUUGGAAUUGCGGA------UUGGAUAUUCAGCUUAAAGCGGUGAGCUCUAUUAAUAGAGUUCAUCGCAUCCACUAGGAACCU---UUGUUGACCUACUAA ((((......(((((((((((...(((((------(.....)))).)).....(((((((((((((....))))))))))))).))))..))))))).---..))))......... ( -36.70) >DroSec_CAF1 20316 107 + 1 CAAUUAAAUUGGUUUCUUGGAAUUGCAGA------UUGGAUUAUCAGCUUAAAGCGGAGAAAUCUAUUAAUAGAGUUCAUCGCAUCCACCAGGAACCU---UUGUUGACCUACUAA ((((......(((((((((((...((.((------(......))).)).....(((..(((.((((....)))).)))..))).))))..))))))).---..))))......... ( -26.30) >DroSim_CAF1 20709 107 + 1 CAAUUAAAUUGGUUUCUUGGAAUUGCAGA------UUGGAUUUUCAGCUUAAAGCGGUGAGAUCUAUUAAUAGAGUUCAUCGCAUCCACCAGGAACCU---UUGUUGACCUACUAA ((((......(((((((((((...((.((------........)).)).....((((((((.((((....)))).)))))))).))))..))))))).---..))))......... ( -29.50) >DroEre_CAF1 21334 107 + 1 CAAUUAAAUUGGUUUCUUGGAAUUGCAGA------UUGGAUUUUCAGCUUAAAACGGGGAGCUCUAUUAAUAGGGUUCAUCGCAUCCACCAGGAACCA---UUGUUGACCUACUAA ((((.....((((((((((((..(((.((------........))..........(..((((((((....))))))))..))))))))..))))))))---..))))......... ( -27.90) >DroYak_CAF1 20404 107 + 1 CAAUUAAAUUGGUUUCUUGGAAUCGCAGA------UUGGAUUUUCAGCUUAAAACGGGGAGCUCUAUUAAUAGAGUUCAACGCAUCCACCAGGAAUCU---UUGUUGACCUACUAA ...((((...((.(((((((....((.((------........)).))......((..((((((((....))))))))..))......))))))).))---...))))........ ( -26.10) >DroAna_CAF1 25347 100 + 1 ---------GGGCUUCUCGGAAUCGGAGACGCCAACUGGAUUUCCAGCUUACUAUUGG----CCUAUUAUUAGUGUCG---GUAUCUGCCAGAUUCCCGACCAAUUGACCCGCCAA ---------((((...(((((((((((((..((..((((....)))).........((----((((....))).))))---)..))).)).)))).))))......).)))..... ( -31.10) >consensus CAAUUAAAUUGGUUUCUUGGAAUUGCAGA______UUGGAUUUUCAGCUUAAAACGGGGAGCUCUAUUAAUAGAGUUCAUCGCAUCCACCAGGAACCU___UUGUUGACCUACUAA ((((......(((((((((((..............((((....))))......((((.(((.((((....)))).))).)))).))))..)))))))......))))......... (-16.49 = -17.50 + 1.01)
| Location | 17,153,174 – 17,153,280 |
|---|---|
| Length | 106 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 74.26 |
| Mean single sequence MFE | -30.71 |
| Consensus MFE | -18.39 |
| Energy contribution | -19.87 |
| Covariance contribution | 1.48 |
| Combinations/Pair | 1.43 |
| Mean z-score | -2.05 |
| Structure conservation index | 0.60 |
| SVM decision value | 1.68 |
| SVM RNA-class probability | 0.971608 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
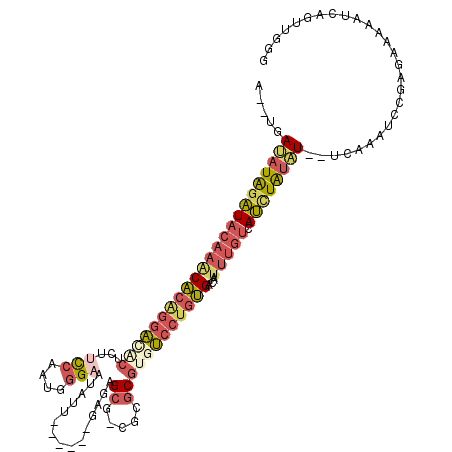
>2R_DroMel_CAF1 17153174 106 + 20766785 A--UGAUAUAGAUACAAACACAGGACACU---CCAAAGGGAAUAUU------GAGAGAG-CGCGCGUGUCCUGUGACACUUUGUCAUCUAUAU--UCAAAUCCGAGAAAAAUCAGUUGGG .--((((((((((((((((((((((((((---((....)))...((------....)).-.....))))))))))....))))).))))))).--)))...((((((....))..)))). ( -31.50) >DroSec_CAF1 20423 109 + 1 A--UGAUAUAGAUACAAACACAGGACACUCUUCCAAUGGGAAUAUU------GAGAGCG-CGCGCGUGUCCUGUGACAAUUUGUCAUCUAUAU--UCAAAUCCGAGAAAAAUCAGUUGGG .--(((((((((((((((((((((((((...(((((((....))))------).))((.-...))))))))))))....))))).))))))).--)))...((((((....))..)))). ( -34.50) >DroSim_CAF1 20816 109 + 1 A--UGAUAUAGAUACAAACACAGGACACUCUUCCAAUGGGAAUAUU------GAGAGCG-CGCGCGUGUCCUGUGACAAUUUGUCAUCUAUAU--UCAAAUCCGAGAAAAAUCAGUUGGG .--(((((((((((((((((((((((((...(((((((....))))------).))((.-...))))))))))))....))))).))))))).--)))...((((((....))..)))). ( -34.50) >DroEre_CAF1 21441 110 + 1 A--AGAUAUACAUACAAGCCCGGGAAGCUCAUUCAACGAGAAUAUU------GAGAGCUACGCGCGUGUCCUGGGACAAUUUGUCAUUUAUAU--UCUAAUCCGACAAAAAUCAGUUGGG .--.((((((.(((((((((((((((((((..((((........))------)))))))((....)).)))))))....))))).)).)))))--).....(((((........))))). ( -27.90) >DroYak_CAF1 20511 95 + 1 A--AGACAUGGAUACAAACACAGGA---------------CAUAUU------GAGAGCGACGCGCGUGUCCUGUGACAAUUUGUCAUUUAUAU--UUAAAUCGGAGAAAAAUCAGUUGGG .--.(((((((((((((((((((((---------------(((...------....(((...)))))))))))))....))))).))))))..--........((......)).)))... ( -21.01) >DroAna_CAF1 25447 117 + 1 AGGUGACCUAGGUC---UCCCAGUUUCCUGUUCCCUGGGGAUUCUUCGUAAAAAUAGCUGGGCGCGUGGCGCAGGCCAAUUUGUCACCUGAGUCCUGAAAUCCGAGGAAAUCCAGUAGGA (((((((...((((---(.((((((......(((....)))..............))))))((((...))))))))).....)))))))...((((........))))..(((....))) ( -34.85) >consensus A__UGAUAUAGAUACAAACACAGGACACUCUUCCAAUGGGAAUAUU______GAGAGCG_CGCGCGUGUCCUGUGACAAUUUGUCAUCUAUAU__UCAAAUCCGAGAAAAAUCAGUUGGG .....(((((((((((((((((((((((...(((....)))...............((.....))))))))))))....))))).))))))))........................... (-18.39 = -19.87 + 1.48)

| Location | 17,153,174 – 17,153,280 |
|---|---|
| Length | 106 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 74.26 |
| Mean single sequence MFE | -30.78 |
| Consensus MFE | -14.19 |
| Energy contribution | -15.58 |
| Covariance contribution | 1.40 |
| Combinations/Pair | 1.31 |
| Mean z-score | -2.58 |
| Structure conservation index | 0.46 |
| SVM decision value | 2.06 |
| SVM RNA-class probability | 0.987024 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 17153174 106 - 20766785 CCCAACUGAUUUUUCUCGGAUUUGA--AUAUAGAUGACAAAGUGUCACAGGACACGCGCG-CUCUCUC------AAUAUUCCCUUUGG---AGUGUCCUGUGUUUGUAUCUAUAUCA--U .....((((......))))...(((--.((((((((.((((....(((((((((((....-)......------.....(((....))---))))))))))))))))))))))))))--. ( -31.00) >DroSec_CAF1 20423 109 - 1 CCCAACUGAUUUUUCUCGGAUUUGA--AUAUAGAUGACAAAUUGUCACAGGACACGCGCG-CGCUCUC------AAUAUUCCCAUUGGAAGAGUGUCCUGUGUUUGUAUCUAUAUCA--U .....((((......))))...(((--.((((((((.((((....((((((((((((...-.))....------....((((....))))..)))))))))))))))))))))))))--. ( -35.00) >DroSim_CAF1 20816 109 - 1 CCCAACUGAUUUUUCUCGGAUUUGA--AUAUAGAUGACAAAUUGUCACAGGACACGCGCG-CGCUCUC------AAUAUUCCCAUUGGAAGAGUGUCCUGUGUUUGUAUCUAUAUCA--U .....((((......))))...(((--.((((((((.((((....((((((((((((...-.))....------....((((....))))..)))))))))))))))))))))))))--. ( -35.00) >DroEre_CAF1 21441 110 - 1 CCCAACUGAUUUUUGUCGGAUUAGA--AUAUAAAUGACAAAUUGUCCCAGGACACGCGCGUAGCUCUC------AAUAUUCUCGUUGAAUGAGCUUCCCGGGCUUGUAUGUAUAUCU--U .....(((((....)))))...(((--.((((....((((..((((....)))).((.((.(((((((------(((......)))))..)))))...)).))))))...)))))))--. ( -27.40) >DroYak_CAF1 20511 95 - 1 CCCAACUGAUUUUUCUCCGAUUUAA--AUAUAAAUGACAAAUUGUCACAGGACACGCGCGUCGCUCUC------AAUAUG---------------UCCUGUGUUUGUAUCCAUGUCU--U ..................(((....--(((((((((((.....)))((((((((.(((...)))....------....))---------------))))))))))))))....))).--. ( -19.50) >DroAna_CAF1 25447 117 - 1 UCCUACUGGAUUUCCUCGGAUUUCAGGACUCAGGUGACAAAUUGGCCUGCGCCACGCGCCCAGCUAUUUUUACGAAGAAUCCCCAGGGAACAGGAAACUGGGA---GACCUAGGUCACCU (((....)))((((((.((..(((.((.(.(((((.(.....).))))).)))..((.....))............)))..)).))))))(((....)))((.---(((....))).)). ( -36.80) >consensus CCCAACUGAUUUUUCUCGGAUUUGA__AUAUAGAUGACAAAUUGUCACAGGACACGCGCG_CGCUCUC______AAUAUUCCCAUUGGAAGAGUGUCCUGUGUUUGUAUCUAUAUCA__U .....((((......))))........(((((((((.((((....(((((((...((.....))...............(((....)))......))))))))))))))))))))..... (-14.19 = -15.58 + 1.40)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:40:59 2006