
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 17,128,225 – 17,128,345 |
| Length | 120 |
| Max. P | 0.663808 |

| Location | 17,128,225 – 17,128,345 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 96.08 |
| Mean single sequence MFE | -37.68 |
| Consensus MFE | -36.44 |
| Energy contribution | -36.12 |
| Covariance contribution | -0.32 |
| Combinations/Pair | 1.11 |
| Mean z-score | -1.10 |
| Structure conservation index | 0.97 |
| SVM decision value | 0.23 |
| SVM RNA-class probability | 0.646367 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
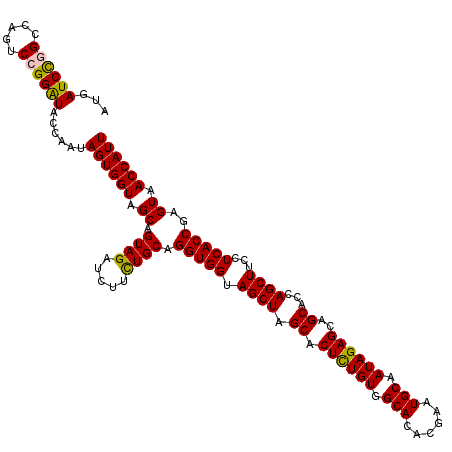
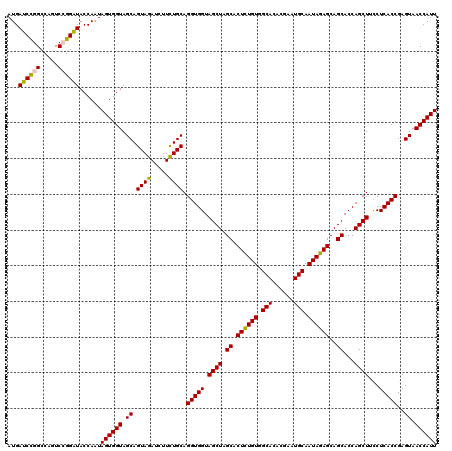
>2R_DroMel_CAF1 17128225 120 + 20766785 AUGAUCCGGCCAGUCGGGAUACCAAUAGUGGUAGCAGUAGAUUUUCUGCAGGUGGUAGCUAGCACUCUGUGGCACACGAAUGCAAUAGAGCAGCACCAGCUUUCUCACCGAGUAACCAUU ....(((((....)))))........((((((.((.((((.....)))).(((((.((((.((.((((((.(((......))).))))))..))...))))...)))))..)).)))))) ( -37.80) >DroSec_CAF1 64118 120 + 1 AUGAUCCGGCCAGUCCGGAUACCAAUAGUGGUAGCAGUAGAUCUUCUGCAGGUGGUAGCUAGCACUCUGUGGCACACGGAUGCAAUAGAGCAGCACCAGCUUCCUCACCGAGUAACCAUU ...((((((.....))))))......((((((.((.((((.....)))).(((((.((((.((.((((((.(((......))).))))))..))...))))...)))))..)).)))))) ( -40.60) >DroSim_CAF1 54963 120 + 1 AUGAUCCGGCCAGUCCGGAUACCAAUAGUGGUAGCAGUAGAUCUUCUGCAGGUGGUAGCUAGCACUCUGUGGCACACGAAUGCAAUAGAGCAGCACCAGCUUCCUCACCGAGUAACCAUU ...((((((.....))))))......((((((.((.((((.....)))).(((((.((((.((.((((((.(((......))).))))))..))...))))...)))))..)).)))))) ( -40.60) >DroEre_CAF1 69646 120 + 1 AUGAUCCGGCCAGUCCGGAUACCAGUAGUGGUAGCAGUAGAUCUUUUGCAGGUGGUAGCUAGCACUCUGUGGCACACGGAUGCAAUAGAGCAGCACCAGCUUUCUCACCGAGUAACCAUU ...((((((.....))))))......((((((.((((........)))).(((((.((((.((.((((((.(((......))).))))))..))...))))...))))).....)))))) ( -37.90) >DroYak_CAF1 71540 120 + 1 AUGAUCUGGCCAGUCAGGGUACCAAUAGUGGUAGCAGUAGAUCUUUUGCAGGUGGUAGCUAGCACUUUGUGGCACACGUAUGCAAUAGAGCAGCACCAGCUUCCUCACCGAGUAACCAUU ..(((((((((......)))((((....)))).....))))))..((((.(((((.((((.((.((((((.(((......))).))))))..))...))))...)))))..))))..... ( -31.50) >consensus AUGAUCCGGCCAGUCCGGAUACCAAUAGUGGUAGCAGUAGAUCUUCUGCAGGUGGUAGCUAGCACUCUGUGGCACACGAAUGCAAUAGAGCAGCACCAGCUUCCUCACCGAGUAACCAUU ...((((((.....))))))......((((((.((.((((.....)))).(((((.((((.((.((((((.(((......))).))))))..))...))))...)))))..)).)))))) (-36.44 = -36.12 + -0.32)

| Location | 17,128,225 – 17,128,345 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 96.08 |
| Mean single sequence MFE | -38.18 |
| Consensus MFE | -35.62 |
| Energy contribution | -36.06 |
| Covariance contribution | 0.44 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.17 |
| Structure conservation index | 0.93 |
| SVM decision value | 0.27 |
| SVM RNA-class probability | 0.663808 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
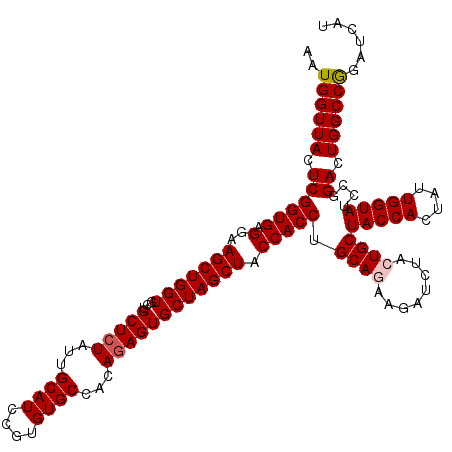
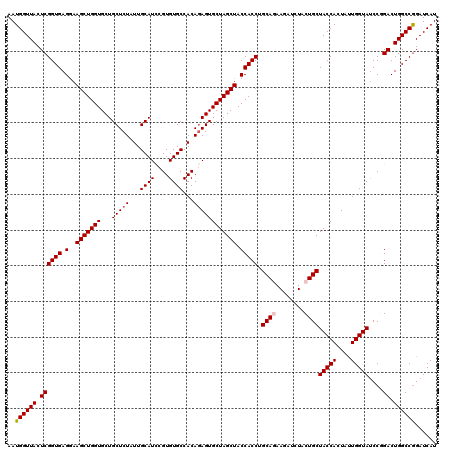
>2R_DroMel_CAF1 17128225 120 - 20766785 AAUGGUUACUCGGUGAGAAAGCUGGUGCUGCUCUAUUGCAUUCGUGUGCCACAGAGUGCUAGCUACCACCUGCAGAAAAUCUACUGCUACCACUAUUGGUAUCCCGACUGGCCGGAUCAU ..((((((.((((.((...(((((((...(((((...((((....))))...)))))))))))).......((((........))))(((((....))))))))))).))))))...... ( -39.20) >DroSec_CAF1 64118 120 - 1 AAUGGUUACUCGGUGAGGAAGCUGGUGCUGCUCUAUUGCAUCCGUGUGCCACAGAGUGCUAGCUACCACCUGCAGAAGAUCUACUGCUACCACUAUUGGUAUCCGGACUGGCCGGAUCAU ..((((.....((((.(..(((((((...(((((...((((....))))...)))))))))))).))))).((((........)))).))))......(.((((((.....))))))).. ( -39.40) >DroSim_CAF1 54963 120 - 1 AAUGGUUACUCGGUGAGGAAGCUGGUGCUGCUCUAUUGCAUUCGUGUGCCACAGAGUGCUAGCUACCACCUGCAGAAGAUCUACUGCUACCACUAUUGGUAUCCGGACUGGCCGGAUCAU ..((((.....((((.(..(((((((...(((((...((((....))))...)))))))))))).))))).((((........)))).))))......(.((((((.....))))))).. ( -40.20) >DroEre_CAF1 69646 120 - 1 AAUGGUUACUCGGUGAGAAAGCUGGUGCUGCUCUAUUGCAUCCGUGUGCCACAGAGUGCUAGCUACCACCUGCAAAAGAUCUACUGCUACCACUACUGGUAUCCGGACUGGCCGGAUCAU .((((((...((((.((..(((....)))((......)).((((..(((((...((((.((((......((.....)).......)))).))))..)))))..)))))).)))))))))) ( -37.52) >DroYak_CAF1 71540 120 - 1 AAUGGUUACUCGGUGAGGAAGCUGGUGCUGCUCUAUUGCAUACGUGUGCCACAAAGUGCUAGCUACCACCUGCAAAAGAUCUACUGCUACCACUAUUGGUACCCUGACUGGCCAGAUCAU ..((((((.((((.(.((.((((((..((((......))....(((...)))..))..)))))).))....(((..........)))(((((....)))))).)))).))))))...... ( -34.60) >consensus AAUGGUUACUCGGUGAGGAAGCUGGUGCUGCUCUAUUGCAUCCGUGUGCCACAGAGUGCUAGCUACCACCUGCAGAAGAUCUACUGCUACCACUAUUGGUAUCCGGACUGGCCGGAUCAU ..((((((.((((((.(..(((((((...(((((...((((....))))...)))))))))))).))))).((((........))))(((((....)))))....)).))))))...... (-35.62 = -36.06 + 0.44)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:40:31 2006