
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 2,808,967 – 2,809,075 |
| Length | 108 |
| Max. P | 0.834281 |

| Location | 2,808,967 – 2,809,075 |
|---|---|
| Length | 108 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 79.75 |
| Mean single sequence MFE | -40.92 |
| Consensus MFE | -23.52 |
| Energy contribution | -22.88 |
| Covariance contribution | -0.64 |
| Combinations/Pair | 1.23 |
| Mean z-score | -2.19 |
| Structure conservation index | 0.57 |
| SVM decision value | 0.73 |
| SVM RNA-class probability | 0.834281 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 2808967 108 + 20766785 ACCGUGAUGGGCAUGUC---CUCCACUUAUGUGCCCAUGUCCAUGGACAGUUCGGAGA------GCCUUGGUCUGGAUCCCUCCACCGAGGAGGUGAUCUUCAUGACCGCCAAUCGA .(((..((((((((((.---........))))))))))(((....)))....))).((------....(((((.((((((((((.....))))).)))))....)))))....)).. ( -41.90) >DroVir_CAF1 1313 117 + 1 ACCGUGAUGGGCAUGUCCUCGUCCAGCUAUGUGCCCAUGUCCAUGGAGAGUGGUGAGGGACUUAGCCACGAUCUGGAUCCCUCCACCGAGGAGGUCAUCUUCAUGACAGCCAAUCGC ...(((((.(((...(((((......(((((..(....)..))))).....(((((((((..(((.......)))..))))).))))))))).(((((....))))).))).))))) ( -43.00) >DroGri_CAF1 1339 102 + 1 ACCGUCAUGGGUAUAUC---CUCCAGUUAUGUGCCCAUGUCCAUGGACAGCG------------GCCACGAUCUGGAUCCCUCUACCGAGGAGGUCAUAUUCAUGACGGCAAAUCGC .(((((((((((((..(---(((((((..((.(((..((((....))))..)------------))))..).)))))..((((....)))).))..)))))))))))))........ ( -45.40) >DroWil_CAF1 22791 108 + 1 ACCGUCAUGGGCAUGUC---CUCGACUUAUGUGCCCAUGUCCAUGGAUAGCGCAGAGA------GUUUAGAUUUGGAUCCCUCAACUGAGGAAGUCAUCUUUAUGACAGCCAAUCGC .((((((((((((((((---...)))....)))))))))...))))............------.....((((.((...((((....))))..(((((....)))))..)))))).. ( -31.70) >DroMoj_CAF1 1288 114 + 1 ACCGUUAUGGGCAUGUC---GUCGAGCUAUGUGCCCAUGUCCAUGGAUAGCGCUGCAGAUGCUAGCCACGAUCUUGAUCCCUCCACGGAGGAGGUCAUCUUUAUGACAGCCAAUCGC .........(((.((((---((((.(((((....((((....)))))))))(((((....)).)))..))))..(((((((((....)))).))))).......)))))))...... ( -38.20) >DroAna_CAF1 15505 108 + 1 ACGGUAAUGGGCAUGUC---CUCCACGUACGUGCCCAUGUCCAUGGACAGUUCCGAAG------GCAUCGGUCUGGAUCCCUCCACCGAGGAGGUGAUCUUCAUGACGGCGAAUCGC .((((.((((((((((.---(.....).))))))))))((((((((((((..((((..------...)))).)))(((((((((.....))))).))))))))))..)))..)))). ( -45.30) >consensus ACCGUCAUGGGCAUGUC___CUCCACUUAUGUGCCCAUGUCCAUGGACAGCGCUGAGG______GCCACGAUCUGGAUCCCUCCACCGAGGAGGUCAUCUUCAUGACAGCCAAUCGC ......((((((((((............))))))))))(((.(((((......................(((....)))(((((.....))))).....)))))))).......... (-23.52 = -22.88 + -0.64)

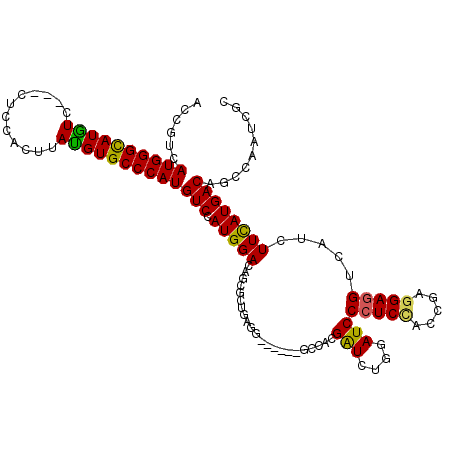
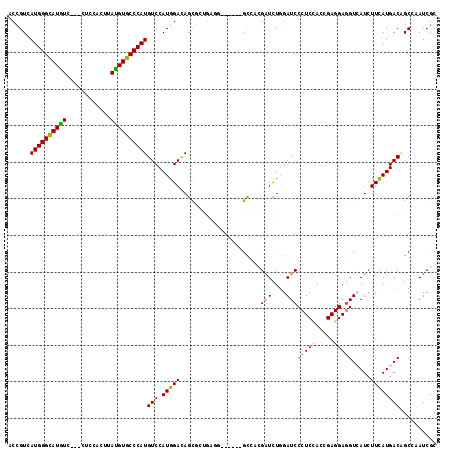
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:40:55 2006