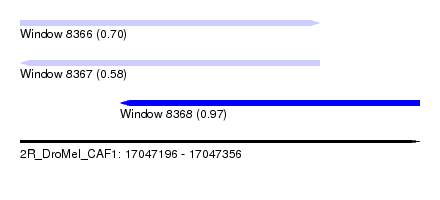
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 17,047,196 – 17,047,356 |
| Length | 160 |
| Max. P | 0.967486 |

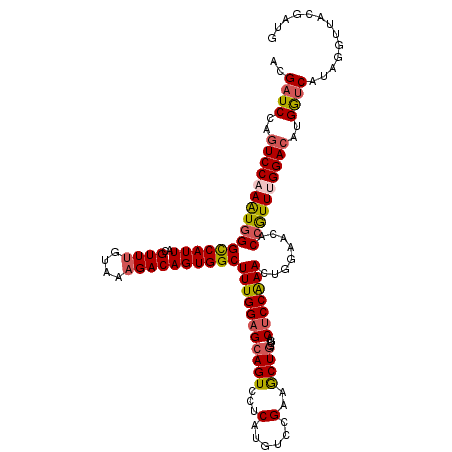
| Location | 17,047,196 – 17,047,316 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 90.75 |
| Mean single sequence MFE | -38.78 |
| Consensus MFE | -31.10 |
| Energy contribution | -31.74 |
| Covariance contribution | 0.64 |
| Combinations/Pair | 1.17 |
| Mean z-score | -1.53 |
| Structure conservation index | 0.80 |
| SVM decision value | 0.34 |
| SVM RNA-class probability | 0.695709 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
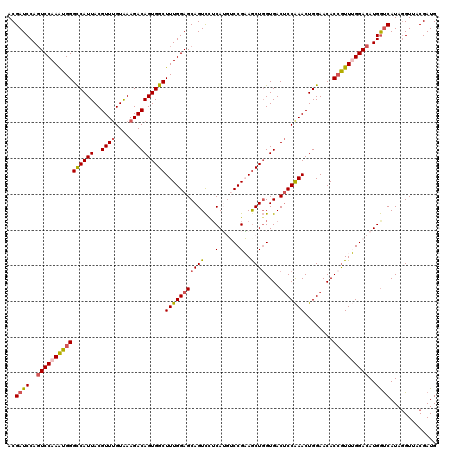
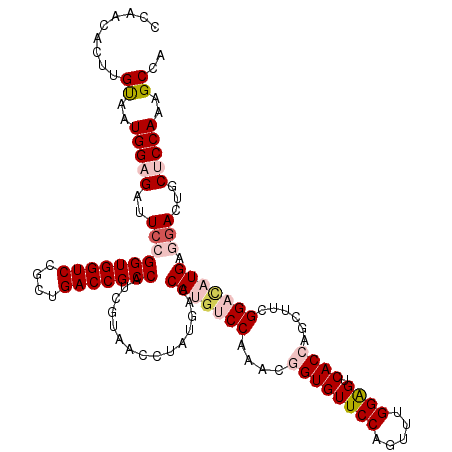
>2R_DroMel_CAF1 17047196 120 + 20766785 ACGAUCCAGUCCAAGUGGGUCAUUACGUUUGUAAAGACAGUGGCUUUGGAGCAGUCCGCAUGUCCGAAACUGGUGACCCCAAACUGGAACACCAUUUGGACAUGGUCAUAGGUCACGAUG ..((((((.......)))))).....((((....)))).(((((((.(((....))).((((((((((..(((((..((......))..))))))))))))))).....))))))).... ( -40.70) >DroSec_CAF1 140752 120 + 1 ACGAUCCAGUCCAAAUGGGCCAUUACGUUUGUAAAGACAGUGGCUUUGGAGCAGUCCUCAUGUCCGAAGCUGGUGACUCCAAACUGGAACACAGUUUGGACAUGGUCAUAUGUUACGAUG (((.....((((....)))).....)))((((((.......(((((((((.((.......))))))))))).((((((((((((((.....)))))))))....)))))...)))))).. ( -38.90) >DroSim_CAF1 149291 120 + 1 ACGAUCCAGUCCAAAUGGGCCAUUACGUUUGUAAAGACAGUGGCUUUGGAGCAGUCCUCAUGUCCGAAGCUGGUGACUCCAAACUGGAACACCGUUUGGACAUGGUCAUAGGUUACGAUG ..((.(((((((((((((.(((....(((((...((.((.((((((((((.((.......)))))))))))).)).)).))))))))....)))))))))).)))))............. ( -43.40) >DroEre_CAF1 144800 120 + 1 ACGAUCCACUCCAAAUGGGCCAUUACGUUGGUGAAGACAGUGGCUUUGGAGCAGUUUUCAGCACCGUAGCUUGUGACUCCAAACUGGAACACCGUUGGGACAUGACCAUAGGUGACGAUG .((.(((.((((((...(((((((..(((......)))))))))))))))).(((((..(((((........))).))..)))))))).((((..(((.......)))..)))).))... ( -34.70) >DroYak_CAF1 154280 120 + 1 ACGAUCCAGUCCAAAUGGGCCAUUACGUUUGUAAAGACAGUGGCUUUGGAGCAGUCUUCAAAACCGUAGCUGGUGACUCCGAACUGGAACACCGUUGGGACAUGGUCAUAGGUAACGAUG ..((.(((((((.(((((((((((((((((....))))...((.(((((((....))))))).)))))).))))...(((.....)))...))))).)))).)))))............. ( -36.20) >consensus ACGAUCCAGUCCAAAUGGGCCAUUACGUUUGUAAAGACAGUGGCUUUGGAGCAGUCCUCAUGUCCGAAGCUGGUGACUCCAAACUGGAACACCGUUUGGACAUGGUCAUAGGUUACGAUG ..((((..((((((((((((((((..((((....))))))))))(((((((((((...(......)..))))....)))))))........))))))))))..))))............. (-31.10 = -31.74 + 0.64)

| Location | 17,047,196 – 17,047,316 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 90.75 |
| Mean single sequence MFE | -35.56 |
| Consensus MFE | -28.44 |
| Energy contribution | -28.84 |
| Covariance contribution | 0.40 |
| Combinations/Pair | 1.16 |
| Mean z-score | -1.42 |
| Structure conservation index | 0.80 |
| SVM decision value | 0.10 |
| SVM RNA-class probability | 0.584851 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 17047196 120 - 20766785 CAUCGUGACCUAUGACCAUGUCCAAAUGGUGUUCCAGUUUGGGGUCACCAGUUUCGGACAUGCGGACUGCUCCAAAGCCACUGUCUUUACAAACGUAAUGACCCACUUGGACUGGAUCGU ...........(((((((.((((((.(((....))).....((((((...((((.(((((.(.((.((.......)))).))))))....))))....))))))..))))))))).)))) ( -35.60) >DroSec_CAF1 140752 120 - 1 CAUCGUAACAUAUGACCAUGUCCAAACUGUGUUCCAGUUUGGAGUCACCAGCUUCGGACAUGAGGACUGCUCCAAAGCCACUGUCUUUACAAACGUAAUGGCCCAUUUGGACUGGAUCGU ...........(((((((.((((((...(((..((((((((((((.....))))))))).(((((((.(((....)))....))))))).........)))..)))))))))))).)))) ( -35.30) >DroSim_CAF1 149291 120 - 1 CAUCGUAACCUAUGACCAUGUCCAAACGGUGUUCCAGUUUGGAGUCACCAGCUUCGGACAUGAGGACUGCUCCAAAGCCACUGUCUUUACAAACGUAAUGGCCCAUUUGGACUGGAUCGU ...........(((((((.(((((((.((((((((.....)))).)))).(((.((....(((((((.(((....)))....)))))))....))....)))...)))))))))).)))) ( -36.80) >DroEre_CAF1 144800 120 - 1 CAUCGUCACCUAUGGUCAUGUCCCAACGGUGUUCCAGUUUGGAGUCACAAGCUACGGUGCUGAAAACUGCUCCAAAGCCACUGUCUUCACCAACGUAAUGGCCCAUUUGGAGUGGAUCGU ((((((((((..(((.......)))..))))....((((((......)))))))))))).......(..(((((((((((.(((........)))...))))...)))))))..)..... ( -33.90) >DroYak_CAF1 154280 120 - 1 CAUCGUUACCUAUGACCAUGUCCCAACGGUGUUCCAGUUCGGAGUCACCAGCUACGGUUUUGAAGACUGCUCCAAAGCCACUGUCUUUACAAACGUAAUGGCCCAUUUGGACUGGAUCGU (((((((......(((...)))..))))))).(((((((((((....(((..((((.((.(((((((.(((....)))....))))))).)).)))).)))....))))))))))).... ( -36.20) >consensus CAUCGUAACCUAUGACCAUGUCCAAACGGUGUUCCAGUUUGGAGUCACCAGCUUCGGACAUGAGGACUGCUCCAAAGCCACUGUCUUUACAAACGUAAUGGCCCAUUUGGACUGGAUCGU ...........(((((((.(((((((.((((((((.....)))).)))).(((.((....(((((((.(((....)))....)))))))....))....)))...)))))))))).)))) (-28.44 = -28.84 + 0.40)

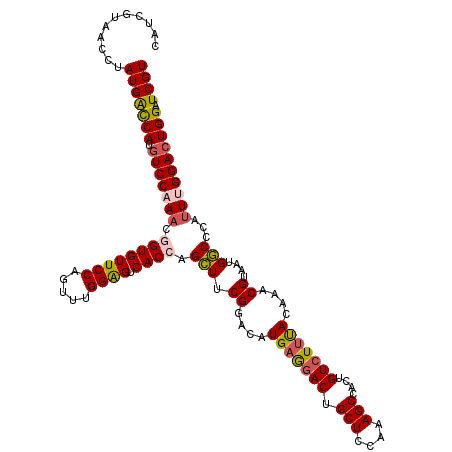

| Location | 17,047,236 – 17,047,356 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 89.58 |
| Mean single sequence MFE | -45.16 |
| Consensus MFE | -33.24 |
| Energy contribution | -35.12 |
| Covariance contribution | 1.88 |
| Combinations/Pair | 1.09 |
| Mean z-score | -2.98 |
| Structure conservation index | 0.74 |
| SVM decision value | 1.61 |
| SVM RNA-class probability | 0.967486 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 17047236 120 - 20766785 CCAACACUUGUAAUGGAGAUUCCGGUGGUCCGCUGACCGCCAUCGUGACCUAUGACCAUGUCCAAAUGGUGUUCCAGUUUGGGGUCACCAGUUUCGGACAUGCGGACUGCUCCAAAGCCA .............(((((..((((((((((....)))))))...............(((((((((((((((((((.....)))).))))).))).))))))).)))...)))))...... ( -45.20) >DroSec_CAF1 140792 120 - 1 CCAACAAUUGCAAUGGAGAUUCCGGUGGUCCGCUGACCGCCAUCGUAACAUAUGACCAUGUCCAAACUGUGUUCCAGUUUGGAGUCACCAGCUUCGGACAUGAGGACUGCUCCAAAGCCA .........((..(((((..((((((((((....))))))).(((((...))))).(((((((((((((.....))))))(((((.....)))))))))))).)))...)))))..)).. ( -45.50) >DroSim_CAF1 149331 120 - 1 CCAACACUUGCAAUGGAGAUUCCGGUGGUCCGCUGACCGCCAUCGUAACCUAUGACCAUGUCCAAACGGUGUUCCAGUUUGGAGUCACCAGCUUCGGACAUGAGGACUGCUCCAAAGCCA .........((..(((((..((((((((((....))))))).(((((...))))).(((((((.(((((((((((.....)))).)))).).)).))))))).)))...)))))..)).. ( -45.90) >DroEre_CAF1 144840 120 - 1 CCAGCACUUGUAGUGGCGAUUCCGGUGGUCCUCUGACCGCCAUCGUCACCUAUGGUCAUGUCCCAACGGUGUUCCAGUUUGGAGUCACAAGCUACGGUGCUGAAAACUGCUCCAAAGCCA .(((((((.(((((.(.(((((((((((((....))))))).....((((..(((.......)))..)))).........)))))).)..))))))))))))......(((....))).. ( -45.50) >DroYak_CAF1 154320 120 - 1 CCAGCACUUGUAGUGGCGAUUCCGGUGGUCCUCUGACCGCCAUCGUUACCUAUGACCAUGUCCCAACGGUGUUCCAGUUCGGAGUCACCAGCUACGGUUUUGAAGACUGCUCCAAAGCCA ..(((..(..((((((((((...(((((((....)))))))))))))).)))..)............((((((((.....)))).)))).)))..(((((((.((....)).))))))). ( -43.70) >consensus CCAACACUUGUAAUGGAGAUUCCGGUGGUCCGCUGACCGCCAUCGUAACCUAUGACCAUGUCCAAACGGUGUUCCAGUUUGGAGUCACCAGCUUCGGACAUGAGGACUGCUCCAAAGCCA .........((..(((((..((((((((((....)))))))...............(((((((....((((((((.....)))).))))......))))))).)))...)))))..)).. (-33.24 = -35.12 + 1.88)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:39:54 2006