
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 16,699,111 – 16,699,271 |
| Length | 160 |
| Max. P | 0.864339 |

| Location | 16,699,111 – 16,699,231 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 78.78 |
| Mean single sequence MFE | -45.07 |
| Consensus MFE | -29.63 |
| Energy contribution | -29.83 |
| Covariance contribution | 0.20 |
| Combinations/Pair | 1.37 |
| Mean z-score | -1.79 |
| Structure conservation index | 0.66 |
| SVM decision value | 0.84 |
| SVM RNA-class probability | 0.864339 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
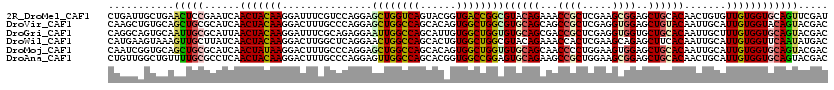
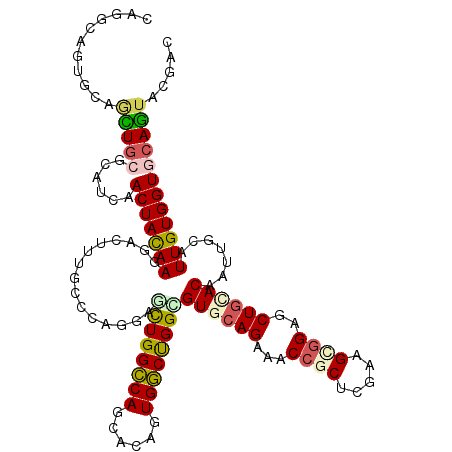
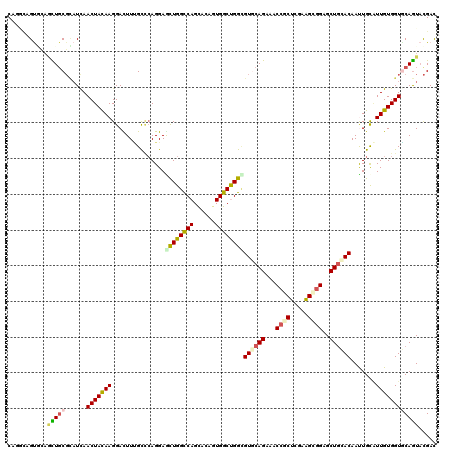
>2R_DroMel_CAF1 16699111 120 + 20766785 CUGAUUGCUGAACUCCGAAUCAACUACAAGGAUUUCGUCCAGGAGCUGGUCAGUACGGUGACCGGCGUACAGAAACCGCUCGAAGCGGAGCUGCACAACUGUGUUGUGGUGCAGUUCGAU (((.(((.((((.(((.............))).))))..)))..((((((((......))))))))...)))....(((.....)))(((((((((.((......)).)))))))))... ( -40.92) >DroVir_CAF1 7645 120 + 1 CAAGCUGUGCAGCUGCGCAUCAACUACAAGGACUUUGCCCAGGAGCUGGCCAGCACAGUGGCUGGCGUGCAGCAGCCGCUCGAGGUGGAGCUGUACAAUUGCAUUGUGGUACAGUACGAC ...(((((((..(((.(((....((....))....))).)))..((..((((((......))))))(((((((..((((.....)))).)))))))....))......)))))))..... ( -45.90) >DroGri_CAF1 8850 120 + 1 CAGGCAGUGCAAUUGCGCAUUAACUACAAGGAUUUCGCAGAGGAAUUGGCCAGCAUUGUGGCUGGUGUGCAGCGACCGCUCGAGGUGGUGCUGCACAAUUGCUUUGUGGUGCAGUACGAC ......((((...(((((.....((....))....(((((((.((((.((((((......))))))(((((((.(((((.....)))))))))))))))).))))))))))))))))... ( -51.10) >DroWil_CAF1 9137 120 + 1 CAUGAAGUAAAGUUGCUUAUCAACUACAAGGACUUGGCUCAGGAACUGGCCAGCACUGUGGCUGGCGUACAGAAACCACUCGAAGCAGAGCUUCACAAUUGCAUUGUGGUUCAAUAUGAC (((((((((....)))))...((((((((.....((((.(((...)))))))(((..(((((((.....)))...))))..(((((...))))).....))).))))))))...)))).. ( -32.40) >DroMoj_CAF1 7772 120 + 1 CAAUCGGUGCAGCUGCGCAUCAACUAUAAGGACUUUGCCCAGGAGCUGGCCAGCACAGUGGCUGGUGUGCAGCAACCCCUGGAAGUGGAGCUGCACAAUUGCAUUGUGGUGCAGUACGAC ...(((((((((((.(((.....((....)).......(((((.((((((((((......))))))...))))....)))))..))).)))))))).(((((((....))))))).))). ( -50.90) >DroAna_CAF1 7329 120 + 1 CUGUUGGCUGUUUUGCGCCUCAACUACAAGGACUUUGCCCAGGAGUUGGCCAGCACGGUGGCCGGAGUGCAGAAGCCGCUGGAAGCGGAGCUGCACAACUGCAUUGUGGUGCAGUACGAC ...(((((..(..(((...((((((....((......))....))))))...)))..)..))))).((((((...((((.....))))..)))))).(((((((....)))))))..... ( -49.20) >consensus CAGGCAGUGCAGCUGCGCAUCAACUACAAGGACUUUGCCCAGGAGCUGGCCAGCACAGUGGCUGGCGUGCAGAAACCGCUCGAAGCGGAGCUGCACAAUUGCAUUGUGGUGCAGUACGAC ...........(((((......(((((((...............((((((((......))))))))((((((...((((.....))))..)))))).......))))))))))))..... (-29.63 = -29.83 + 0.20)
| Location | 16,699,151 – 16,699,271 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 81.44 |
| Mean single sequence MFE | -45.20 |
| Consensus MFE | -29.69 |
| Energy contribution | -29.67 |
| Covariance contribution | -0.02 |
| Combinations/Pair | 1.42 |
| Mean z-score | -2.04 |
| Structure conservation index | 0.66 |
| SVM decision value | 0.53 |
| SVM RNA-class probability | 0.770112 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
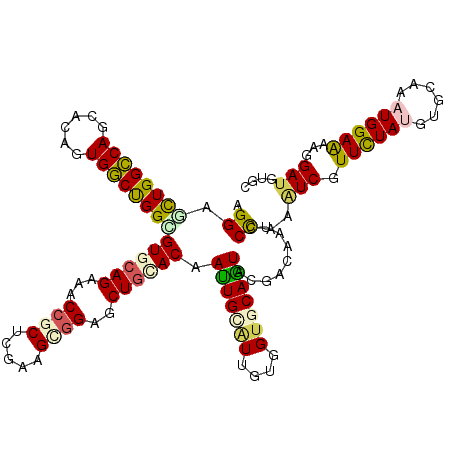
>2R_DroMel_CAF1 16699151 120 + 20766785 AGGAGCUGGUCAGUACGGUGACCGGCGUACAGAAACCGCUCGAAGCGGAGCUGCACAACUGUGUUGUGGUGCAGUUCGAUGGCCCCAUGUCCUUCUACGUCCAGAUGGAGAGCGAUGUGC ....((((((((......))))))))(((((.....(((((((((.((((((((((.((......)).))))))))).(((....)))..)))))....(((....)))))))).))))) ( -49.20) >DroVir_CAF1 7685 120 + 1 AGGAGCUGGCCAGCACAGUGGCUGGCGUGCAGCAGCCGCUCGAGGUGGAGCUGUACAAUUGCAUUGUGGUACAGUACGACAAUGCCAAAUCUUUCUAUGUGCAAAUGGAACAGGAUGUGC ....((..((((......))))..))(((((((..((((.....)))).)))))))....(((((((.(((...))).)))))))((.((((((((((......))))))..)))).)). ( -47.00) >DroGri_CAF1 8890 120 + 1 AGGAAUUGGCCAGCAUUGUGGCUGGUGUGCAGCGACCGCUCGAGGUGGUGCUGCACAAUUGCUUUGUGGUGCAGUACGACAAUCCCAAGUCCUUCUAUGUGCAAAUGGAACAGGAUGUGC .((.((((((((((......))))))(((((((.(((((.....)))))))))))).(((((........)))))....)))).))..((((((((((......)))))..))))).... ( -48.80) >DroWil_CAF1 9177 120 + 1 AGGAACUGGCCAGCACUGUGGCUGGCGUACAGAAACCACUCGAAGCAGAGCUUCACAAUUGCAUUGUGGUUCAAUAUGACGAUCCCACUUCGUUUUAUGUGCAAAUGGAGAAGGAUGUGC ........((((((......))))))(((((....((.((((((((...))))).((.((((((.((((.((........))..))))..........)))))).)))))..)).))))) ( -35.50) >DroMoj_CAF1 7812 120 + 1 AGGAGCUGGCCAGCACAGUGGCUGGUGUGCAGCAACCCCUGGAAGUGGAGCUGCACAAUUGCAUUGUGGUGCAGUACGACAAUGCCAAAUCGUUCUAUGUGCAAAUGGAACAGGAUGUGC ..((..((((((((......))))))(((((((..((.(.....).)).)))))))....(((((((.(((...))).)))))))))..))(((((((......)))))))......... ( -43.10) >DroAna_CAF1 7369 120 + 1 AGGAGUUGGCCAGCACGGUGGCCGGAGUGCAGAAGCCGCUGGAAGCGGAGCUGCACAACUGCAUUGUGGUGCAGUACGACGAUCCUAGAUCGUUCUACGUGCAGAUGGAGAGUGAUGUGG .....(((((((......))))))).((((((...((((.....))))..))))))..((.((((...(..(.(((.(((((((...))))))).))))..).)))).)).......... ( -47.60) >consensus AGGAGCUGGCCAGCACAGUGGCUGGCGUGCAGAAACCGCUCGAAGCGGAGCUGCACAAUUGCAUUGUGGUGCAGUACGACAAUCCCAAAUCGUUCUAUGUGCAAAUGGAAAAGGAUGUGC .((.((((((((......))))))))((((((...((((.....))))..)))))).(((((((....))))))).........))..(((.((((((......))))))...))).... (-29.69 = -29.67 + -0.02)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:37:22 2006