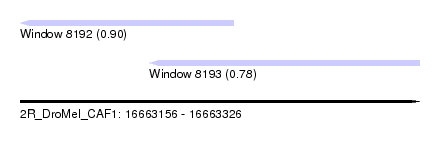
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 16,663,156 – 16,663,326 |
| Length | 170 |
| Max. P | 0.897603 |

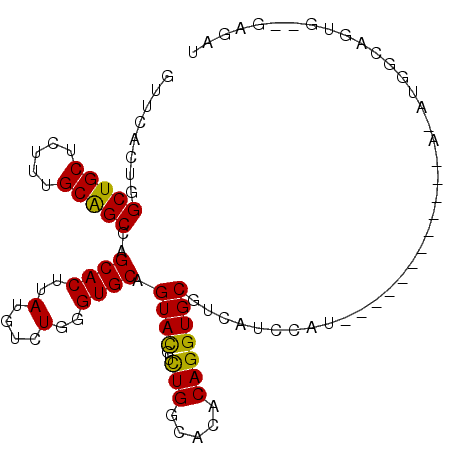
| Location | 16,663,156 – 16,663,247 |
|---|---|
| Length | 91 |
| Sequences | 6 |
| Columns | 104 |
| Reading direction | reverse |
| Mean pairwise identity | 75.39 |
| Mean single sequence MFE | -32.53 |
| Consensus MFE | -18.85 |
| Energy contribution | -18.35 |
| Covariance contribution | -0.50 |
| Combinations/Pair | 1.18 |
| Mean z-score | -1.84 |
| Structure conservation index | 0.58 |
| SVM decision value | 1.00 |
| SVM RNA-class probability | 0.897603 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 16663156 91 - 20766785 GUUCACUGGCUGCUCUUUGCAGCGAGCACUUAUGUGUGGGUGCAGUACGUCUGGCACACAGGUGCGUCAUCCGU------------A-GUGGCAGUGGGGAGAU .((((((((((((.....)))))((((((((.(((((.((.((.....)))).))))).)))))).))......------------.-....)))))))..... ( -35.90) >DroVir_CAF1 9670 84 - 1 GUACAUUGGCUGCUUUUUGCAGCCAGCACAUACGUCUGGGUGCAGUACGUUUGGCAUACAGGUGCGUUA------------------AAUAAUAAUA--UUGUU ((((..(((((((.....)))))))((.((.(((((((....))).)))).))))......))))....------------------..........--..... ( -28.60) >DroPse_CAF1 11379 103 - 1 GUUCACUGGCUGCUCUUUGCGGCCAGCACAUACGUCUGGGUGCAGUACGUCUGGCAUACAGGUGCGUCAUCCAUCUCCGGCUAUAAA-ACAGGAACAGGCAGCA ((((.((((((((.....)))))))).......(((((((((..((((..(((.....)))))))..)))))).....)))......-....))))........ ( -33.50) >DroSec_CAF1 9067 91 - 1 GUUCACUGGCUGCUCUUCGCAGCGAGCACUUAUGUGUGGGUGCAGUACGUCUGGCAUACAGGUGCGUCUUCCGU------------A-GUGGCAGUGGGGAGAU .((((((((((((.....)))))((((((((.(((((.((.((.....)))).))))).)))))).))......------------.-....)))))))..... ( -32.90) >DroEre_CAF1 9147 87 - 1 GUUCACUGGCUGCUCUUUGCAGCAAGCACUUAUGUGUGGGUGCAGUACGUCUGGCACACAGGUGCGUCAUCCA---------------CUGGCAGUG--GAGAU .((((((((((((.....)))))..((((((.(((((.((.((.....)))).))))).))))))........---------------....)))))--))... ( -35.90) >DroAna_CAF1 9011 82 - 1 GUUCACUGGCUGCUCUUCGCGGCCAGCACGUAUGUCUGGGUGCAGUAUGUCUGGCACACAGGUGCGUCAUCUUA------------A-CU-CCA--------AU .....((((((((.....)))))))).((((((..(((.((((((.....)).)))).))))))))).......------------.-..-...--------.. ( -28.40) >consensus GUUCACUGGCUGCUCUUUGCAGCCAGCACUUAUGUCUGGGUGCAGUACGUCUGGCACACAGGUGCGUCAUCCAU____________A_AUGGCAGUG__GAGAU ........(((((.....)))))..((((..(....)..)))).((((..(((.....)))))))....................................... (-18.85 = -18.35 + -0.50)

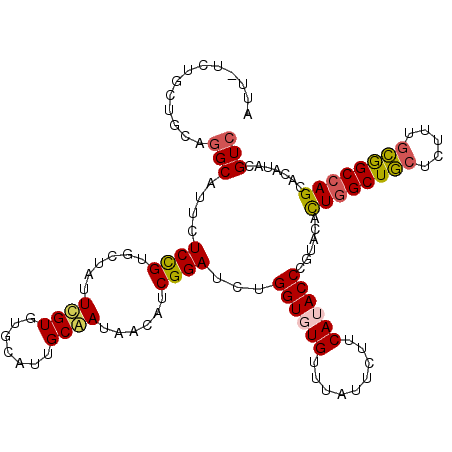
| Location | 16,663,211 – 16,663,326 |
|---|---|
| Length | 115 |
| Sequences | 6 |
| Columns | 116 |
| Reading direction | reverse |
| Mean pairwise identity | 82.89 |
| Mean single sequence MFE | -32.95 |
| Consensus MFE | -24.45 |
| Energy contribution | -24.15 |
| Covariance contribution | -0.30 |
| Combinations/Pair | 1.20 |
| Mean z-score | -2.16 |
| Structure conservation index | 0.74 |
| SVM decision value | 0.55 |
| SVM RNA-class probability | 0.778605 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 16663211 115 - 20766785 AUU-UCAUUUGCAGGCAUUCUCCGUACUGUUCGUGUGCAUAGCGAUAACGUCGGAUCUGGUCUGUUUAUUCUUCAUACCCGUUCACUGGCUGCUCUUUGCAGCGAGCACUUAUGUG ...-..((..((((((....((((.((...((((.......))))....))))))....))))))..))................((.(((((.....))))).))(((....))) ( -29.40) >DroVir_CAF1 9718 116 - 1 UUGACUUGCUGCAGGCAUUUUCUGUGCUUUUUGUGUGCAUUGCAAUAACAUCGGAUCUGGUGUGUUUGUUCUUCAUACCCGUACAUUGGCUGCUUUUUGCAGCCAGCACAUACGUC ..(((..((.(((((.....)))))))....(((((((...((((..((((((....))))))..)))).................(((((((.....)))))))))))))).))) ( -36.50) >DroPse_CAF1 11446 115 - 1 AUU-ACUUGUACAGGCAUUCUCCGUGCUAUUCGUCUGUAUUGCGAUAACAUCGGAUCUGGUCUGCUUAUUUUUCAUACCCGUUCACUGGCUGCUCUUUGCGGCCAGCACAUACGUC ...-.......(((((....((((((.(((.(((.......)))))).)).))))....)))))...............(((...((((((((.....)))))))).....))).. ( -31.10) >DroGri_CAF1 10190 115 - 1 CUU-UGUGCAACAGGCCUUUUCCGUGCUAUUUGUGUGCAUUGCAAUAACAUCGGAUCUGGUGUGUUUGUUCUUCAUACCCGUACAUUGGCUGCUCUUUGCGGCCAGCACAUACGUC ...-(((((((((.(((...((((((.((((.(((.....))))))).)).))))...))).))))....................(((((((.....))))))))))))...... ( -36.80) >DroWil_CAF1 9890 115 - 1 AUC-CUUUUUGCAGGCAUUCUCCGUACUUUUCGUGUGCAUAGCGAUUACAUCGGAUUUGGUGUGUCUAUUUUUCAUACCAGUUCACUGGCUACUCUUUUUGGCCAGCACAUAUGUC ...-.........(((((..((((......((((.......))))......)))).((((((((.........))))))))....(((((((.......))))))).....))))) ( -28.00) >DroMoj_CAF1 9638 115 - 1 UUU-UGUGCUCCAGGCAUUUUCUGUGCUAUUCGUGUGCAUUGCAAUAACAUCGGAUCUGGUGUGCUUGUUCUUCAUACCCGUACAUUGGCUGCUCUUUGCGGCCAGCACGUACGUC ...-.(..(.((((((((.....)))).((((((((...........))).))))))))).)..)..............(((((.((((((((.....))))))))...))))).. ( -35.90) >consensus AUU_UCUGCUGCAGGCAUUCUCCGUGCUAUUCGUGUGCAUUGCAAUAACAUCGGAUCUGGUGUGUUUAUUCUUCAUACCCGUACACUGGCUGCUCUUUGCGGCCAGCACAUACGUC .............(((....((((......((((.......))))......))))...((((((.........))))))......((((((((.....)))))))).......))) (-24.45 = -24.15 + -0.30)

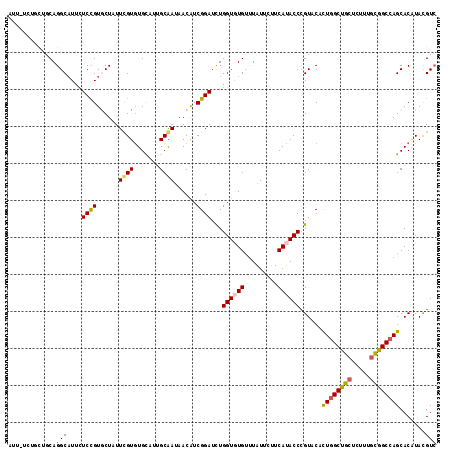
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:37:08 2006