
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 16,652,013 – 16,652,159 |
| Length | 146 |
| Max. P | 0.994272 |

| Location | 16,652,013 – 16,652,120 |
|---|---|
| Length | 107 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 80.37 |
| Mean single sequence MFE | -37.33 |
| Consensus MFE | -28.81 |
| Energy contribution | -28.82 |
| Covariance contribution | 0.00 |
| Combinations/Pair | 1.21 |
| Mean z-score | -2.42 |
| Structure conservation index | 0.77 |
| SVM decision value | 1.30 |
| SVM RNA-class probability | 0.939657 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
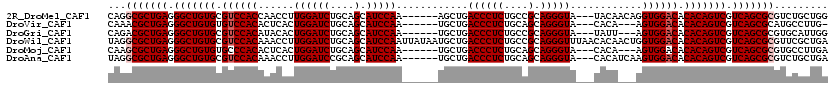
>2R_DroMel_CAF1 16652013 107 + 20766785 --CAACACCAGAUGCAU---CAGCCAAUCCAACGCCAGCGCAGGCGCUGAGGGCUGUGCGUCCACCAACCUUGGAUCUGCAGCAUCCAA------AGCUGACCCUCUGCCGCAGGGUA-- --........((((((.---(((((....((.((((......)))).))..)))))))))))..(((....))).....((((......------.))))((((((....).))))).-- ( -37.40) >DroPse_CAF1 1962 104 + 1 --AAACAUCACCUGCAA---CAGC---UCCAAUGCCAACACAGGCGCUGAGGGCUGUGCGUCCACAAUCCUUGGAUCCGCAGCAUCCAA------UGCUGACCCUCUGCCGCAGGGUA-- --........(((((.(---((((---((((.((((......)))).)).)))))))(((((((.......)))))...((((((...)------))))).......)).)))))...-- ( -38.70) >DroGri_CAF1 930 104 + 1 --CAACAACAGUAGCAACCGC------UCUGGCGUCAAUGCAGACGCUGAGGGCUGUGCGUCCACAUACACUGGAUCUGCAGCAUCCAA------UGCUGACCCUCUGCCGCAGGGUA-- --......(((..(((...((------(((((((((......)))))).)))))..)))(((((.......))))))))((((((...)------)))))((((((....).))))).-- ( -36.80) >DroWil_CAF1 400 112 + 1 --UAACAGCAACGGUAU---CAGCUCACC---AGUCAGCUUAGGCGCUGAGGGCUGUGCGUCCACAAACCUUGGAUCUGCAGCAUCCAAUUAUAAUGCUGACCCUCUGCCGCAGGGUUUA --...(((((..(((..---......)))---..(((((......)))))((((((((.(((((.......))))).))))))..))........)))))((((((....).)))))... ( -34.90) >DroAna_CAF1 585 106 + 1 ACCAACACCA---GCAA---CAGCCAAUCCAACGCCAGCAUAGGCGCUGAGGGCUGUGCGUCCACAAACCUUGGAUCCGCAGCAUCCAA------UGCUGACCCUCUGCAGCAGGGUA-- ........((---((((---(((((....((.((((......)))).))..))))))(((((((.......))))..))).........------)))))((((((....).))))).-- ( -37.50) >DroPer_CAF1 1956 104 + 1 --AAACAUCACCUGCAA---CAGC---UCCAAUGCCAACACAGGCGCUGAGGGCUGUGCGUCCACAAUCCUUGGAUCCGCAGCAUCCAA------UGCUGACCCUCUGCCGCAGGGUA-- --........(((((.(---((((---((((.((((......)))).)).)))))))(((((((.......)))))...((((((...)------))))).......)).)))))...-- ( -38.70) >consensus __CAACACCACCUGCAA___CAGC___UCCAACGCCAACACAGGCGCUGAGGGCUGUGCGUCCACAAACCUUGGAUCCGCAGCAUCCAA______UGCUGACCCUCUGCCGCAGGGUA__ ....................((((.....((.((((......)))).)).((((((((.(((((.......))))).))))))..)).........))))((((((....).)))))... (-28.81 = -28.82 + 0.00)

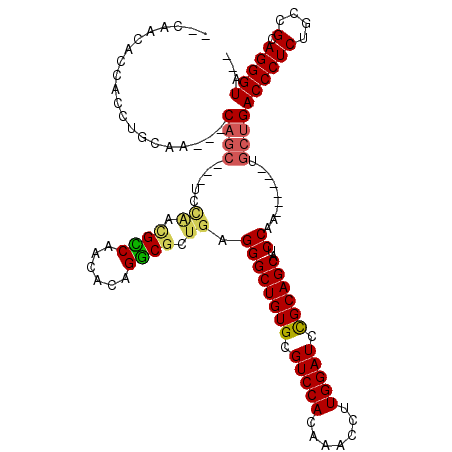
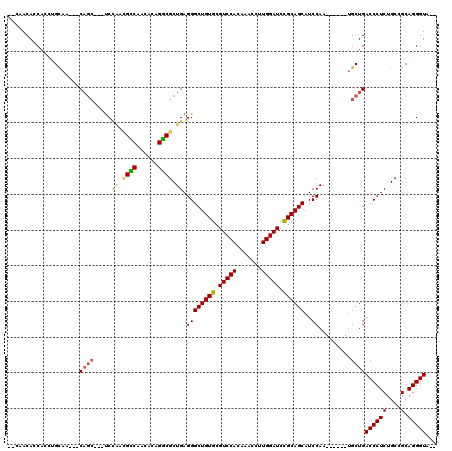
| Location | 16,652,013 – 16,652,120 |
|---|---|
| Length | 107 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 80.37 |
| Mean single sequence MFE | -43.85 |
| Consensus MFE | -24.24 |
| Energy contribution | -24.38 |
| Covariance contribution | 0.14 |
| Combinations/Pair | 1.09 |
| Mean z-score | -2.01 |
| Structure conservation index | 0.55 |
| SVM decision value | 0.19 |
| SVM RNA-class probability | 0.624988 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 16652013 107 - 20766785 --UACCCUGCGGCAGAGGGUCAGCU------UUGGAUGCUGCAGAUCCAAGGUUGGUGGACGCACAGCCCUCAGCGCCUGCGCUGGCGUUGGAUUGGCUG---AUGCAUCUGGUGUUG-- --.(((((.(....))))))(((((------((((((.......)))))))))))..(((.((((((((.((((((((......))))))))...)))))---.))).))).......-- ( -47.40) >DroPse_CAF1 1962 104 - 1 --UACCCUGCGGCAGAGGGUCAGCA------UUGGAUGCUGCGGAUCCAAGGAUUGUGGACGCACAGCCCUCAGCGCCUGUGUUGGCAUUGGA---GCUG---UUGCAGGUGAUGUUU-- --..(((((((((((...(((((((------(.((.(((((.(((((....)))((((....))))..)).))))))).))))))))......---.)))---))))))).)......-- ( -44.50) >DroGri_CAF1 930 104 - 1 --UACCCUGCGGCAGAGGGUCAGCA------UUGGAUGCUGCAGAUCCAGUGUAUGUGGACGCACAGCCCUCAGCGUCUGCAUUGACGCCAGA------GCGGUUGCUACUGUUGUUG-- --......(((((((.(.(((.(((------((((((.......))))))))).....))).).((((((((.(((((......)))))..))------).)))))...)))))))..-- ( -41.10) >DroWil_CAF1 400 112 - 1 UAAACCCUGCGGCAGAGGGUCAGCAUUAUAAUUGGAUGCUGCAGAUCCAAGGUUUGUGGACGCACAGCCCUCAGCGCCUAAGCUGACU---GGUGAGCUG---AUACCGUUGCUGUUA-- ........(((((((.((((((((........((..(((..(((((.....)))))..).))..))(((.(((((......)))))..---)))..))))---)).)).)))))))..-- ( -40.20) >DroAna_CAF1 585 106 - 1 --UACCCUGCUGCAGAGGGUCAGCA------UUGGAUGCUGCGGAUCCAAGGUUUGUGGACGCACAGCCCUCAGCGCCUAUGCUGGCGUUGGAUUGGCUG---UUGC---UGGUGUUGGU --.(((((.......)))))(((((------(..(..((.((.(((((.(((.(((((....))))).))).((((((......))))))))))).)).)---)..)---..)))))).. ( -45.40) >DroPer_CAF1 1956 104 - 1 --UACCCUGCGGCAGAGGGUCAGCA------UUGGAUGCUGCGGAUCCAAGGAUUGUGGACGCACAGCCCUCAGCGCCUGUGUUGGCAUUGGA---GCUG---UUGCAGGUGAUGUUU-- --..(((((((((((...(((((((------(.((.(((((.(((((....)))((((....))))..)).))))))).))))))))......---.)))---))))))).)......-- ( -44.50) >consensus __UACCCUGCGGCAGAGGGUCAGCA______UUGGAUGCUGCAGAUCCAAGGUUUGUGGACGCACAGCCCUCAGCGCCUGUGCUGGCGUUGGA___GCUG___UUGCAGCUGAUGUUG__ ........((.((.((((((...........((((((.......))))))....((((....)))))))))).))(((......)))..................))............. (-24.24 = -24.38 + 0.14)

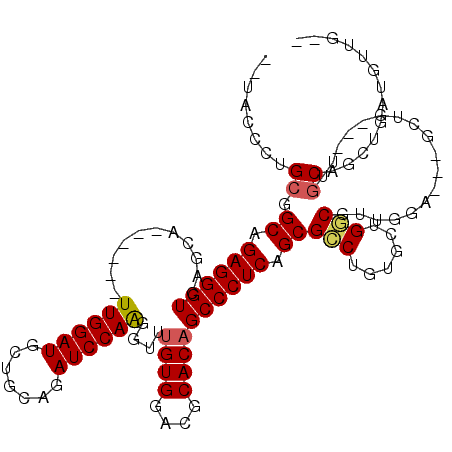
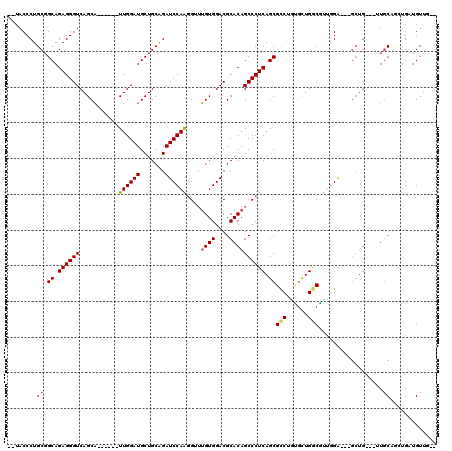
| Location | 16,652,048 – 16,652,159 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 84.71 |
| Mean single sequence MFE | -52.48 |
| Consensus MFE | -47.20 |
| Energy contribution | -47.70 |
| Covariance contribution | 0.50 |
| Combinations/Pair | 1.00 |
| Mean z-score | -4.55 |
| Structure conservation index | 0.90 |
| SVM decision value | 2.46 |
| SVM RNA-class probability | 0.994272 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
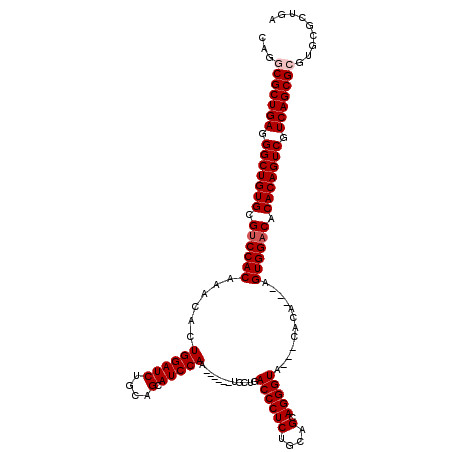
>2R_DroMel_CAF1 16652048 111 + 20766785 CAGGCGCUGAGGGCUGUGCGUCCACCAACCUUGGAUCUGCAGCAUCCAA------AGCUGACCCUCUGCCGCAGGGUA---UACAACAGGUGGACACACAGUCGUCAGCGCGUCUGCUGG ((((((((((.(((((((.(((((((....(((((((....).))))))------.....((((((....).))))).---.......))))))).))))))).)))))))..))).... ( -55.10) >DroVir_CAF1 601 107 + 1 CAAACGCUGAGGGCUGUGUGUCCACACUCACUGGAUCUGCAGCAUCCAA------UGCUGACCCUCUGCAGCAGGGUA---CACA---AGUGGACACACAGUCGUCAGCGCAUGCCUUG- ....((((((.((((((((((((((..............((((((...)------)))))((((((....).))))).---....---.)))))))))))))).)))))).........- ( -52.70) >DroGri_CAF1 962 108 + 1 CAGACGCUGAGGGCUGUGCGUCCACAUACACUGGAUCUGCAGCAUCCAA------UGCUGACCCUCUGCCGCAGGGUA---UAUU---AGUGGACACACAGUCGUCAGCGCGUGCAUUGG ..(.((((((.(((((((.((((((..............((((((...)------)))))((((((....).))))).---....---.)))))).))))))).)))))))......... ( -49.10) >DroWil_CAF1 432 120 + 1 UAGGCGCUGAGGGCUGUGCGUCCACAAACCUUGGAUCUGCAGCAUCCAAUUAUAAUGCUGACCCUCUGCCGCAGGGUUUAACACAACUGGUGGACACACAGUCGUCAGCGCGUUCGCUGA ...(((((((.(((((((.((((((((((((((((....((((((.........))))))....)).....))))))))..((....)))))))).))))))).)))))))......... ( -52.90) >DroMoj_CAF1 615 108 + 1 CAAGCGCUGAGGGCUGUGUGCCCACACUCACUGGAUCUGCAGCAUCCAA------UGCUGACCCUCUGCAGCAGGGUA---CACA---AGUGGACACACAGUCGUCAGCGCGUGCCUUGA ...(((((((.(((((((((.((((..............((((((...)------)))))((((((....).))))).---....---.)))).))))))))).)))))))......... ( -52.10) >DroAna_CAF1 619 111 + 1 UAGGCGCUGAGGGCUGUGCGUCCACAAACCUUGGAUCCGCAGCAUCCAA------UGCUGACCCUCUGCAGCAGGGUA---CACAUCAAGUGGACACACAGUCGUCAGCGCGUCUGCUGA ...(((((((.(((((((.((((((..............((((((...)------)))))((((((....).))))).---........)))))).))))))).)))))))......... ( -53.00) >consensus CAGGCGCUGAGGGCUGUGCGUCCACAAACACUGGAUCUGCAGCAUCCAA______UGCUGACCCUCUGCAGCAGGGUA___CACA___AGUGGACACACAGUCGUCAGCGCGUGCGCUGA ...(((((((.(((((((.((((((......((((((....).)))))............((((((....).)))))............)))))).))))))).)))))))......... (-47.20 = -47.70 + 0.50)
| Location | 16,652,048 – 16,652,159 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 84.71 |
| Mean single sequence MFE | -53.10 |
| Consensus MFE | -47.05 |
| Energy contribution | -46.22 |
| Covariance contribution | -0.83 |
| Combinations/Pair | 1.12 |
| Mean z-score | -4.06 |
| Structure conservation index | 0.89 |
| SVM decision value | 2.35 |
| SVM RNA-class probability | 0.992790 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
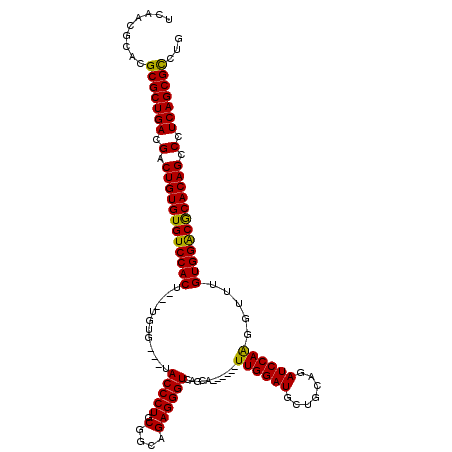
>2R_DroMel_CAF1 16652048 111 - 20766785 CCAGCAGACGCGCUGACGACUGUGUGUCCACCUGUUGUA---UACCCUGCGGCAGAGGGUCAGCU------UUGGAUGCUGCAGAUCCAAGGUUGGUGGACGCACAGCCCUCAGCGCCUG ....(((..(((((((.(.((((((((((((((((((((---.....)))))))).....(((((------((((((.......))))))))))))))))))))))).).)))))))))) ( -58.80) >DroVir_CAF1 601 107 - 1 -CAAGGCAUGCGCUGACGACUGUGUGUCCACU---UGUG---UACCCUGCUGCAGAGGGUCAGCA------UUGGAUGCUGCAGAUCCAGUGAGUGUGGACACACAGCCCUCAGCGUUUG -.....((.(((((((.(.((((((((((((.---..((---.(((((.......))))))).((------((((((.......))))))))...)))))))))))).).))))))).)) ( -51.60) >DroGri_CAF1 962 108 - 1 CCAAUGCACGCGCUGACGACUGUGUGUCCACU---AAUA---UACCCUGCGGCAGAGGGUCAGCA------UUGGAUGCUGCAGAUCCAGUGUAUGUGGACGCACAGCCCUCAGCGUCUG ......((.(((((((.(.((((((((((((.---....---.(((((.(....))))))..(((------((((((.......)))))))))..)))))))))))).).))))))).)) ( -51.90) >DroWil_CAF1 432 120 - 1 UCAGCGAACGCGCUGACGACUGUGUGUCCACCAGUUGUGUUAAACCCUGCGGCAGAGGGUCAGCAUUAUAAUUGGAUGCUGCAGAUCCAAGGUUUGUGGACGCACAGCCCUCAGCGCCUA .........(((((((.(.((((((((((.(((((((((....(((((.(....))))))......))))))))).....((((((.....)))))))))))))))).).)))))))... ( -52.40) >DroMoj_CAF1 615 108 - 1 UCAAGGCACGCGCUGACGACUGUGUGUCCACU---UGUG---UACCCUGCUGCAGAGGGUCAGCA------UUGGAUGCUGCAGAUCCAGUGAGUGUGGGCACACAGCCCUCAGCGCUUG .........(((((((.(.((((((((((((.---..((---.(((((.......))))))).((------((((((.......))))))))...)))))))))))).).)))))))... ( -52.80) >DroAna_CAF1 619 111 - 1 UCAGCAGACGCGCUGACGACUGUGUGUCCACUUGAUGUG---UACCCUGCUGCAGAGGGUCAGCA------UUGGAUGCUGCGGAUCCAAGGUUUGUGGACGCACAGCCCUCAGCGCCUA .........(((((((.(.((((((((((((..(((((.---.(((((.......)))))..)))------))((((.(...).)))).......)))))))))))).).)))))))... ( -51.10) >consensus UCAACGCACGCGCUGACGACUGUGUGUCCACU___UGUG___UACCCUGCGGCAGAGGGUCAGCA______UUGGAUGCUGCAGAUCCAAGGUUUGUGGACGCACAGCCCUCAGCGCCUG .........(((((((.(.((((((((((((............(((((.(....))))))...........((((((.......)))))).....)))))))))))).).)))))))... (-47.05 = -46.22 + -0.83)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:36:59 2006