
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
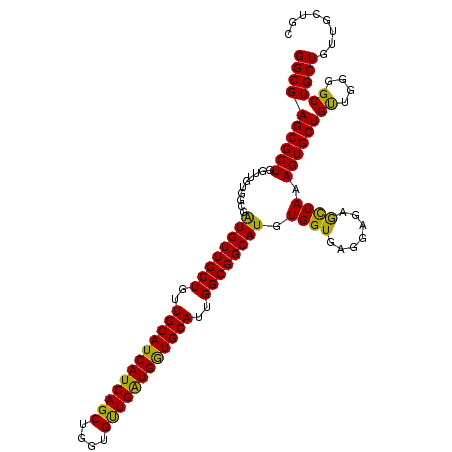
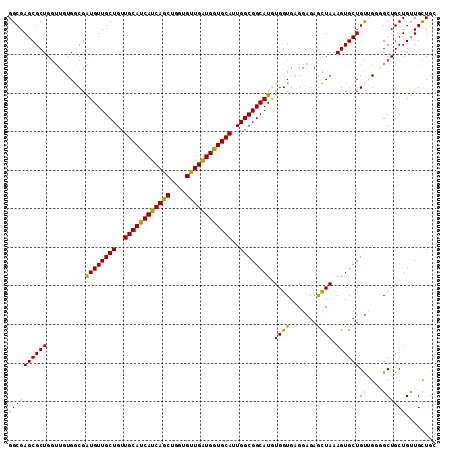
| Location | 16,512,614 – 16,512,762 |
| Length | 148 |
| Max. P | 0.998779 |

| Location | 16,512,614 – 16,512,725 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 111 |
| Reading direction | reverse |
| Mean pairwise identity | 89.91 |
| Mean single sequence MFE | -48.02 |
| Consensus MFE | -42.17 |
| Energy contribution | -40.70 |
| Covariance contribution | -1.47 |
| Combinations/Pair | 1.19 |
| Mean z-score | -3.83 |
| Structure conservation index | 0.88 |
| SVM decision value | 3.22 |
| SVM RNA-class probability | 0.998779 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 16512614 111 - 20766785 GGCGAGCGCUGGUUGUGGCGAUGUUGCUGUUGCAUCAUCAGCUGGUGUUGAUGGUGCAUUGGCGGCAUGUGGUGCGGAGAGCUAAAGUGCUGUUGGGGCUGCUGUUGUUGC (((....)))....(..(((((((((((..((((((((((((....))))))))))))..))))))..(..((.(.((.(((......))).)).).))..).)))))..) ( -49.70) >DroPse_CAF1 15617 111 - 1 GGCGAGCGCUGGCUAUGACGGUGUUGCUGCUGCAUCAUCAGCUGGUGCUGGUGGUGCAUUGGCGGCAUAUGGUGAGGGGAAUUAAAGUGCUGCGGUUGCUGCUGCUGCUGG ((((((((((..((.(.((.((((.(((((((((((((((((....)))))))))))...)))))))))).)).)..))......))))))(((((....))))))))).. ( -50.00) >DroSim_CAF1 9908 111 - 1 GGCGAGCGCUGGUUGUGGCGAUGUUGCUGUUGCAUCAUCAGCUGGUGUUGAUGGUGCAUUGGCGGCAUGUGGUGCGGAGAGCUAAAGUGCUGUUGUGGCUGCUGUUGUUGA (((.((((((..(((((.((((((((((..((((((((((((....))))))))))))..)))))))).)).)))))..)))......((....)).))))))........ ( -44.90) >DroEre_CAF1 9778 111 - 1 GGCGAGCGCUGAUUGUGGCGAUGUUGCUGUUGCAUCAUCAGCUGGUGUUGAUGAUGCAUUGGCGGCAUGUGGUGAGGGGAGCUAAAGUGCUGUUGGGGCUGCUGUUGUUGC (((.(((.(..((..(.((.((((((((..((((((((((((....))))))))))))..)))))))))).)..)....(((......))).)..).))))))........ ( -46.90) >DroYak_CAF1 9641 111 - 1 GGCGAGCGCUGGUUGUGGCGAUGUUGCUGUUGCAUCAUCAGCUGGUGUUGAUGGUGCAUUGGCGGCAUGUGGUGAGGAGAGCUAAAGUGCUGUUGGGGCUGCUGUUGCUGC (((.(((.((..(..(.((.((((((((..((((((((((((....))))))))))))..)))))))))).)..).((.(((......))).)))).))))))........ ( -46.60) >DroPer_CAF1 15555 111 - 1 GGCGAGCGCUGGCUAUGACGGUGUUGCUGCUGCAUCAUCAGCUGGUGCUGGUGGUGCAUUGGCGGCAUAUGGUGAGGGGAAUUAAAGUGCUGCGGUUGCUGCUGCUGCUGG ((((((((((..((.(.((.((((.(((((((((((((((((....)))))))))))...)))))))))).)).)..))......))))))(((((....))))))))).. ( -50.00) >consensus GGCGAGCGCUGGUUGUGGCGAUGUUGCUGUUGCAUCAUCAGCUGGUGUUGAUGGUGCAUUGGCGGCAUGUGGUGAGGAGAGCUAAAGUGCUGUUGGGGCUGCUGUUGCUGC ((((((((((..........((((((((..((((((((((((....))))))))))))..)))))))).((((.......)))).))))))((....))))))........ (-42.17 = -40.70 + -1.47)

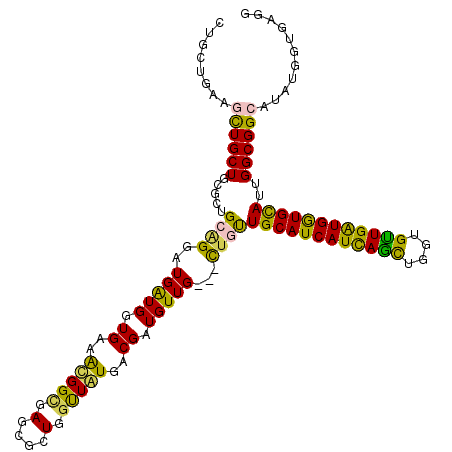
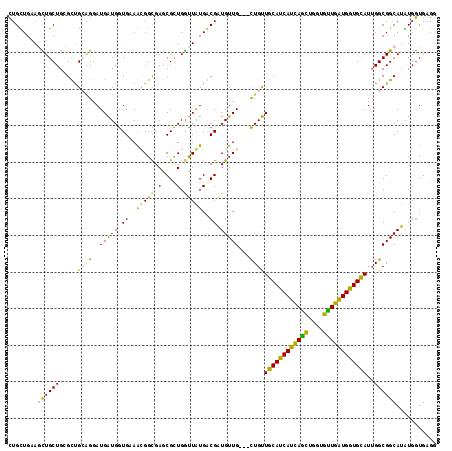
| Location | 16,512,648 – 16,512,762 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 83.54 |
| Mean single sequence MFE | -45.97 |
| Consensus MFE | -31.57 |
| Energy contribution | -29.60 |
| Covariance contribution | -1.97 |
| Combinations/Pair | 1.46 |
| Mean z-score | -3.05 |
| Structure conservation index | 0.69 |
| SVM decision value | 0.68 |
| SVM RNA-class probability | 0.821492 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 16512648 114 - 20766785 CUGCUGAAGCUGCUGCGCCGCAGGAUGAUGGUGAAGCGGCGAGCGCUGGUUGUGGCGAUGUUG---CUGUUGCAUCAUCAGCUGGUGUUGAUGGUGCAUUGGCGGCAUGUGGUGCGG ((((....((((((.((((((.....).))))).))))))...((((......))))((((((---((..((((((((((((....))))))))))))..)))))))).....)))) ( -53.80) >DroPse_CAF1 15651 114 - 1 CUGUUGCAGCUGCUGCGUUGAGGGAUGGUGGUGCAAUGGCGAGCGCUGGCUAUGACGGUGUUG---CUGCUGCAUCAUCAGCUGGUGCUGGUGGUGCAUUGGCGGCAUAUGGUGAGG .....((.(((..((((..(........)..))))..)))..)).((.(((((....((((((---((..((((((((((((....))))))))))))..))))))))))))).)). ( -48.40) >DroEre_CAF1 9812 114 - 1 CUGUUGAAUUUGCUGGGCUGCAGGAUGAUGGUGAAGCGGCGAGCGCUGAUUGUGGCGAUGUUG---CUGUUGCAUCAUCAGCUGGUGUUGAUGAUGCAUUGGCGGCAUGUGGUGAGG ........((..((..((..(((.............((((....)))).)))..)).((((((---((..((((((((((((....))))))))))))..))))))))..))..)). ( -44.24) >DroWil_CAF1 23446 117 - 1 UUGCUGCAAUUGCUGGGCUGCCGCAUGAUGAUGCAAAGGUGAACGUUGGUUAUGACGAUGUUGUUGUUGUUGCAUCAUUAAUUGAUGUUGAUGAUGUAUAGGCGGCAUAUGAUGAGG ..(((((....))..)))((((((........((((..(..(.(((((.......))))))..)..))))((((((((((((....))))))))))))...)))))).......... ( -35.00) >DroYak_CAF1 9675 114 - 1 CUGCUGAAGUUGCUGGGCUGCAGGAUGAUGGUGAAGCGGCGAGCGCUGGUUGUGGCGAUGUUG---CUGUUGCAUCAUCAGCUGGUGUUGAUGGUGCAUUGGCGGCAUGUGGUGAGG ..(((...((((((..(((((.....).))))..)))))).))).....(..(.((.((((((---((..((((((((((((....))))))))))))..)))))))))).)..).. ( -46.00) >DroPer_CAF1 15589 114 - 1 CUGUUGCAGCUGCUGCGUUGAGGGAUGGUGGUGCAAUGGCGAGCGCUGGCUAUGACGGUGUUG---CUGCUGCAUCAUCAGCUGGUGCUGGUGGUGCAUUGGCGGCAUAUGGUGAGG .....((.(((..((((..(........)..))))..)))..)).((.(((((....((((((---((..((((((((((((....))))))))))))..))))))))))))).)). ( -48.40) >consensus CUGCUGAAGCUGCUGCGCUGCAGGAUGAUGGUGAAACGGCGAGCGCUGGUUAUGACGAUGUUG___CUGUUGCAUCAUCAGCUGGUGUUGAUGGUGCAUUGGCGGCAUAUGGUGAGG ........((((((.....((((..(((((.((..(((((.(....).)))))..)).)))))...))))((((((((((((....))))))))))))..))))))........... (-31.57 = -29.60 + -1.97)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:36:06 2006