
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 16,503,225 – 16,503,385 |
| Length | 160 |
| Max. P | 0.874206 |

| Location | 16,503,225 – 16,503,345 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 86.22 |
| Mean single sequence MFE | -44.56 |
| Consensus MFE | -36.56 |
| Energy contribution | -35.23 |
| Covariance contribution | -1.33 |
| Combinations/Pair | 1.43 |
| Mean z-score | -2.52 |
| Structure conservation index | 0.82 |
| SVM decision value | 0.88 |
| SVM RNA-class probability | 0.874206 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
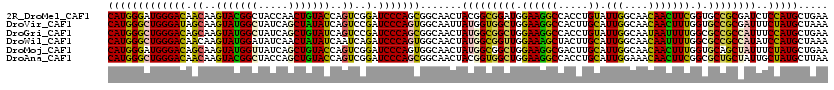
>2R_DroMel_CAF1 16503225 120 + 20766785 CAUGGGAUGGGACAACAAGUACGGCUACCAACUGUACCAGUCGGAUCCCAGCGGCAACUACGGCGGAUGGAAGGCCACCUGUAUUGGCAACAACUUCGGUGCCGCGAUCUCCAUGCUGAA (((((((((((((.((..((((((.......))))))..))..).)))).((((((....((((((.(((....))).)))).(((....)))...)).)))))).))).)))))..... ( -43.20) >DroVir_CAF1 3146 120 + 1 CAUGGGCUGGGAUAGCAAGUAUGGCUAUCAGCUAUAUCAGUCCGAUCCCAGUGGCAAUUAUGGUGGCUGGAAGGCCACUUGCAUUGGCAACAACUUUGGUGCCGCGAUUUCUAUGCUAAA (((((((((((((.((..(((((((.....)))))))..))...))))))))((((.....(((((((....)))))))....(((....)))......)))).......)))))..... ( -44.40) >DroGri_CAF1 2786 120 + 1 CAUGGGCUGGGACAGCAAGUAUGGCUAUCAGCUGUAUCAGUCCGAUCCCAGCGGCAACUAUGGCGGCUGGAAGGCCACCUGUAUUGGCAAUAAUUUUGGCGCCGCCAUUUCCAUGCUGAA ..(((((((..(((((..(((....)))..)))))..)))))))....(((((......(((((((((((....)))........(.(((.....))).))))))))).....))))).. ( -46.10) >DroWil_CAF1 199 120 + 1 CAUGGGCUGGGACAACAAGUAUGGAUAUCAACUAUAUCAAUCAGAUCCCAGUGGCAACUAUGGCGGUUGGAAAGCUACUUGCAUUGGCAACAAUUUUGGCGCCGCCAUAUCCAUGCUAAA ((((((((((((......((((((.......))))))........)))))))(....)((((((((((.((((((.....)).(((....))))))).).))))))))).)))))..... ( -39.24) >DroMoj_CAF1 4199 120 + 1 CAUGGGAUGGGACAGCAAGUAUGGUUAUCAGCUGUACCAGUCGGAUCCCAGUGGCAACUAUGGCGGCUGGAAGGCGACUUGCAUUGGCAACAACUUUGGUGCAGCUAUUUCUAUGCUGAA .................(((((((..((.((((((((((((((..(.(((((.((.......)).))))).)..)))).....(((....)))...))))))))))))..)))))))... ( -44.60) >DroAna_CAF1 202 120 + 1 CAUGGGCUGGGACAACAAGUACGGCUACCAGCUGUACCAGUCGGAUCCCAGCGGCAACUACGGUGGCUGGAAGGCCACCUGCAUUGGAAACAACUUCGGCGCUGCUAUUGCUAUGCUUAA (((((((((((((.((..(((((((.....)))))))..))..).)))))))((((.....(((((((....))))))).((.(((....)))(....).))))))....)))))..... ( -49.80) >consensus CAUGGGCUGGGACAACAAGUAUGGCUAUCAGCUGUACCAGUCGGAUCCCAGCGGCAACUAUGGCGGCUGGAAGGCCACCUGCAUUGGCAACAACUUUGGCGCCGCCAUUUCCAUGCUGAA (((((((((((((.((..(((((((.....)))))))..))..).))))))).......(((((((((.((((((.....)).(((....))))))).).))))))))..)))))..... (-36.56 = -35.23 + -1.33)
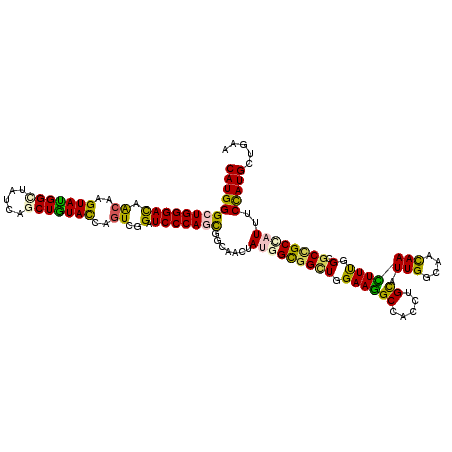
| Location | 16,503,265 – 16,503,385 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 82.09 |
| Mean single sequence MFE | -38.39 |
| Consensus MFE | -28.82 |
| Energy contribution | -28.52 |
| Covariance contribution | -0.30 |
| Combinations/Pair | 1.41 |
| Mean z-score | -1.25 |
| Structure conservation index | 0.75 |
| SVM decision value | -0.05 |
| SVM RNA-class probability | 0.506231 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
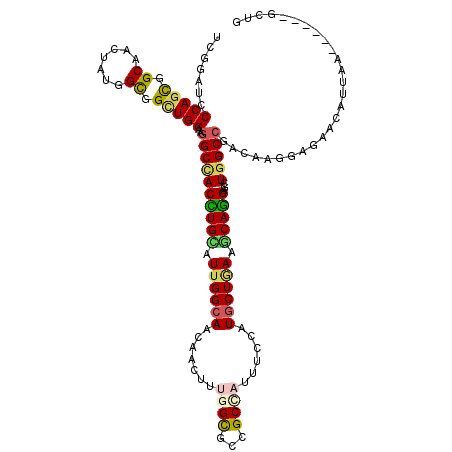
>2R_DroMel_CAF1 16503265 120 + 20766785 UCGGAUCCCAGCGGCAACUACGGCGGAUGGAAGGCCACCUGUAUUGGCAACAACUUCGGUGCCGCGAUCUCCAUGCUGAAGCAGGAGCUGGCCGAUAAGGAGAACGUGAAGCUGACGCUG ........(((((.........((((.(((....))).)))).(((....)))..(((((..((((.(((((.(((....)))((......)).....))))).))))..)))))))))) ( -40.10) >DroVir_CAF1 3186 114 + 1 UCCGAUCCCAGUGGCAAUUAUGGUGGCUGGAAGGCCACUUGCAUUGGCAACAACUUUGGUGCCGCGAUUUCUAUGCUAAAGCAGGAGCUGGCCGACAAGGACAACAUUAA------GUUG (((....(((((.((.......)).)))))..((((((((((.((((((......(((......)))......)))))).)))))...))))).....)))((((.....------)))) ( -36.60) >DroGri_CAF1 2826 114 + 1 UCCGAUCCCAGCGGCAACUAUGGCGGCUGGAAGGCCACCUGUAUUGGCAAUAAUUUUGGCGCCGCCAUUUCCAUGCUGAAACAGGAGUUGGCCGAUAAGGAUAACAUUAA------GUUG (((.((((((((.((.......)).)))))..((((((((((.(..(((.......((((...))))......)))..).)))))...))))))))..))).........------.... ( -42.32) >DroWil_CAF1 239 114 + 1 UCAGAUCCCAGUGGCAACUAUGGCGGUUGGAAAGCUACUUGCAUUGGCAACAAUUUUGGCGCCGCCAUAUCCAUGCUAAAGCAAGAGUUGGCCGACAAGGAGAACAUUAA------GCUA ((.(((((((..(....)..))).)))).)).((((.(((((.((((((.......((((...))))......)))))).))))).(((..((.....))..)))....)------))). ( -34.62) >DroMoj_CAF1 4239 114 + 1 UCGGAUCCCAGUGGCAACUAUGGCGGCUGGAAGGCGACUUGCAUUGGCAACAACUUUGGUGCAGCUAUUUCUAUGCUGAAGCAAGAGUUAGCCGAUAAGGAGAGCAUAAA------AUUG (((..(.(((((.((.......)).))))).)..)))(((((.(((....)))........((((.........))))..))))).(((..((.....))..))).....------.... ( -32.80) >DroAna_CAF1 242 120 + 1 UCGGAUCCCAGCGGCAACUACGGUGGCUGGAAGGCCACCUGCAUUGGAAACAACUUCGGCGCUGCUAUUGCUAUGCUUAAACAGGAGCUGGCCGACAAGGAGAACGUACAGCUGAAGCUG .((..(((.((((((......(((((((....))))))).((.(((....))).....))))))))...((((.((((......))))))))......)))...))..((((....)))) ( -43.90) >consensus UCGGAUCCCAGCGGCAACUAUGGCGGCUGGAAGGCCACCUGCAUUGGCAACAACUUUGGCGCCGCCAUUUCCAUGCUGAAGCAGGAGCUGGCCGACAAGGAGAACAUUAA______GCUG .......(((((.((.......)).)))))..((((((((((.((((((.......((((...))))......)))))).)))))...)))))........................... (-28.82 = -28.52 + -0.30)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:35:58 2006