
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 16,452,532 – 16,452,691 |
| Length | 159 |
| Max. P | 0.974929 |

| Location | 16,452,532 – 16,452,651 |
|---|---|
| Length | 119 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | forward |
| Mean pairwise identity | 95.63 |
| Mean single sequence MFE | -35.43 |
| Consensus MFE | -33.94 |
| Energy contribution | -34.08 |
| Covariance contribution | 0.14 |
| Combinations/Pair | 1.08 |
| Mean z-score | -0.89 |
| Structure conservation index | 0.96 |
| SVM decision value | 0.41 |
| SVM RNA-class probability | 0.725363 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
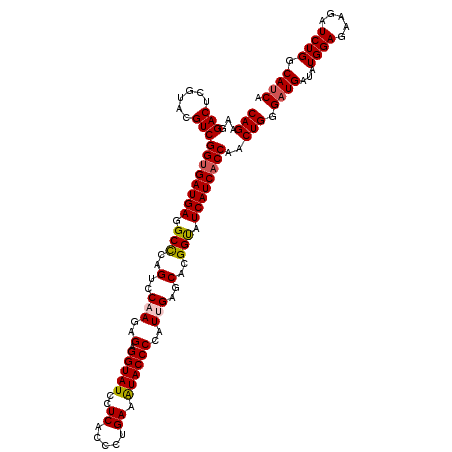
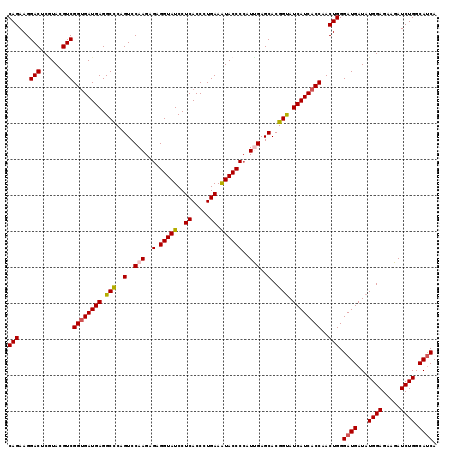
>2R_DroMel_CAF1 16452532 119 + 20766785 CAGAAGGACUCGUACGUCGGUGAUGAGGCCCAGUCCAAGCGUGGUAUCCUCACCCUGAAAUACCCCAUCGAGCACGGUAUCAUCACCAACUGGGAUGAUAUGGAGAAGAUCUGGCACCA ((((.((....(((..((((.(.(((((.(((((....)).)))...))))).)))))..))).)).((...(.(.((((((((.((....)))))))))).).)..))))))...... ( -34.40) >DroVir_CAF1 191505 119 + 1 CAGAAGGACUCGUAUGUCGGUGAUGAGGCUCAGUCCAAGAGAGGUAUCCUCACCCUGAAAUACCCAAUUGAGCACGGUAUCAUCACCAACUGGGAUGAUAUGGAGAAGAUCUGGCAUCA (((...(((......)))((((((((.(((..(..(((..(.(((((..((.....)).))))))..)))..)..))).))))))))..))).((((...((((.....)))).)))). ( -36.10) >DroGri_CAF1 150680 119 + 1 CAGAAGGACUCAUAUGUCGGUGAUGAGGCUCAGUCCAAGAGAGGUAUCCUCACCCUGAAGUACCCCAUUGAGCACGGUAUCAUCACCAACUGGGAUGAUAUGGAGAAGAUCUGGCAUCA (((...(((......)))((((((((.(((..(..(((..(.(((((..((.....)).))))))..)))..)..))).))))))))..))).((((...((((.....)))).)))). ( -36.50) >DroWil_CAF1 284517 119 + 1 CAGAAGGACUCCUAUGUCGGUGAUGAGGCCCAGUCCAAGCGUGGUAUCCUCACCCUGAAAUACCCCAUUGAGCACGGUAUCAUCACCAACUGGGAUGAUAUGGAGAAGAUCUGGCAUCA (((...(((......)))((((((((.(((..(..(((..(.(((((..((.....)).))))).).)))..)..))).))))))))..))).((((...((((.....)))).)))). ( -37.20) >DroMoj_CAF1 211879 119 + 1 CAGAAGGACUCGUACGUCGGUGAUGAGGCUCAGUCCAAGAGAGGUAUCCUCACCCUGAAAUACCCCAUUGAGCACGGUAUCAUCACCAACUGGGAUGAUAUGGAGAAGAUCUGGCAUCA (((...(((......)))((((((((.(((..(..(((..(.(((((..((.....)).))))))..)))..)..))).))))))))..))).((((...((((.....)))).)))). ( -35.60) >DroAna_CAF1 124542 119 + 1 CAGAAGGACUCGUACGUCGGCGAUGAGGCCCAGUCCAAGCGUGGUAUCCUCACCCUGAAAUACCCCAUCGAGCACGGCAUCAUCACCAACUGGGAUGAUAUGGAGAAGAUCUGGCAUCA ((((.((.(((((.((....)))))))..))..((((.(((.(((((..((.....)).))))).)..((....))))((((((.((....)))))))).)))).....))))...... ( -32.80) >consensus CAGAAGGACUCGUACGUCGGUGAUGAGGCCCAGUCCAAGAGAGGUAUCCUCACCCUGAAAUACCCCAUUGAGCACGGUAUCAUCACCAACUGGGAUGAUAUGGAGAAGAUCUGGCAUCA (((...(((......)))((((((((.(((..(..(((..(.(((((..((.....)).))))))..)))..)..))).))))))))..))).((((...((((.....)))).)))). (-33.94 = -34.08 + 0.14)

| Location | 16,452,532 – 16,452,651 |
|---|---|
| Length | 119 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | reverse |
| Mean pairwise identity | 95.63 |
| Mean single sequence MFE | -38.99 |
| Consensus MFE | -35.70 |
| Energy contribution | -35.28 |
| Covariance contribution | -0.42 |
| Combinations/Pair | 1.08 |
| Mean z-score | -1.34 |
| Structure conservation index | 0.92 |
| SVM decision value | 0.95 |
| SVM RNA-class probability | 0.887237 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
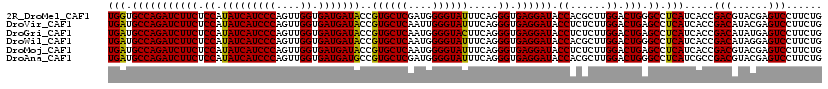
>2R_DroMel_CAF1 16452532 119 - 20766785 UGGUGCCAGAUCUUCUCCAUAUCAUCCCAGUUGGUGAUGAUACCGUGCUCGAUGGGGUAUUUCAGGGUGAGGAUACCACGCUUGGACUGGGCCUCAUCACCGACGUACGAGUCCUUCUG ......((((....(((..(((((((((....)).)))))))..((((((((((((((..(((((((((.(....)))).))))))....))))))))...)).)))))))....)))) ( -40.60) >DroVir_CAF1 191505 119 - 1 UGAUGCCAGAUCUUCUCCAUAUCAUCCCAGUUGGUGAUGAUACCGUGCUCAAUUGGGUAUUUCAGGGUGAGGAUACCUCUCUUGGACUGAGCCUCAUCACCGACAUACGAGUCCUUCUG (((.(((((((((((.((.(((((((((....)).)))))))..((((((....)))))).....)).)))))).((......)).))).)).))).....(((......)))...... ( -35.10) >DroGri_CAF1 150680 119 - 1 UGAUGCCAGAUCUUCUCCAUAUCAUCCCAGUUGGUGAUGAUACCGUGCUCAAUGGGGUACUUCAGGGUGAGGAUACCUCUCUUGGACUGAGCCUCAUCACCGACAUAUGAGUCCUUCUG ......((((....(((.((((.......(((((((((((....((((((....))))))(((((((.((((...))))))))))).......))))))))))))))))))....)))) ( -38.81) >DroWil_CAF1 284517 119 - 1 UGAUGCCAGAUCUUCUCCAUAUCAUCCCAGUUGGUGAUGAUACCGUGCUCAAUGGGGUAUUUCAGGGUGAGGAUACCACGCUUGGACUGGGCCUCAUCACCGACAUAGGAGUCCUUCUG ......((((....((((.(((.......(((((((((((..((((((((....))))))(((((((((.(....)))).))))))...))..))))))))))))))))))....)))) ( -40.11) >DroMoj_CAF1 211879 119 - 1 UGAUGCCAGAUCUUCUCCAUAUCAUCCCAGUUGGUGAUGAUACCGUGCUCAAUGGGGUAUUUCAGGGUGAGGAUACCUCUCUUGGACUGAGCCUCAUCACCGACGUACGAGUCCUUCUG ......((((....(((..(((((((((....)).)))))))..((((((.(((((((..(((((((.((((...)))))))))))....)))))))....)).)))))))....)))) ( -38.10) >DroAna_CAF1 124542 119 - 1 UGAUGCCAGAUCUUCUCCAUAUCAUCCCAGUUGGUGAUGAUGCCGUGCUCGAUGGGGUAUUUCAGGGUGAGGAUACCACGCUUGGACUGGGCCUCAUCGCCGACGUACGAGUCCUUCUG ......((((....(((..(((((((((....)).)))))))..((((.(((((((((..(((((((((.(....)))).))))))....))))))))).....)))))))....)))) ( -41.20) >consensus UGAUGCCAGAUCUUCUCCAUAUCAUCCCAGUUGGUGAUGAUACCGUGCUCAAUGGGGUAUUUCAGGGUGAGGAUACCACGCUUGGACUGAGCCUCAUCACCGACAUACGAGUCCUUCUG (((.(((((((((((.((.(((((((((....)).)))))))..((((((....)))))).....)).)))))).((......)).))).)).))).....(((......)))...... (-35.70 = -35.28 + -0.42)

| Location | 16,452,572 – 16,452,691 |
|---|---|
| Length | 119 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | reverse |
| Mean pairwise identity | 95.85 |
| Mean single sequence MFE | -43.63 |
| Consensus MFE | -41.04 |
| Energy contribution | -40.85 |
| Covariance contribution | -0.19 |
| Combinations/Pair | 1.08 |
| Mean z-score | -1.45 |
| Structure conservation index | 0.94 |
| SVM decision value | 1.74 |
| SVM RNA-class probability | 0.974929 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
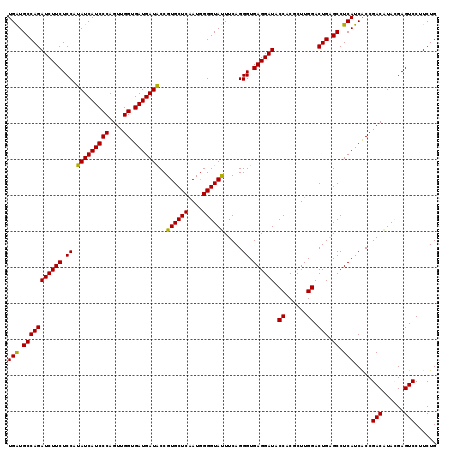
>2R_DroMel_CAF1 16452572 119 - 20766785 GUGCUCCUCGGGGGCAACACGCAGCUCGUUGUAGAAGGUGUGGUGCCAGAUCUUCUCCAUAUCAUCCCAGUUGGUGAUGAUACCGUGCUCGAUGGGGUAUUUCAGGGUGAGGAUACCAC ((((((((((((.((..............((.(((((((.((....)).))))))).))(((((((((....)).)))))))..)).))))).))))))).....((((....)))).. ( -42.60) >DroVir_CAF1 191545 119 - 1 GUGCUCCUCGGGGGCAACACGCAGCUCAUUGUAGAAGGUGUGAUGCCAGAUCUUCUCCAUAUCAUCCCAGUUGGUGAUGAUACCGUGCUCAAUUGGGUAUUUCAGGGUGAGGAUACCUC .........(((((((.(((((..((......))...))))).))))..((((((.((.(((((((((....)).)))))))..((((((....)))))).....)).)))))).))). ( -40.90) >DroPse_CAF1 160782 119 - 1 GUGCUCCUCUGGGGCCACACGCAGCUCGUUGUAGAAGGUGUGGUGCCAGAUCUUCUCCAUAUCAUCCCAGUUGGUGAUGAUACCGUGCUCAAUGGGGUACUUCAGGGUGAGGAUACCUC .......(((((.(((((((((((....)))).....))))))).)))))(((((.((.(((((((((....)).)))))))..((((((....)))))).....)).)))))...... ( -47.00) >DroGri_CAF1 150720 119 - 1 GUGCUCCUCUGGGGCAACACGCAGCUCAUUGUAGAAGGUGUGAUGCCAGAUCUUCUCCAUAUCAUCCCAGUUGGUGAUGAUACCGUGCUCAAUGGGGUACUUCAGGGUGAGGAUACCUC ..........((((((.(((((..((......))...))))).))))..((((((.((.(((((((((....)).)))))))..((((((....)))))).....)).)))))).)).. ( -42.10) >DroAna_CAF1 124582 119 - 1 GUGCUCCUCGGGGGCAACGCGCAGCUCGUUGUAGAAGGUGUGAUGCCAGAUCUUCUCCAUAUCAUCCCAGUUGGUGAUGAUGCCGUGCUCGAUGGGGUAUUUCAGGGUGAGGAUACCAC (((((((((((.(....)((((.((....((.(((((((.((....)).))))))).)).((((((((....)).)))))))).)))))))).))))))).....((((....)))).. ( -42.20) >DroPer_CAF1 161628 119 - 1 GUGCUCCUCUGGGGCCACACGCAGCUCGUUGUAGAAGGUGUGGUGCCAGAUCUUCUCCAUAUCAUCCCAGUUGGUGAUGAUACCGUGCUCAAUGGGGUACUUCAGGGUGAGGAUACCUC .......(((((.(((((((((((....)))).....))))))).)))))(((((.((.(((((((((....)).)))))))..((((((....)))))).....)).)))))...... ( -47.00) >consensus GUGCUCCUCGGGGGCAACACGCAGCUCGUUGUAGAAGGUGUGAUGCCAGAUCUUCUCCAUAUCAUCCCAGUUGGUGAUGAUACCGUGCUCAAUGGGGUACUUCAGGGUGAGGAUACCUC ..........((((((.(((((...((......))..))))).))))..((((((.((.(((((((((....)).)))))))..((((((....)))))).....)).)))))).)).. (-41.04 = -40.85 + -0.19)

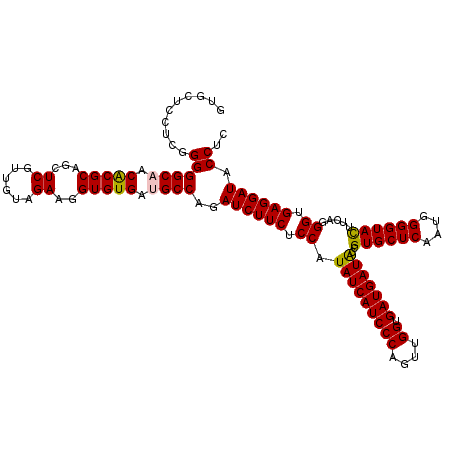
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:35:36 2006