
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 2,742,802 – 2,742,931 |
| Length | 129 |
| Max. P | 0.976199 |

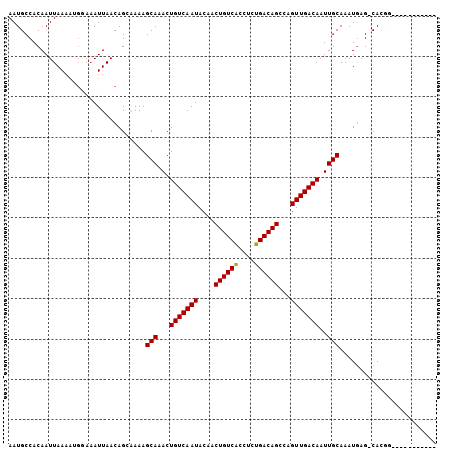
| Location | 2,742,802 – 2,742,902 |
|---|---|
| Length | 100 |
| Sequences | 6 |
| Columns | 106 |
| Reading direction | forward |
| Mean pairwise identity | 84.06 |
| Mean single sequence MFE | -21.73 |
| Consensus MFE | -17.10 |
| Energy contribution | -16.97 |
| Covariance contribution | -0.14 |
| Combinations/Pair | 1.06 |
| Mean z-score | -2.41 |
| Structure conservation index | 0.79 |
| SVM decision value | 0.72 |
| SVM RNA-class probability | 0.831111 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 2742802 100 + 20766785 AAUGCCACAAUUAAAAUGGAAAUUAACAGGGAAAGCACACUGUCAAUACGACUGUCACCUCUGACAGCCAGUUGACAAUUGCAAAUGAG-CACGGGCCACA----- ...(((........(((....)))..........(((...(((((((..(.((((((....)))))))..)))))))..))).......-....)))....----- ( -21.50) >DroPse_CAF1 26091 105 + 1 AAUGCCACAAUUAAAACGAAAAUUAACAGCAAAAGCAAACUGUCAAUACAACUGUCACUUCUGACAGCCAGUUGACAAUUGCAAAUGAG-CACAGGACGCAGGACA ..((((............................((((..(((((((....((((((....))))))...))))))).))))......(-(.......)).)).)) ( -20.80) >DroGri_CAF1 31512 88 + 1 AAUGCCACAAUUAAU------AUUAACAGCAAAAGCAAACUGUCAAUACAGCUGUCGCUCCUGACAGCCAGUUGACAAUUGCAAAUGAGGCAAC------------ ..((((..(((....------)))..........((((..(((((((...(((((((....)))))))..))))))).))))......))))..------------ ( -25.30) >DroEre_CAF1 18090 94 + 1 AAUGCCACAAUUAAAAUGGAAAUUAACAGGGAAAGCACACUGUCAAUACGACUGUCACCUCUGACAGCCGGUUGACAAUUGCAAAUAAG-CACGG----------- ..(((.........(((....)))..........(((...(((((((.((.((((((....)))))).)))))))))..)))......)-))...----------- ( -22.00) >DroYak_CAF1 27676 93 + 1 AAUGCCACAAUUAAAAUGGAAAUUAACAGGGAAAGCACACUGUCAAUACGACUGUCACCUCUGACAGCCAGUUGACAAUUGCAAAUGAG-CACG------------ ..(((((.......(((....)))..........(((...(((((((..(.((((((....)))))))..)))))))..)))...)).)-))..------------ ( -20.00) >DroPer_CAF1 26494 105 + 1 AAUGCCACAAUUAAAACGAAAAUUAACAGCAAAAGCAAACUGUCAAUACAACUGUCACUUCUGACAGCCAGUUGACAAUUGCAAAUGAG-CACAGGACGCAGGACA ..((((............................((((..(((((((....((((((....))))))...))))))).))))......(-(.......)).)).)) ( -20.80) >consensus AAUGCCACAAUUAAAAUGGAAAUUAACAGCAAAAGCAAACUGUCAAUACAACUGUCACCUCUGACAGCCAGUUGACAAUUGCAAAUGAG_CACGG___________ ..................................(((...(((((((....((((((....))))))...)))))))..)))........................ (-17.10 = -16.97 + -0.14)


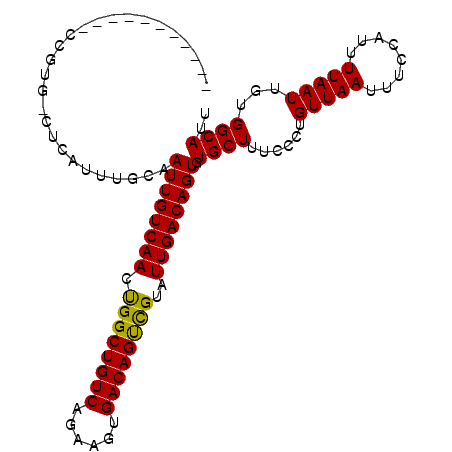
| Location | 2,742,802 – 2,742,902 |
|---|---|
| Length | 100 |
| Sequences | 6 |
| Columns | 106 |
| Reading direction | reverse |
| Mean pairwise identity | 84.06 |
| Mean single sequence MFE | -28.03 |
| Consensus MFE | -22.19 |
| Energy contribution | -21.67 |
| Covariance contribution | -0.53 |
| Combinations/Pair | 1.12 |
| Mean z-score | -2.49 |
| Structure conservation index | 0.79 |
| SVM decision value | 0.80 |
| SVM RNA-class probability | 0.853969 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 2742802 100 - 20766785 -----UGUGGCCCGUG-CUCAUUUGCAAUUGUCAACUGGCUGUCAGAGGUGACAGUCGUAUUGACAGUGUGCUUUCCCUGUUAAUUUCCAUUUUAAUUGUGGCAUU -----....(((((..-.......(((((((((((.(((((((((....)))))))))..)))))))).))).......(((((........))))))).)))... ( -27.20) >DroPse_CAF1 26091 105 - 1 UGUCCUGCGUCCUGUG-CUCAUUUGCAAUUGUCAACUGGCUGUCAGAAGUGACAGUUGUAUUGACAGUUUGCUUUUGCUGUUAAUUUUCGUUUUAAUUGUGGCAUU .............(((-(.(((.(((((((((((.(((.....)))...)))))))))))((((((((........))))))))..............))))))). ( -27.70) >DroGri_CAF1 31512 88 - 1 ------------GUUGCCUCAUUUGCAAUUGUCAACUGGCUGUCAGGAGCGACAGCUGUAUUGACAGUUUGCUUUUGCUGUUAAU------AUUAAUUGUGGCAUU ------------..((((......(((((((......(((((((......)))))))(((((((((((........)))))))))------))))))))))))).. ( -30.10) >DroEre_CAF1 18090 94 - 1 -----------CCGUG-CUUAUUUGCAAUUGUCAACCGGCUGUCAGAGGUGACAGUCGUAUUGACAGUGUGCUUUCCCUGUUAAUUUCCAUUUUAAUUGUGGCAUU -----------...((-(......)))((((((((.(((((((((....)))))))))..))))))))(((((......(((((........)))))...))))). ( -28.30) >DroYak_CAF1 27676 93 - 1 ------------CGUG-CUCAUUUGCAAUUGUCAACUGGCUGUCAGAGGUGACAGUCGUAUUGACAGUGUGCUUUCCCUGUUAAUUUCCAUUUUAAUUGUGGCAUU ------------.(((-(.(((..(((((((((((.(((((((((....)))))))))..)))))))).))).......(((((........))))).))))))). ( -27.20) >DroPer_CAF1 26494 105 - 1 UGUCCUGCGUCCUGUG-CUCAUUUGCAAUUGUCAACUGGCUGUCAGAAGUGACAGUUGUAUUGACAGUUUGCUUUUGCUGUUAAUUUUCGUUUUAAUUGUGGCAUU .............(((-(.(((.(((((((((((.(((.....)))...)))))))))))((((((((........))))))))..............))))))). ( -27.70) >consensus ___________CCGUG_CUCAUUUGCAAUUGUCAACUGGCUGUCAGAAGUGACAGUCGUAUUGACAGUGUGCUUUCCCUGUUAAUUUCCAUUUUAAUUGUGGCAUU ...........................((((((((.((((((((......))))))))..)))))))).((((......(((((........)))))...)))).. (-22.19 = -21.67 + -0.53)

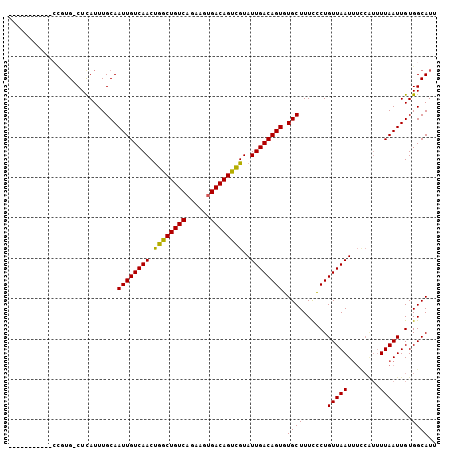
| Location | 2,742,834 – 2,742,931 |
|---|---|
| Length | 97 |
| Sequences | 6 |
| Columns | 113 |
| Reading direction | forward |
| Mean pairwise identity | 77.94 |
| Mean single sequence MFE | -27.30 |
| Consensus MFE | -17.09 |
| Energy contribution | -17.45 |
| Covariance contribution | 0.36 |
| Combinations/Pair | 1.05 |
| Mean z-score | -1.94 |
| Structure conservation index | 0.63 |
| SVM decision value | 1.04 |
| SVM RNA-class probability | 0.905060 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 2742834 97 + 20766785 AAGCACACUGUCAAUACGACUGUCACCUCUGACAGCCAGUUGACAAUUGCAAAUGAGCACGGGCCACA------------CAGCCCGCAGAG----AUCCAUGGUAUCCGUAU ..(((...(((((((..(.((((((....)))))))..)))))))..)))......((.(((((....------------..)))))..(.(----(((....).)))))).. ( -27.20) >DroSec_CAF1 27026 97 + 1 AAGCACACUGUCAAUACGACUGUCACCUCUGACAGCCAGUUGACAAUUGCAAAUGAGCACGGGCCACA------------CAGCCCGCAGAG----AUCCAUGGUGUCCGGAU ..(((...(((((((..(.((((((....)))))))..)))))))..)))...((..(((((((....------------..)))).((...----.....)))))..))... ( -29.20) >DroSim_CAF1 13454 97 + 1 AAGCACACUGUCAAUACGACUGUCACCUCUGACAGCCAGUUGACAAUUGCAAAUGAGCACGGGCCACA------------CAGCCCGCAGAG----AUCCAUGGUGUCCGGAU ..(((...(((((((..(.((((((....)))))))..)))))))..)))...((..(((((((....------------..)))).((...----.....)))))..))... ( -29.20) >DroEre_CAF1 18122 80 + 1 AAGCACACUGUCAAUACGACUGUCACCUCUGACAGCCGGUUGACAAUUGCAAAUAAGCACGG---------------------------GAG----UUCCAUGGUGUCCGC-- ..(((...(((((((.((.((((((....)))))).)))))))))..)))......((((.(---------------------------(..----..))...))))....-- ( -24.90) >DroYak_CAF1 27708 78 + 1 AAGCACACUGUCAAUACGACUGUCACCUCUGACAGCCAGUUGACAAUUGCAAAUGAGCACG-----------------------------AG----UUCCAUGGUAUCCAG-- ..(((...(((((((..(.((((((....)))))))..)))))))..)))....((((...-----------------------------.)----)))..(((...))).-- ( -19.60) >DroPer_CAF1 26526 111 + 1 AAGCAAACUGUCAAUACAACUGUCACUUCUGACAGCCAGUUGACAAUUGCAAAUGAGCACAGGACGCAGGACACGGUAUACAGUACACAGUGCACAGUGCACAGUGCCCAG-- ..((((..(((((((....((((((....))))))...))))))).))))...((.((((.(..(((.(..(((.((((...))))...)))..).)))..).)))).)).-- ( -33.70) >consensus AAGCACACUGUCAAUACGACUGUCACCUCUGACAGCCAGUUGACAAUUGCAAAUGAGCACGGGCCACA____________CAGCCCGCAGAG____AUCCAUGGUGUCCGG__ ..((((..(((((((....((((((....))))))...)))))))..(((......)))............................................))))...... (-17.09 = -17.45 + 0.36)

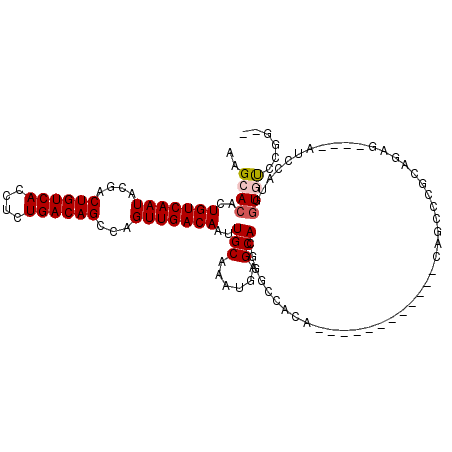
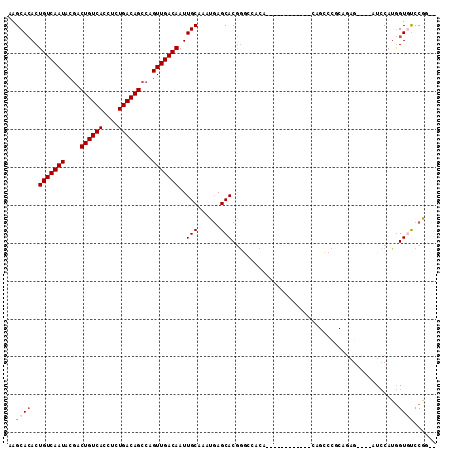
| Location | 2,742,834 – 2,742,931 |
|---|---|
| Length | 97 |
| Sequences | 6 |
| Columns | 113 |
| Reading direction | reverse |
| Mean pairwise identity | 77.94 |
| Mean single sequence MFE | -34.33 |
| Consensus MFE | -23.97 |
| Energy contribution | -23.55 |
| Covariance contribution | -0.42 |
| Combinations/Pair | 1.17 |
| Mean z-score | -2.44 |
| Structure conservation index | 0.70 |
| SVM decision value | 1.77 |
| SVM RNA-class probability | 0.976199 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 2742834 97 - 20766785 AUACGGAUACCAUGGAU----CUCUGCGGGCUG------------UGUGGCCCGUGCUCAUUUGCAAUUGUCAACUGGCUGUCAGAGGUGACAGUCGUAUUGACAGUGUGCUU ....((((.(....)))----))..(((((((.------------...)))))))........(((((((((((.(((((((((....)))))))))..)))))))).))).. ( -36.30) >DroSec_CAF1 27026 97 - 1 AUCCGGACACCAUGGAU----CUCUGCGGGCUG------------UGUGGCCCGUGCUCAUUUGCAAUUGUCAACUGGCUGUCAGAGGUGACAGUCGUAUUGACAGUGUGCUU (((((.......)))))----....(((((((.------------...)))))))........(((((((((((.(((((((((....)))))))))..)))))))).))).. ( -37.70) >DroSim_CAF1 13454 97 - 1 AUCCGGACACCAUGGAU----CUCUGCGGGCUG------------UGUGGCCCGUGCUCAUUUGCAAUUGUCAACUGGCUGUCAGAGGUGACAGUCGUAUUGACAGUGUGCUU (((((.......)))))----....(((((((.------------...)))))))........(((((((((((.(((((((((....)))))))))..)))))))).))).. ( -37.70) >DroEre_CAF1 18122 80 - 1 --GCGGACACCAUGGAA----CUC---------------------------CCGUGCUUAUUUGCAAUUGUCAACCGGCUGUCAGAGGUGACAGUCGUAUUGACAGUGUGCUU --(((((...(((((..----...---------------------------)))))....))))).((((((((.(((((((((....)))))))))..))))))))...... ( -29.80) >DroYak_CAF1 27708 78 - 1 --CUGGAUACCAUGGAA----CU-----------------------------CGUGCUCAUUUGCAAUUGUCAACUGGCUGUCAGAGGUGACAGUCGUAUUGACAGUGUGCUU --........((((...----..-----------------------------)))).......(((((((((((.(((((((((....)))))))))..)))))))).))).. ( -23.60) >DroPer_CAF1 26526 111 - 1 --CUGGGCACUGUGCACUGUGCACUGUGUACUGUAUACCGUGUCCUGCGUCCUGUGCUCAUUUGCAAUUGUCAACUGGCUGUCAGAAGUGACAGUUGUAUUGACAGUUUGCUU --.(((((((.(..(.(.(..(((.(((((...))))).)))..).).)..).)))))))...(((((((((((..((((((((....))))))))...))))))).)))).. ( -40.90) >consensus __CCGGACACCAUGGAU____CUCUGCGGGCUG____________UGUGGCCCGUGCUCAUUUGCAAUUGUCAACUGGCUGUCAGAGGUGACAGUCGUAUUGACAGUGUGCUU ....(.((((............................................(((......)))..((((((.(((((((((....)))))))))..)))))))))).).. (-23.97 = -23.55 + -0.42)
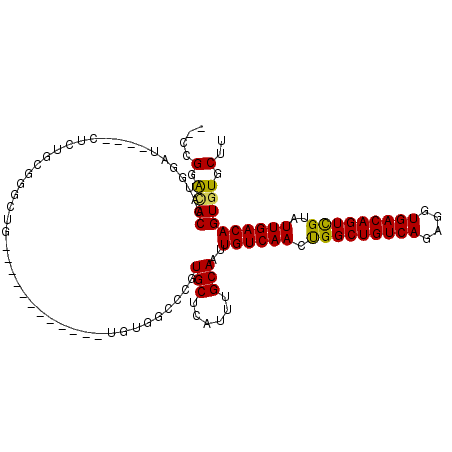
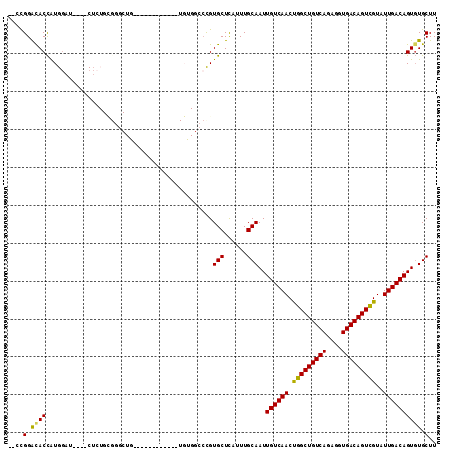
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:40:24 2006