
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 16,066,409 – 16,066,569 |
| Length | 160 |
| Max. P | 0.996994 |

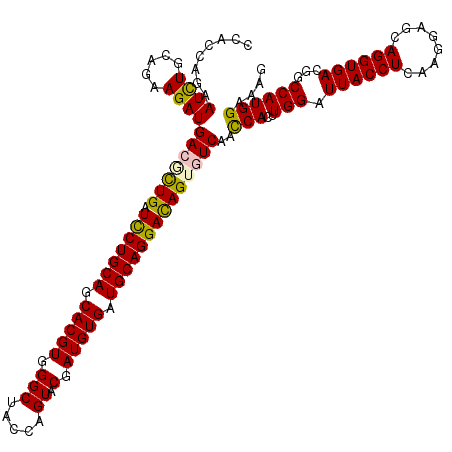
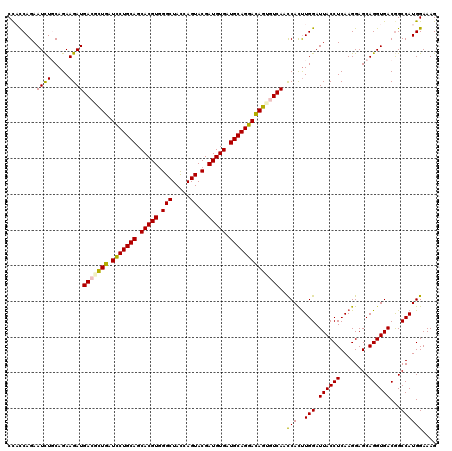
| Location | 16,066,409 – 16,066,529 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 93.28 |
| Mean single sequence MFE | -43.22 |
| Consensus MFE | -38.84 |
| Energy contribution | -38.65 |
| Covariance contribution | -0.19 |
| Combinations/Pair | 1.16 |
| Mean z-score | -2.49 |
| Structure conservation index | 0.90 |
| SVM decision value | 2.78 |
| SVM RNA-class probability | 0.996994 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 16066409 120 - 20766785 CCACCAGAAUCUGCAGAAGAUGACGCUGAUUCUGCAGCACGUGGGCUACCAGUACGAUGUGAUGCAGGACAGUGUCAACCACUUGGAUUACCUCAAGGAGCAGGUGACGGCCAUGGAAAG (((((...((((((......((((((((.(((((((.(((((.(((.....)).).))))).)))))))))))))))....(((((......)))))..))))))...))...))).... ( -45.40) >DroPse_CAF1 141325 120 - 1 CUCUCAGAAUCUUCAGAAGAUGAGCUUGAUCCUGCAGCACGUUGGCUAUCAGUACGAUGUGAUGCAGGACAGCGUCAAUCAUUUGGAUUACCUCAAGGAGCAGGUGACGGCCAUGGAAAG ((((..((...((((((.((((((((.(.(((((((.(((((((((.....)).))))))).))))))))))).)).))).)))))).....))..))))..(((....)))........ ( -36.30) >DroSim_CAF1 124042 120 - 1 CCACCAGAAUCUGCAGAAGAUGACGCUGAUCCUGCAGCACGUGGGCUACCAGUACGAUGUGAUGCAGGACAGUGUCAACCACUUGGAUUACCUCAAGGAGCAGGUGACGGCCAUGGAAAG (((((...((((((......((((((((.(((((((.(((((.(((.....)).).))))).)))))))))))))))....(((((......)))))..))))))...))...))).... ( -48.30) >DroEre_CAF1 126895 120 - 1 CCACCAGAAUUUGCAGAAGAUGACACUGAUCCUGCAGCACGUGGGCUACCAGUACGAUGUGAUGCAGGAUAGUGUCAACCACUUGGAUUACCUCAAGGAGCAGGUGACGGCCAUGGAAAG (((((...((((((......((((((((.(((((((.(((((.(((.....)).).))))).)))))))))))))))....(((((......)))))..))))))...))...))).... ( -44.30) >DroYak_CAF1 131428 120 - 1 CCACCAGAAUCUGCAGAAGAUGACGCUGAUCCUGCAGCACGUGGGCUACCAGUACGAUGUGAUGCAGGACAGUGUCAACCACCUGGAUUACCUCAAGGAGCAGGUGACAGCCAUGGAAAG ...(((....(((.......((((((((.(((((((.(((((.(((.....)).).))))).)))))))))))))))..((((((.....((....))..)))))).)))...))).... ( -46.60) >DroPer_CAF1 134990 120 - 1 CUCUCAGAAUCUUCAGAAGAUGAGCCUGAUCCUGCAGCACGUUGGCUAUCAGUACGAUGUGAUGCAGGACAGCGUCAAUCAUUUGGAUUACCUCAAGGAGCAGGUGACGGCCAUGGAAAG ((((..((...((((((.(((..(((((.(((((((.(((((((((.....)).))))))).)))))))))).))..))).)))))).....))..))))..(((....)))........ ( -38.40) >consensus CCACCAGAAUCUGCAGAAGAUGACGCUGAUCCUGCAGCACGUGGGCUACCAGUACGAUGUGAUGCAGGACAGUGUCAACCACUUGGAUUACCUCAAGGAGCAGGUGACGGCCAUGGAAAG ........((((.....))))(((((((.(((((((.(((((.(((.....)).).))))).))))))))))))))..(((..(((.((((((........))))))...)))))).... (-38.84 = -38.65 + -0.19)

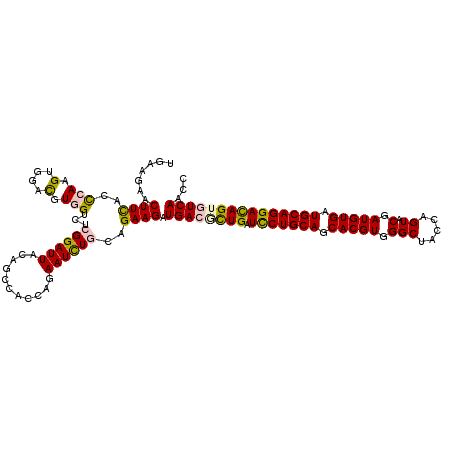
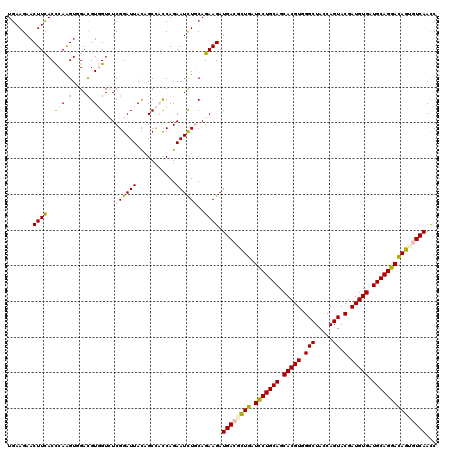
| Location | 16,066,449 – 16,066,569 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 90.50 |
| Mean single sequence MFE | -41.40 |
| Consensus MFE | -32.80 |
| Energy contribution | -32.83 |
| Covariance contribution | 0.03 |
| Combinations/Pair | 1.22 |
| Mean z-score | -2.18 |
| Structure conservation index | 0.79 |
| SVM decision value | -0.08 |
| SVM RNA-class probability | 0.500000 |
| Prediction | OTHER |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 16066449 120 - 20766785 UGAAGAACUUCACCCAAGUGGACGUGGUCUCGGAUUACAGCCACCAGAAUCUGCAGAAGAUGACGCUGAUUCUGCAGCACGUGGGCUACCAGUACGAUGUGAUGCAGGACAGUGUCAACC .......((((......((((..((((((...))))))..))))(((...)))..)))).((((((((.(((((((.(((((.(((.....)).).))))).)))))))))))))))... ( -43.00) >DroPse_CAF1 141365 120 - 1 UGAAGAACUUUACUCAGGUCGAUGUCGUGUCGGAUUAUAGCUCUCAGAAUCUUCAGAAGAUGAGCUUGAUCCUGCAGCACGUUGGCUAUCAGUACGAUGUGAUGCAGGACAGCGUCAAUC ..........((((..(((((((((..(((.((((((.(((((.....((((.....))))))))))))))).)))..)))))))))...)))).(((.((((((......))))))))) ( -36.40) >DroSim_CAF1 124082 120 - 1 UGAAGAACUUCACCCAAGUGGACGUGGUCUCGGAUUACAGCCACCAGAAUCUGCAGAAGAUGACGCUGAUCCUGCAGCACGUGGGCUACCAGUACGAUGUGAUGCAGGACAGUGUCAACC .......((((......((((..((((((...))))))..))))(((...)))..)))).((((((((.(((((((.(((((.(((.....)).).))))).)))))))))))))))... ( -45.90) >DroEre_CAF1 126935 120 - 1 UGAAGAACUUCACCCAAGUUGACGUGGUCUCGGAUUAUAGCCACCAGAAUUUGCAGAAGAUGACACUGAUCCUGCAGCACGUGGGCUACCAGUACGAUGUGAUGCAGGAUAGUGUCAACC .......((((...((((((...(((((...........)))))...))))))..)))).((((((((.(((((((.(((((.(((.....)).).))))).)))))))))))))))... ( -42.60) >DroYak_CAF1 131468 120 - 1 UGAAGAACUUCACCCAAGUGGACGUGGUCUCGGAUUAUAGCCACCAGAAUCUGCAGAAGAUGACGCUGAUCCUGCAGCACGUGGGCUACCAGUACGAUGUGAUGCAGGACAGUGUCAACC .......((((......((((..((((((...))))))..))))(((...)))..)))).((((((((.(((((((.(((((.(((.....)).).))))).)))))))))))))))... ( -44.90) >DroPer_CAF1 135030 120 - 1 UGAAGAACUUUACUCAGGUCGAUGUCGUGUCGGAUUACAGCUCUCAGAAUCUUCAGAAGAUGAGCCUGAUCCUGCAGCACGUUGGCUAUCAGUACGAUGUGAUGCAGGACAGCGUCAAUC ((((((((((.....))))((((.....)))).................))))))...(((..(((((.(((((((.(((((((((.....)).))))))).)))))))))).))..))) ( -35.60) >consensus UGAAGAACUUCACCCAAGUGGACGUGGUCUCGGAUUACAGCCACCAGAAUCUGCAGAAGAUGACGCUGAUCCUGCAGCACGUGGGCUACCAGUACGAUGUGAUGCAGGACAGUGUCAACC .......((((..(((.(....).)))...((((((...........))))))..)))).((((((((.(((((((.(((((.(((.....)).).))))).)))))))))))))))... (-32.80 = -32.83 + 0.03)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:32:08 2006