| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
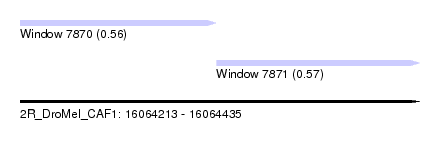
| Location | 16,064,213 – 16,064,435 |
| Length | 222 |
| Max. P | 0.565503 |

| Location | 16,064,213 – 16,064,322 |
|---|---|
| Length | 109 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 79.82 |
| Mean single sequence MFE | -34.92 |
| Consensus MFE | -25.33 |
| Energy contribution | -24.92 |
| Covariance contribution | -0.41 |
| Combinations/Pair | 1.26 |
| Mean z-score | -1.11 |
| Structure conservation index | 0.73 |
| SVM decision value | 0.06 |
| SVM RNA-class probability | 0.561908 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
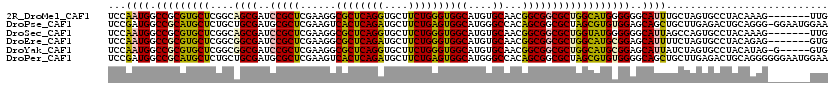
>2R_DroMel_CAF1 16064213 109 + 20766785 UUUCACAUCAUUCAGCGAACU-----------CCAUCACCUACGGAUGGUACUGUCCAUCUUCGAUCUCUCCAUGCUCAGAUUGAGCGGCCAGCGCCAGACGCUGUGCCUCCUGUUGGCG ..............((.(((.-----------...........((((((......)))))).............((((.....))))((((((((.....))))).)))....))).)). ( -31.60) >DroSec_CAF1 117837 109 + 1 UUUCACAUCAUUCAGCGAACU-----------GCAUCUCCUACGGAUGGUACUGUCCAUCUUCGAUCUCUCCAUGCUCGGAUUGGGCGGCCAGUGCCAGACGCUGUGCCUCCUGUUGCCG ............(((((....-----------((((......((..((((((((.((...((((((((..........)))))))).)).))))))))..))..))))....)))))... ( -28.20) >DroSim_CAF1 121849 109 + 1 UUUCACAUCAUUCAGCGAACU-----------GCAUCUCCUACGGAUGGUACUGUCCAUCUUCGAUCUCUCCAUGCUCGGACUGGGCGGCCAGUGCCAGACGCUGUGCCUCCUGUUGCCG ..............((((...-----------.(((((.....))))).....(((((..(((((.(.......).))))).)))))((((((((.....))))).))).....)))).. ( -27.90) >DroEre_CAF1 124690 120 + 1 UUUCAUAUCAUUCAGCAGACUGGCUCAGCACAGCACCUCUUAUGGAUGGUACUGACCAUCCUCGAUCUCUCCAUGCUCGGACUGGGCGGCCAGUGCCAGACGCUGUGCCUCCUGUUGGCG ...........(((((((...(((.((((......((......)).((((((((.((..((((((.(.......).))))...))..)).))))))))...)))).)))..))))))).. ( -43.30) >DroYak_CAF1 129229 120 + 1 UUUCAUAUCAUUCAGCAGACUGUUUCGGCACAGCACCUCUUAUGGAUGGUACUGACCAUCCUCGAUCCCUCCAUGCUCGGAUUGAGCGGCCAGUGCCAGACGCUGUGCCUCCUGUUGGCG ...........(((((((........((((((((.((......)).((((((((.((...((((((((..........)))))))).)).))))))))...))))))))..))))))).. ( -47.60) >DroPer_CAF1 132708 99 + 1 UU--AAUACUUGCAGAG---U----------------GCUCAAGGAUGAUACUGUCCAUCCUCCACCUCUCCAUGCUCCGACUGGGCAGCUAGAGCCAGGCGCUGCGCCUCCUGCUGGGC ..--....((.((((.(---.----------------((((.((((((........))))))...........(((((.....)))))....)))))(((((...))))).)))).)).. ( -30.90) >consensus UUUCACAUCAUUCAGCGAACU___________GCAUCUCCUACGGAUGGUACUGUCCAUCCUCGAUCUCUCCAUGCUCGGACUGGGCGGCCAGUGCCAGACGCUGUGCCUCCUGUUGGCG ............(((((..........................((((((......)))))).............((((.....))))((((((((.....))))).)))...)))))... (-25.33 = -24.92 + -0.41)

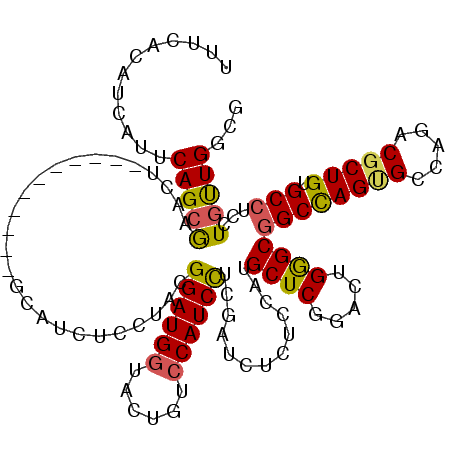
| Location | 16,064,322 – 16,064,435 |
|---|---|
| Length | 113 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 82.41 |
| Mean single sequence MFE | -52.02 |
| Consensus MFE | -37.17 |
| Energy contribution | -36.12 |
| Covariance contribution | -1.05 |
| Combinations/Pair | 1.34 |
| Mean z-score | -1.49 |
| Structure conservation index | 0.71 |
| SVM decision value | 0.06 |
| SVM RNA-class probability | 0.565503 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 16064322 113 + 20766785 UCCAAUGGCCGCGUGCUCGGCAGCGAUCCGCUCGAAGGCGCUCAGGUGCUUCUGGGUGGCAUGUGCAACGGCGGCGCUGGCAUGGGGGGCAUUUGCUAGUGCCUACAAAG-------UUG ((((...((((((((((..(((.((..(((((((.((((((....)))))).)))))))..)))))...))).)))).))).))))(((((((....)))))))......-------... ( -47.90) >DroPse_CAF1 139142 119 + 1 UCCGAUGGCCGCAUGCUCUGCUGCGAUGCGCUCGAAGUCACUCAGAUGCUUCUGAGUGGCAUGGGCCACAGCGGCGCUAGCGUGUGGAGCAGCUGCUUGAGACUGCAGGG-GGAAUGGAA ((((....(((((((((.((((((..((.(((((..((((((((((....)))))))))).)))))))..))))))..))))))))).((((((.....)).))))....-....)))). ( -57.50) >DroSec_CAF1 117946 113 + 1 UCCAAUGGCCGCGUGCUCGGCAGCGAUCCGCUCGAAGGCGCUCAGGUGCUUCUGGGUGGCAUGUGCAACGGCGGCGCUGGUAUGGGGGGCAUUAGCCAGUGCCUACAAAG-------UUG ..((((.((((..((((((....))).(((((((.((((((....)))))).))))))).....))).))))((((((((((((.....)))..)))))))))......)-------))) ( -46.40) >DroEre_CAF1 124810 113 + 1 UCCAAUGGCCGCGUGCUCGGCGGCGAUCCGCUCGAAGGCGCUCAGAUGCUUCUGGGUGGCAUGUGCAACGGCGGCGCUGGCAUGCGGAGCAUUUUCUAGUGCCUACAGAG-------GUG .((..((((((..((((((((((....)))).))).(.((((((((....)))))))).)....))).))))((((((((.((((...))))...))))))))..))..)-------).. ( -50.10) >DroYak_CAF1 129349 114 + 1 UCCAAUGGCCGCGUGCUCGGCGGCGAUCCGCUCGAAGGCGCUCAGGUGCUUCUGGGUGGCAUGUGCAACGGCGGCGCUGGCAUGCGGAGCAUUAUCUAGUGCCUACAUAG-G-----GUG (((.(((.((((((((.(((((.(...(((((((.((((((....)))))).))))))).....((....))).))))))))))))).(((((....)))))...))).)-)-----).. ( -52.70) >DroPer_CAF1 132807 120 + 1 UCCGAUGGCCGCAUGCUCUGCUGCGAUGCGCUCGAAGUCACUCAGAUGCUUCUGAGUGGCAUGGGCCACAGCGGCGCUAGCGUGUGGGGCAGCUGCUUGAGACUGCAGGGGGGAAUGGAA ((((....(((((((((.((((((..((.(((((..((((((((((....)))))))))).)))))))..))))))..))))))))).((((((.....)).)))).........)))). ( -57.50) >consensus UCCAAUGGCCGCGUGCUCGGCAGCGAUCCGCUCGAAGGCGCUCAGAUGCUUCUGGGUGGCAUGUGCAACGGCGGCGCUGGCAUGCGGAGCAUUUGCUAGUGCCUACAGAG_______GUG ...((((.(((((((((....((((..(((((......((((((((....))))))))((....))...))))))))))))))))))..))))........................... (-37.17 = -36.12 + -1.05)
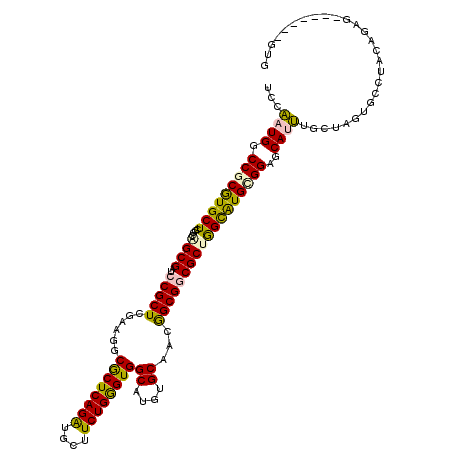
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:32:03 2006