
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 15,326,036 – 15,326,316 |
| Length | 280 |
| Max. P | 0.909757 |

| Location | 15,326,036 – 15,326,151 |
|---|---|
| Length | 115 |
| Sequences | 5 |
| Columns | 116 |
| Reading direction | reverse |
| Mean pairwise identity | 93.76 |
| Mean single sequence MFE | -32.88 |
| Consensus MFE | -27.56 |
| Energy contribution | -27.88 |
| Covariance contribution | 0.32 |
| Combinations/Pair | 1.09 |
| Mean z-score | -1.48 |
| Structure conservation index | 0.84 |
| SVM decision value | 0.53 |
| SVM RNA-class probability | 0.771021 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 15326036 115 - 20766785 UCUGAAUAUGGACUUGGGAUG-GGUGAGACUGAUAUGUGAUAUUAGUUUGAUCUAUUGUGAUUUCAUGCAGGGCUGCAACGUUCUCUGGAAAAUGAAUAAGUCCGCAAGAGGCACC .........(((((((.((((-(((.((((((((((...)))))))))).))))))).....(((((.(((((..((...)).)))))....))))))))))))((.....))... ( -32.40) >DroSec_CAF1 132010 115 - 1 UCUGAAUAUGGACUCGGGAUG-GGUGAGACUGAUAUGUGAUAUUAGUUUGAUCUAUUGUGAUUUCAUGCAGGGAUGCAACGUUCUCUGGAAAAUGAAUAAGCCCGCAAAAGGCACC ......((((((.(((.((((-(((.((((((((((...)))))))))).))))))).))).))))))((((((........))))))............(((.......)))... ( -35.30) >DroSim_CAF1 109771 116 - 1 UCUGAAUAUGGACUCGGGAUGGGGUGAGACUGAUAUGUGAUAUUAGUUUGAUCUAUUGUGAUUUCAUGCAGGGAUGCAACGUUCUCUGGAAAAUGAAUAAGCCCGCAAAAGGCACC ......((((((.(((.((((((.(.((((((((((...)))))))))).))))))).))).))))))((((((........))))))............(((.......)))... ( -31.50) >DroEre_CAF1 118645 115 - 1 CCUGAAUAUGGACUGUGGAUG-GGUGAGACUGAUAUGCGAUAUUAGUUUGAUCUAUUGUGAUUUCAUGCAGGGAUGCAGCGUUCCCUGGAAAAUGUAAAAGCCCGCAAGAGGCAGC ((((..((((((.....((((-(((.((((((((((...)))))))))).))))))).....))))))))))..((((...(((....)))..))))...((((....).)))... ( -36.20) >DroYak_CAF1 122715 115 - 1 UCUGAAUAUGGACUUGGUAUG-GGUGAGACUGAUAUGCGAUAUUAGUUUGAUCUAUUGUGAUUUCAUGCAGGGAUGCAACGUUCUCUGGAAAAUGUCUAAGCCCGCAAAAGGCAGC ........(((((.....(((-(((.((((((((((...)))))))))).))))))..........(.((((((........)))))).)....))))).(((.......)))... ( -29.00) >consensus UCUGAAUAUGGACUCGGGAUG_GGUGAGACUGAUAUGUGAUAUUAGUUUGAUCUAUUGUGAUUUCAUGCAGGGAUGCAACGUUCUCUGGAAAAUGAAUAAGCCCGCAAAAGGCACC ......((((((.(((.((((.(((.((((((((((...)))))))))).))))))).))).))))))((((((........))))))............(((.......)))... (-27.56 = -27.88 + 0.32)

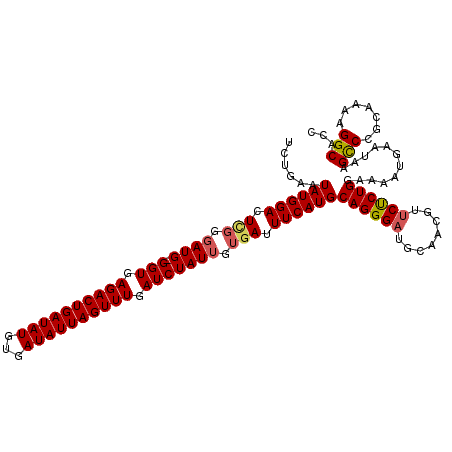
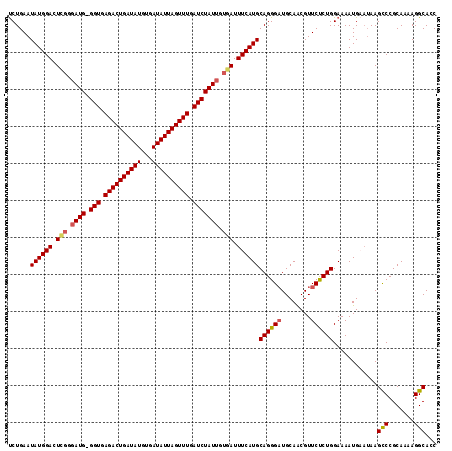
| Location | 15,326,072 – 15,326,178 |
|---|---|
| Length | 106 |
| Sequences | 6 |
| Columns | 109 |
| Reading direction | reverse |
| Mean pairwise identity | 79.36 |
| Mean single sequence MFE | -27.75 |
| Consensus MFE | -16.83 |
| Energy contribution | -17.82 |
| Covariance contribution | 0.99 |
| Combinations/Pair | 1.16 |
| Mean z-score | -1.98 |
| Structure conservation index | 0.61 |
| SVM decision value | 0.71 |
| SVM RNA-class probability | 0.829539 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 15326072 106 - 20766785 ACUUAUGUAC--AUAUAUGCGUGUGUUCGUCUGAAUAUGGACUUGGGAUG-GGUGAGACUGAUAUGUGAUAUUAGUUUGAUCUAUUGUGAUUUCAUGCAGGGCUGCAAC .....(((((--.....((((((...((((((......))))..(.((((-(((.((((((((((...)))))))))).))))))).)))...))))))..).)))).. ( -29.90) >DroPse_CAF1 158093 80 - 1 GUACGU----------GUACAUGUAUGUGUC------------------G-GGGGGUACUGAUAUGUGAUAUUAGUUUGAUCUAUUGUGAUUUCGUUCAGGGCUGCAAA ((((((----------....)))))).....------------------.-.(.((..((((...((.(((.(((......))).))).)).....))))..)).)... ( -16.00) >DroSec_CAF1 132046 108 - 1 ACUUAUGUACAUAUAUAUGCGUGUGUUCGUCUGAAUAUGGACUCGGGAUG-GGUGAGACUGAUAUGUGAUAUUAGUUUGAUCUAUUGUGAUUUCAUGCAGGGAUGCAAC ((.((((((....)))))).))((((((..(((..((((((.(((.((((-(((.((((((((((...)))))))))).))))))).))).)))))))))))))))... ( -31.90) >DroSim_CAF1 109807 109 - 1 ACUUAUGUACGAAUAUAUGCGUGUGUUCGUCUGAAUAUGGACUCGGGAUGGGGUGAGACUGAUAUGUGAUAUUAGUUUGAUCUAUUGUGAUUUCAUGCAGGGAUGCAAC .....((((((((((((....))))))))((((..((((((.(((.((((((.(.((((((((((...)))))))))).))))))).))).))))))))))..)))).. ( -31.70) >DroEre_CAF1 118681 98 - 1 AGUUGU----------GUGCGUGUGUUCGCCUGAAUAUGGACUGUGGAUG-GGUGAGACUGAUAUGCGAUAUUAGUUUGAUCUAUUGUGAUUUCAUGCAGGGAUGCAGC .(((((----------((.(..((((((....))))))...((((.((((-(((.((((((((((...)))))))))).)))))))(((....)))))))).))))))) ( -30.00) >DroYak_CAF1 122751 98 - 1 AUUCGU----------AUGCGUGUGUUCGUCUGAAUAUGGACUUGGUAUG-GGUGAGACUGAUAUGCGAUAUUAGUUUGAUCUAUUGUGAUUUCAUGCAGGGAUGCAAC ((((..----------.((((((...((((((......)))).....(((-(((.((((((((((...)))))))))).))))))...))...)))))).))))..... ( -27.00) >consensus ACUUAU__________AUGCGUGUGUUCGUCUGAAUAUGGACUCGGGAUG_GGUGAGACUGAUAUGUGAUAUUAGUUUGAUCUAUUGUGAUUUCAUGCAGGGAUGCAAC .................((((((...((((((......)))).........(((.((((((((((...)))))))))).)))......))...)))))).......... (-16.83 = -17.82 + 0.99)

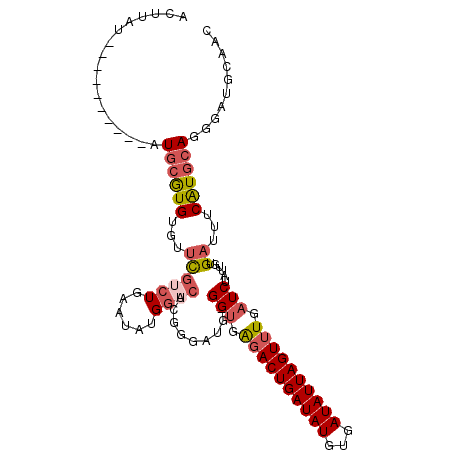
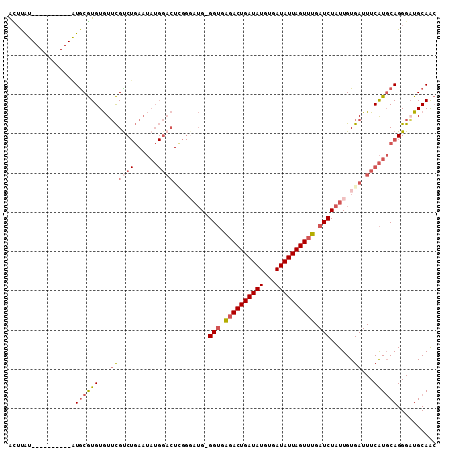
| Location | 15,326,151 – 15,326,258 |
|---|---|
| Length | 107 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 78.92 |
| Mean single sequence MFE | -35.47 |
| Consensus MFE | -23.54 |
| Energy contribution | -25.07 |
| Covariance contribution | 1.53 |
| Combinations/Pair | 1.12 |
| Mean z-score | -1.49 |
| Structure conservation index | 0.66 |
| SVM decision value | 0.43 |
| SVM RNA-class probability | 0.734169 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 15326151 107 + 20766785 CGAACACA-CGCAUAUAU--GUAC----------AUAAGUGCGACGAAGGGCUUGUCAGCGGGCCAUCUUUGUCGCCGCGGGCUCAGACAGAGGAGUGCACUGAGGAAAAUAGCGUGUUA .....(((-(((......--....----------....(((((((((((((((((....))))))..)))))))).)))(.((((........)))).).............)))))).. ( -37.90) >DroSec_CAF1 132125 109 + 1 CGAACACA-CGCAUAUAUAUGUAC----------AUAAGUGCGACGAAGGGCUUGUCAGCGGGCCAUCUUUGUCGCCGCGGGCUCAGACAGAGGACUGCACUGGGAAAAAUAGCGUGUUA .....(((-(((............----------....(.(((((((((((((((....))))))..))))))))))((((.(((.....)))..)))).............)))))).. ( -40.00) >DroSim_CAF1 109887 109 + 1 CGAACACA-CGCAUAUAUUCGUAC----------AUAAGUGCGACGAAGGGCUUGUCAGCGGGCCAUCUUUGUCGCCGCGGGCUCAGACAGAGGACUGCACUGAGAAAAAUAGCGUGUUA .....(((-(((.(((.(((....----------....(.(((((((((((((((....))))))..))))))))))((((.(((.....)))..)))).....)))..))))))))).. ( -40.10) >DroEre_CAF1 118760 99 + 1 CGAACACA-CGCAC--------------------ACAACUGCGACGAAGGACUUGUCAGCGGGCCAUCGUUGUCGCCGCGGGCUCAGACACAGGACUGCACUGAGCGAAAAAACGCCUUG ........-.((..--------------------.(..(.(((((((.((.((((....))))))....))))))).)..)((((((...((....))..))))))........)).... ( -26.90) >DroYak_CAF1 122830 99 + 1 CGAACACA-CGCAU--------------------ACGAAUGCGACGAAGGGCUUGUCAGCGGGCCAUCGCUGUCGCCGCGGGCUCAGACACAGGACGGCACUAAGAAAAAUAACGUGUUA ..(((((.-(((((--------------------....)))))..((..((((((....)))))).))(((((((((...))).(.......)))))))...............))))). ( -29.90) >DroAna_CAF1 105846 116 + 1 --AACUCAUCGUAUAUAU--GGACUAUGUGUAGAGCUACUGCGACGAAGGGCUUGUCAACAGGCCAUCUUUGUCGCCGCGGGCACUGACACAUGCAGGCACUGAGAGAAAUUACUGAUCA --..((((..((.....(--(...(((((((((.(((...(((((((((((((((....))))))..)))))))))....))).)).))))))))).))..))))............... ( -38.00) >consensus CGAACACA_CGCAUAUAU__GUAC__________ACAAGUGCGACGAAGGGCUUGUCAGCGGGCCAUCUUUGUCGCCGCGGGCUCAGACACAGGACUGCACUGAGAAAAAUAACGUGUUA ..(((((.................................(((((((((((((((....))))))..))))))))).((((.((........)).))))...............))))). (-23.54 = -25.07 + 1.53)

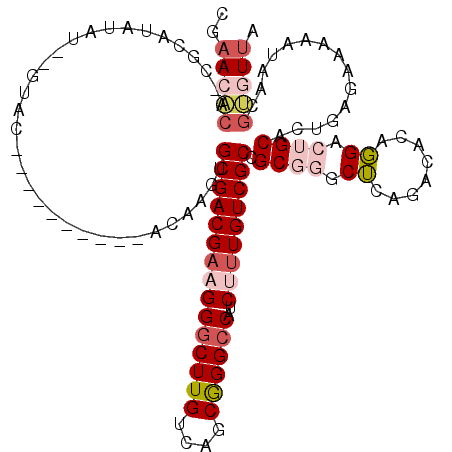
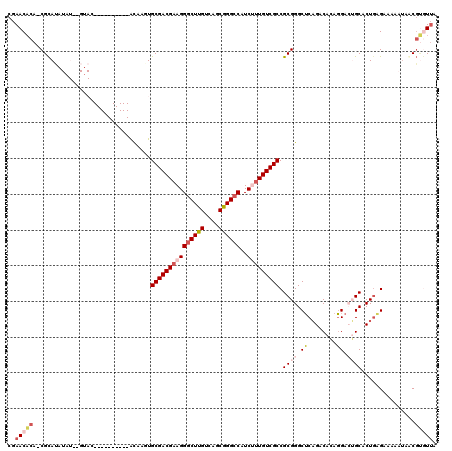
| Location | 15,326,178 – 15,326,294 |
|---|---|
| Length | 116 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 76.66 |
| Mean single sequence MFE | -41.00 |
| Consensus MFE | -21.67 |
| Energy contribution | -22.58 |
| Covariance contribution | 0.92 |
| Combinations/Pair | 1.10 |
| Mean z-score | -2.15 |
| Structure conservation index | 0.53 |
| SVM decision value | 1.07 |
| SVM RNA-class probability | 0.909757 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 15326178 116 + 20766785 GCGACGAAGGGCUUGUCAGCGGGCCAUCUUUGUCGCCGCGGGCUCAGACAGAGGAGUGCACUGAGGAAAAUAGCGUGUUAGUUAGGCAUAAGCUGCAUGCUAUAUGU----UUAAGCAAA (((((((((((((((....))))))..))))))))).(((.((((........)))).)..........(((((((((.((((.......)))))))))))))....----....))... ( -42.70) >DroSec_CAF1 132154 115 + 1 GCGACGAAGGGCUUGUCAGCGGGCCAUCUUUGUCGCCGCGGGCUCAGACAGAGGACUGCACUGGGAAAAAUAGCGUGUUAGUU-GGAAUAAGCUGCAUGCUGUAUGU----UUGAGCAAA (((((((((((((((....))))))..))))))))).....(((((((((.((.......)).......(((((((((.((((-......))))))))))))).)))----))))))... ( -48.70) >DroSim_CAF1 109916 115 + 1 GCGACGAAGGGCUUGUCAGCGGGCCAUCUUUGUCGCCGCGGGCUCAGACAGAGGACUGCACUGAGAAAAAUAGCGUGUUAGUU-GUAAUAAGCUUCAUGCUGUAUGU----UUGAGCAAA (((((((((((((((....))))))..))))))))).....(((((((((.((.......)).......((((((((..((((-......)))).)))))))).)))----))))))... ( -47.00) >DroEre_CAF1 118779 112 + 1 GCGACGAAGGACUUGUCAGCGGGCCAUCGUUGUCGCCGCGGGCUCAGACACAGGACUGCACUGAGCGAAAAAACGCCUUGGUU-AGCA--A-----GUCCUGUAUGUGCGACUUUGGAGC (((((((.((.((((....)))))).)))))))..(((.((.(.((.(.((((((((((((..((((......)).))..)).-.)).--)-----))))))).).)).).)).)))... ( -41.30) >DroYak_CAF1 122849 115 + 1 GCGACGAAGGGCUUGUCAGCGGGCCAUCGCUGUCGCCGCGGGCUCAGACACAGGACGGCACUAAGAAAAAUAACGUGUUAGUU-GGCAUAAGCUCUGUGCUGUAUGU----UUCAAGAGA ((((((...((((((....)))))).....))))))......(((.((.(((..(((((((..((.....((((......)))-).......))..))))))).)))----.))..))). ( -37.00) >DroAna_CAF1 105882 97 + 1 GCGACGAAGGGCUUGUCAACAGGCCAUCUUUGUCGCCGCGGGCACUGACACAUGCAGGCACUGAGAGAAAUUACUGAUCAUCU-AACGU-------UUACUAU---------UG------ (((((((((((((((....))))))..)))))))))(.(((((.(((.(....)))))).))).)..................-.....-------.......---------..------ ( -29.30) >consensus GCGACGAAGGGCUUGUCAGCGGGCCAUCUUUGUCGCCGCGGGCUCAGACACAGGACUGCACUGAGAAAAAUAACGUGUUAGUU_GGCAUAAGCU_CAUGCUGUAUGU____UUGAGCAAA (((((((((((((((....))))))..))))))))).((((.((........)).))))............................................................. (-21.67 = -22.58 + 0.92)

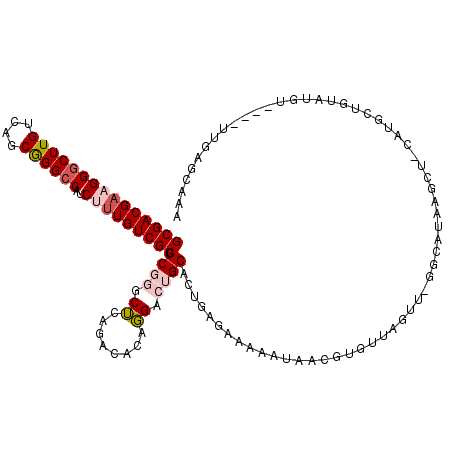
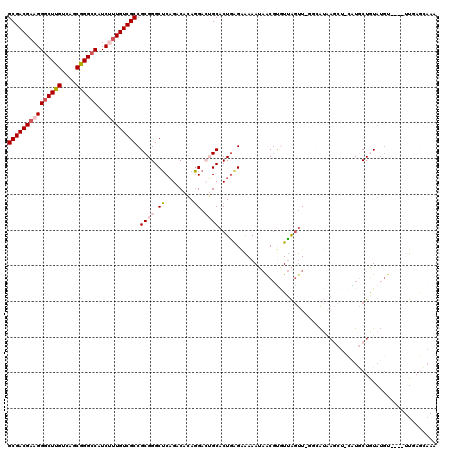
| Location | 15,326,218 – 15,326,316 |
|---|---|
| Length | 98 |
| Sequences | 4 |
| Columns | 116 |
| Reading direction | forward |
| Mean pairwise identity | 77.43 |
| Mean single sequence MFE | -26.26 |
| Consensus MFE | -18.09 |
| Energy contribution | -18.02 |
| Covariance contribution | -0.06 |
| Combinations/Pair | 1.31 |
| Mean z-score | -1.59 |
| Structure conservation index | 0.69 |
| SVM decision value | 0.20 |
| SVM RNA-class probability | 0.631444 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 15326218 98 + 20766785 GGCUCAGACAGAGGAGUGCACUGAGGAAAAUAGCGUGUUAGUUAGGCAUAAGCUGCAUGCUAUAUGUUUAAGCAAAUAUGUUUUUAAAAAAGUCCUCU------------------ .((((........)))).....(((((....((((((((.(((((((((((((.....))).))))))).))).))))))))..........))))).------------------ ( -25.14) >DroSec_CAF1 132194 96 + 1 GGCUCAGACAGAGGACUGCACUGGGAAAAAUAGCGUGUUAGUU-GGAAUAAGCUGCAUGCUGUAUGUUUGAGCAAAUAUGUUUUUAAAGAAGUCCUG------------------- .(((((((((.((.......)).......(((((((((.((((-......))))))))))))).)))))))))........................------------------- ( -27.30) >DroSim_CAF1 109956 96 + 1 GGCUCAGACAGAGGACUGCACUGAGAAAAAUAGCGUGUUAGUU-GUAAUAAGCUUCAUGCUGUAUGUUUGAGCAAAUAUGUUUUCAAAGAAGUUCUC------------------- ..........(((((((....((((((..((((((((..((((-......)))).))))))))((((((....)))))).))))))....)))))))------------------- ( -26.20) >DroYak_CAF1 122889 115 + 1 GGCUCAGACACAGGACGGCACUAAGAAAAAUAACGUGUUAGUU-GGCAUAAGCUCUGUGCUGUAUGUUUCAAGAGAUAAGUUUUAAGAGAUGUCCAGUUUGGUCGCUUGUCAAGUU (((.(((((...(((((((((..((.....((((......)))-).......))..)))))....(((((..((((....))))..))))))))).))))))))............ ( -26.40) >consensus GGCUCAGACAGAGGACUGCACUGAGAAAAAUAGCGUGUUAGUU_GGAAUAAGCUGCAUGCUGUAUGUUUGAGCAAAUAUGUUUUUAAAGAAGUCCUC___________________ ..........(((((((....((((((..(((((((((.((((.......)))))))))))))((((((....)))))).))))))....)))))))................... (-18.09 = -18.02 + -0.06)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:26:47 2006