
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 2,630,602 – 2,630,713 |
| Length | 111 |
| Max. P | 0.809807 |

| Location | 2,630,602 – 2,630,713 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 76.96 |
| Mean single sequence MFE | -43.77 |
| Consensus MFE | -20.22 |
| Energy contribution | -20.50 |
| Covariance contribution | 0.28 |
| Combinations/Pair | 1.32 |
| Mean z-score | -2.21 |
| Structure conservation index | 0.46 |
| SVM decision value | 0.64 |
| SVM RNA-class probability | 0.809807 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 2630602 111 + 20766785 CUGAACUCGGUG---GCCUCCCCGCUCCGCACCGCCAGCUACAAGUCGGCGGCGGCGGUC---GCUGGUCAUGGCUUCCA---CCACAGCCAUCACCAGCAGCUGGACUUCCAGCGGAAC ........((((---(.....)))))((((.(((((.((.....)).)))))..))))..---((((((.((((((....---....)))))).)))))).(((((....)))))..... ( -51.60) >DroVir_CAF1 76900 108 + 1 CUGAACUCGGUGGUGGCCACGCCGCUGCGCACCGCCAGCUACAAGACGGCGGCCACG------GCCA---GUGUCUAUCA---UCAUAGCCACCAUCAGCAGCUCGAUUUUCAGCGCAAC ........(((((((((......((((.((...))))))....(((((..(((....------))).---.)))))....---.....))))))))).((.(((.(.....))))))... ( -39.10) >DroGri_CAF1 69802 111 + 1 CUGAACUCGGUGGUGGCGACGCCGCUGCGCACCGCCAGCUACAAGACGGCGGCAUCG------GCCAGCAGCGUCUAUCA---UCAUAGCCACCAUCAGCAGCUGGAUUUUCAGCGCAAC ........((((((((((((((.((((.((.(((((..(.....)..))))).....------)))))).)))))(((..---..)))))))))))).((.(((((....)))))))... ( -50.70) >DroWil_CAF1 86122 117 + 1 CUGAACUCGGUG---GCCUCGCCAUUGCGGCAGGCCAGCUAUAAGUCAGCGGCCGCUGUCCAGGCCAGCCAUGGCUAUCAUAGUCAUAGUCACCAUCAGCAGUUGGACUUUCAGCGCAAC ........((((---((((.(((...(((((.((((.(((.......))))))))))))...))).))..(((((((...))))))).))))))....((.(((((....)))))))... ( -42.40) >DroMoj_CAF1 78575 108 + 1 CUGAACUCGGUGGUUGCCACGCCGCUGCGCACCGCUAGCUACAAGACGGCCGCAACC------GCCA---GUGUCUAUCA---UCAUAGCCACCAUCAGCAGCUGGACUUUCAGCGCAAU ((((....(((((((((...((((((..((.......))....)).)))).))))))------))).---(((.((((..---..)))).)))..))))..(((((....)))))..... ( -38.20) >DroAna_CAF1 64217 111 + 1 CUCAACUCUGUG---GCCACGCCCCUGCGCACCGCCAGCUACAAGACGGCAGCGGCAGUG---GCGGGGCACGGCUUCCA---CCACAGCCACCACCAGCAGCUGGACUUCCAAAGGAAC .(((.(((((((---(....(((((.((.(((.(((.(((.(......).)))))).)))---)))))))..((((....---....)))).))).))).)).)))..((((...)))). ( -40.60) >consensus CUGAACUCGGUG___GCCACGCCGCUGCGCACCGCCAGCUACAAGACGGCGGCAGCG______GCCAG_CAUGGCUAUCA___UCAUAGCCACCAUCAGCAGCUGGACUUUCAGCGCAAC ........((((.......))))..(((((....((((((...(((((..(((..........))).....)))))............((........)))))))).......))))).. (-20.22 = -20.50 + 0.28)

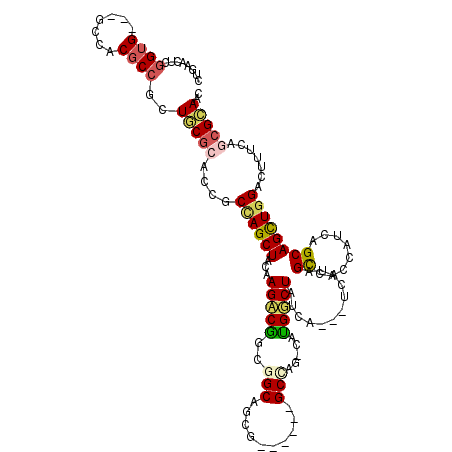
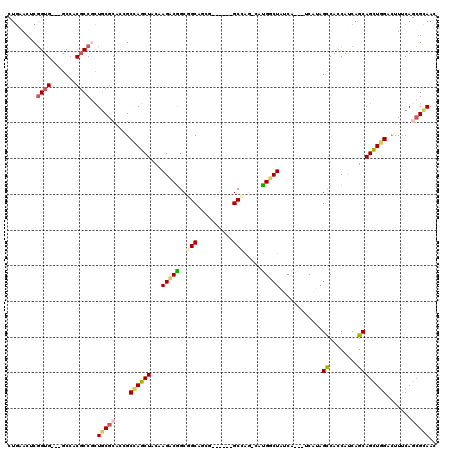
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:39:13 2006