
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 14,964,181 – 14,964,376 |
| Length | 195 |
| Max. P | 0.999847 |

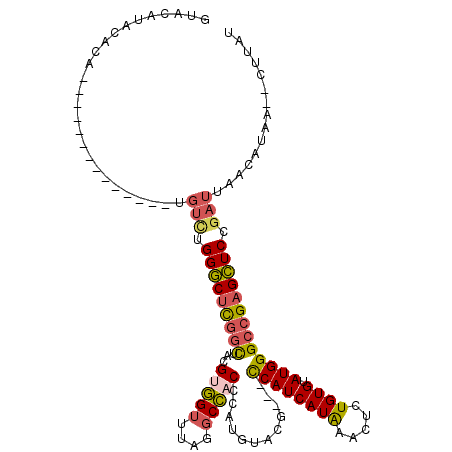
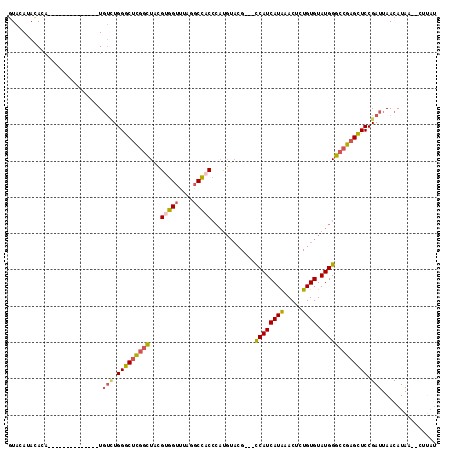
| Location | 14,964,181 – 14,964,280 |
|---|---|
| Length | 99 |
| Sequences | 6 |
| Columns | 118 |
| Reading direction | forward |
| Mean pairwise identity | 80.32 |
| Mean single sequence MFE | -31.37 |
| Consensus MFE | -25.15 |
| Energy contribution | -25.43 |
| Covariance contribution | 0.28 |
| Combinations/Pair | 1.28 |
| Mean z-score | -2.13 |
| Structure conservation index | 0.80 |
| SVM decision value | 1.18 |
| SVM RNA-class probability | 0.926505 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 14964181 99 + 20766785 GUACAUACACA--------------UGUCUGGGCUCGGCUACGUGGUUUAGGCCACCCAUGUACG---CCAUCAUAAACUCUGUGUAUGGGCCGAGCUCCGAUUAACAUAA--CUUAU ...........--------------.(((.((((((((((((((((..........)))))))..---((((((((.....)))).))))))))))))).)))........--..... ( -32.70) >DroSec_CAF1 4053 99 + 1 GUACAUACACA--------------UGUUUGGGCUCGGCUACGCGGUUUAGGCCACCCAUGUACG---CCAUCAUAAACUCUGUGUAUGGGCCGAGCUCCGAUUAACAUAA--CUUAU ..........(--------------((((((((((((((.....(((....)))...........---((((((((.....)))).))))))))))).))))...))))..--..... ( -29.60) >DroSim_CAF1 4297 95 + 1 GUACAUACACA--------------UGUCUGGGCUCGGCUACGCGGUUUAGGCCACCCAUGUACG---CCAUCAUAAACUCUGUGUAUGGGCCGAGCUCCGAUUAACAUAA--C---- ...........--------------.(((.(((((((((.....(((....)))...........---((((((((.....)))).))))))))))))).)))........--.---- ( -29.20) >DroEre_CAF1 4159 99 + 1 GUACAUACAUA--------------UGUCUGGGCUCGGCUAUGUGGUUUAGGCCACCCAUGUACG---CCAUCAUAAACUCUGUGUAUGGGCCGAGCUCCGAUUAACAUAA--CUUAU ...........--------------.(((.(((((((((...(((((....))))).........---((((((((.....)))).))))))))))))).)))........--..... ( -32.30) >DroYak_CAF1 4346 116 + 1 GUACAUAUAUACGUAUGUCAUGUCAUGUCUGGGCUCGGCUGUGUGGUUUUGGCCACCCAUGUACGCCGCCAUCAUAAACUCUGUGUAUGGGCCGAGCUCCGAUUAACAUAA--CUUAU (.((((((....)))))))((((...(((.(((((((((((.(((((....))))).)).........((((((((.....)))).))))))))))))).)))..))))..--..... ( -39.50) >DroAna_CAF1 3592 91 + 1 -UACAUACAUG--------------UCCAUGGACCUAUUUGUGUCGGCGGGGCGAC-------CG---UCAUCAUGAAC--UGUGUAUGGGCCGAGUUCCGAUUAACAUAAGUCUCAU -.........(--------------((((((.((...((..((..(((((.....)-------))---))..))..)).--.)).)))))))........((((......)))).... ( -24.90) >consensus GUACAUACACA______________UGUCUGGGCUCGGCUACGUGGUUUAGGCCACCCAUGUACG___CCAUCAUAAACUCUGUGUAUGGGCCGAGCUCCGAUUAACAUAA__CUUAU ..........................(((.(((((((((...(((((....)))))............((((((((.....)))).))))))))))))).)))............... (-25.15 = -25.43 + 0.28)

| Location | 14,964,181 – 14,964,280 |
|---|---|
| Length | 99 |
| Sequences | 6 |
| Columns | 118 |
| Reading direction | reverse |
| Mean pairwise identity | 80.32 |
| Mean single sequence MFE | -28.77 |
| Consensus MFE | -21.35 |
| Energy contribution | -20.97 |
| Covariance contribution | -0.39 |
| Combinations/Pair | 1.26 |
| Mean z-score | -1.11 |
| Structure conservation index | 0.74 |
| SVM decision value | 0.03 |
| SVM RNA-class probability | 0.546270 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 14964181 99 - 20766785 AUAAG--UUAUGUUAAUCGGAGCUCGGCCCAUACACAGAGUUUAUGAUGG---CGUACAUGGGUGGCCUAAACCACGUAGCCGAGCCCAGACA--------------UGUGUAUGUAC (((..--.((((((....((.(((((((((((.......(((......))---)....))))((((......))))...))))))))).))))--------------))..))).... ( -28.40) >DroSec_CAF1 4053 99 - 1 AUAAG--UUAUGUUAAUCGGAGCUCGGCCCAUACACAGAGUUUAUGAUGG---CGUACAUGGGUGGCCUAAACCGCGUAGCCGAGCCCAAACA--------------UGUGUAUGUAC (((..--.((((((....((.(((((((((((.......(((......))---)....))))((((......))))...))))))))).))))--------------))..))).... ( -27.60) >DroSim_CAF1 4297 95 - 1 ----G--UUAUGUUAAUCGGAGCUCGGCCCAUACACAGAGUUUAUGAUGG---CGUACAUGGGUGGCCUAAACCGCGUAGCCGAGCCCAGACA--------------UGUGUAUGUAC ----.--.((((((....((.(((((((((((.......(((......))---)....))))((((......))))...))))))))).))))--------------))......... ( -27.60) >DroEre_CAF1 4159 99 - 1 AUAAG--UUAUGUUAAUCGGAGCUCGGCCCAUACACAGAGUUUAUGAUGG---CGUACAUGGGUGGCCUAAACCACAUAGCCGAGCCCAGACA--------------UAUGUAUGUAC (((..--.((((((....((.(((((((((((.......(((......))---)....))))((((......))))...))))))))).))))--------------))..))).... ( -28.70) >DroYak_CAF1 4346 116 - 1 AUAAG--UUAUGUUAAUCGGAGCUCGGCCCAUACACAGAGUUUAUGAUGGCGGCGUACAUGGGUGGCCAAAACCACACAGCCGAGCCCAGACAUGACAUGACAUACGUAUAUAUGUAC ....(--((((((((.((((.(((((((((((..((...(((......)))...))..))))((((......))))...))))))))).))..))))))))).(((((....))))). ( -39.20) >DroAna_CAF1 3592 91 - 1 AUGAGACUUAUGUUAAUCGGAACUCGGCCCAUACACA--GUUCAUGAUGA---CG-------GUCGCCCCGCCGACACAAAUAGGUCCAUGGA--------------CAUGUAUGUA- (((.(((((((.....((((....((((((((.((..--.....))))).---.)-------)))).....)))).....))))))))))...--------------..........- ( -21.10) >consensus AUAAG__UUAUGUUAAUCGGAGCUCGGCCCAUACACAGAGUUUAUGAUGG___CGUACAUGGGUGGCCUAAACCACAUAGCCGAGCCCAGACA______________UAUGUAUGUAC ..................((.(((((((((((.((.........))))))............((((......))))...))))))))).............................. (-21.35 = -20.97 + -0.39)
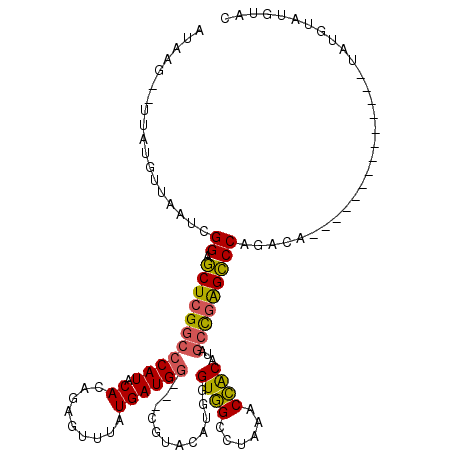
| Location | 14,964,280 – 14,964,376 |
|---|---|
| Length | 96 |
| Sequences | 5 |
| Columns | 103 |
| Reading direction | reverse |
| Mean pairwise identity | 91.50 |
| Mean single sequence MFE | -35.78 |
| Consensus MFE | -32.98 |
| Energy contribution | -33.46 |
| Covariance contribution | 0.48 |
| Combinations/Pair | 1.08 |
| Mean z-score | -2.57 |
| Structure conservation index | 0.92 |
| SVM decision value | 4.24 |
| SVM RNA-class probability | 0.999847 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 14964280 96 - 20766785 GAUCC-----GAAACGAAAACCCACGCCAGAUGACGUUAUCGUUGGUGUGGCGAUGG--UGCUGCUGGUCGUGGUGGUGGAAACGCACCAAAAAUGGCAGCGA .....-----...........((((((((.((((((....)))).)).))))).)))--((((((((....(((((.((....)))))))....)))))))). ( -38.20) >DroSec_CAF1 4152 96 - 1 GAUCC-----GAAACGAAAACCCACGCCAGAUGACGUUAUCGUUGGUGUGGCGAUGG--UGCUGCUGGUCGUGGUGGUGGAAACGCACCAAAAAUGGCAGCGA .....-----...........((((((((.((((((....)))).)).))))).)))--((((((((....(((((.((....)))))))....)))))))). ( -38.20) >DroSim_CAF1 4392 91 - 1 GAUCC-----GAAACGAAAACCCACGCCAGAUGACGUUAUCGUUGGUGUGGCGAUGG--UGCUGCUGGUCGUGGUGGUGGAAACGCACCAAAAAUGGU----- .....-----..........((((((((((.(..((..((((((.....))))))..--))..))))).))))).)(((....)))((((....))))----- ( -30.90) >DroEre_CAF1 4258 98 - 1 GAUCC-----GAAACGAAAACCCACGCCAGAUGACGUUAUCGUUGGUGUGGCGAUGGUGUGCUGCUGGUCGUGGUGGUGGAAACGCACCAAAAAUGGCAGCGA .....-----.........((((((((((.((((((....)))).)).))))).))).))(((((((....(((((.((....)))))))....))))))).. ( -39.30) >DroYak_CAF1 4462 95 - 1 GAUCCAAAACGAAACGAAAACCCACGCCAGAUGACGUUAUCGUUGGUGUGC--------UGCUGUUGGUCGUGGUGGUGGAAACGCACCAAAAAUGGCAGCGA .......................((((((.((((.....))))))))))((--------(((((((.....(((((.((....)))))))..))))))))).. ( -32.30) >consensus GAUCC_____GAAACGAAAACCCACGCCAGAUGACGUUAUCGUUGGUGUGGCGAUGG__UGCUGCUGGUCGUGGUGGUGGAAACGCACCAAAAAUGGCAGCGA .....................((((((((.((((.....)))))))))))).........(((((((....(((((.((....)))))))....))))))).. (-32.98 = -33.46 + 0.48)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:23:40 2006