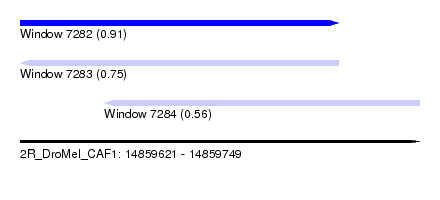
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 14,859,621 – 14,859,749 |
| Length | 128 |
| Max. P | 0.910535 |

| Location | 14,859,621 – 14,859,723 |
|---|---|
| Length | 102 |
| Sequences | 6 |
| Columns | 116 |
| Reading direction | forward |
| Mean pairwise identity | 70.91 |
| Mean single sequence MFE | -22.42 |
| Consensus MFE | -13.72 |
| Energy contribution | -14.36 |
| Covariance contribution | 0.64 |
| Combinations/Pair | 1.36 |
| Mean z-score | -1.57 |
| Structure conservation index | 0.61 |
| SVM decision value | 1.07 |
| SVM RNA-class probability | 0.910535 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
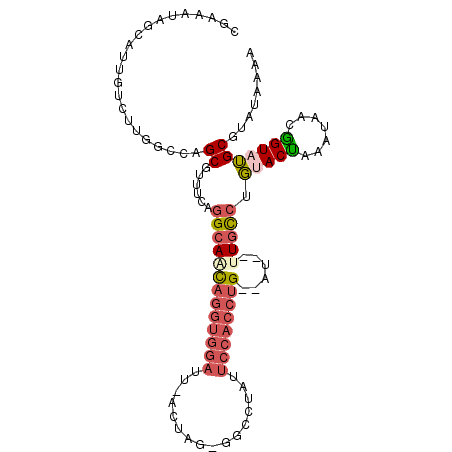
>2R_DroMel_CAF1 14859621 102 + 20766785 ----AAUCUC---UCUAGCCCUCGAUAUCC--UUGUUUUUAUACGCAUACCGUUAUUUAGUACAGGCAA--UU--CAGGUGGAAUAGGCU-CUGGAAAAUCCACCUGUUGCCUGAA ----......---.((((....((.(((.(--.(((....))).).))).))....))))..(((((((--..--((((((((.....(.-...)....))))))))))))))).. ( -24.90) >DroSec_CAF1 31881 101 + 1 ----AUUCUC---UCUAUCCCUCGAUAUCC--UUGUUUUUAUACGCAUACCGUUAUUUAGUACAGGCAA--AU--CAGGUGGAAUAGGCC-CUAGU-AAUCCACCUGUUGCCUGAA ----......---.(((.....((.(((.(--.(((....))).).))).)).....)))..(((((((--..--((((((((....((.-...))-..))))))))))))))).. ( -25.70) >DroSim_CAF1 33482 101 + 1 ----AUUCUC---UCUAUCCCUCGAUAUCC--UUGUUUUUAUAUGCAUACCGUUAUUUAGUGCAGGCAA--AU--CAGGUGGAAUAGGCC-CUAGU-AAUCCACCUGUUGCCUGAA ----.....(---.(((.....((.(((.(--.(((....))).).))).)).....))).)(((((((--..--((((((((....((.-...))-..))))))))))))))).. ( -25.50) >DroEre_CAF1 31501 108 + 1 ----UUUCUC-AAUCUAUUCCUCGAUAUACAAUCUAUUUUAUACGCAUACAAUUGUUUCGUACGGGCAA--GUGCCAGGUGGAGUAGGUCGCCGGG-AAUCCACCUGAUGCCUGAA ----......-..............................((((...((....))..))))((((((.--....((((((((...((...))...-..)))))))).)))))).. ( -24.80) >DroYak_CAF1 32950 98 + 1 ----UGUCUCUAAAAUAUUCCUCGAUAUCC--UUGUUUUUAUACGCAUACAAUUGUUUUGUACGGGCAAAAAUAGCAGGUGGAA-----------A-AAUCCACCUGAUGCCUGAA ----(((....((((((.............--.)))))).....)))(((((.....)))))((((((.......((((((((.-----------.-..)))))))).)))))).. ( -23.94) >DroAna_CAF1 35046 92 + 1 AAACAAUC-C---U-CAAUCCUCGAUAAUC--UUAUCUUUAUACGCCAACAGUUAUUUCGUAGUGAUAA--G---CAGG----AUA---U-CUA----AUACCAUAACUGCUUCAG ........-.---.-........((((...--.))))............(((((((...(((..((((.--.---....----.))---)-)..----.))).)))))))...... ( -9.70) >consensus ____AUUCUC___UCUAUCCCUCGAUAUCC__UUGUUUUUAUACGCAUACAGUUAUUUAGUACAGGCAA__AU__CAGGUGGAAUAGGCC_CUAGA_AAUCCACCUGUUGCCUGAA ..............................................................((((((.......((((((((................)))))))).)))))).. (-13.72 = -14.36 + 0.64)

| Location | 14,859,621 – 14,859,723 |
|---|---|
| Length | 102 |
| Sequences | 6 |
| Columns | 116 |
| Reading direction | reverse |
| Mean pairwise identity | 70.91 |
| Mean single sequence MFE | -25.79 |
| Consensus MFE | -12.47 |
| Energy contribution | -13.47 |
| Covariance contribution | 1.00 |
| Combinations/Pair | 1.33 |
| Mean z-score | -1.71 |
| Structure conservation index | 0.48 |
| SVM decision value | 0.47 |
| SVM RNA-class probability | 0.750249 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 14859621 102 - 20766785 UUCAGGCAACAGGUGGAUUUUCCAG-AGCCUAUUCCACCUG--AA--UUGCCUGUACUAAAUAACGGUAUGCGUAUAAAAACAA--GGAUAUCGAGGGCUAGA---GAGAUU---- ..(((((((((((((((........-.......))))))))--..--)))))))..(((..(..((((((.(............--).))))))..)..))).---......---- ( -29.66) >DroSec_CAF1 31881 101 - 1 UUCAGGCAACAGGUGGAUU-ACUAG-GGCCUAUUCCACCUG--AU--UUGCCUGUACUAAAUAACGGUAUGCGUAUAAAAACAA--GGAUAUCGAGGGAUAGA---GAGAAU---- ..(((((((((((((((.(-(....-....)).))))))))--..--)))))))..(((..(..((((((.(............--).))))))..)..))).---......---- ( -30.10) >DroSim_CAF1 33482 101 - 1 UUCAGGCAACAGGUGGAUU-ACUAG-GGCCUAUUCCACCUG--AU--UUGCCUGCACUAAAUAACGGUAUGCAUAUAAAAACAA--GGAUAUCGAGGGAUAGA---GAGAAU---- ..(((((((((((((((.(-(....-....)).))))))))--..--)))))))..(((..(..((((((.(............--).))))))..)..))).---......---- ( -29.20) >DroEre_CAF1 31501 108 - 1 UUCAGGCAUCAGGUGGAUU-CCCGGCGACCUACUCCACCUGGCAC--UUGCCCGUACGAAACAAUUGUAUGCGUAUAAAAUAGAUUGUAUAUCGAGGAAUAGAUU-GAGAAA---- (((.(((((((((((((..-...(....)....)))))))))...--.))))(((((((.....))))))).((((((......)))))).((((........))-))))).---- ( -26.00) >DroYak_CAF1 32950 98 - 1 UUCAGGCAUCAGGUGGAUU-U-----------UUCCACCUGCUAUUUUUGCCCGUACAAAACAAUUGUAUGCGUAUAAAAACAA--GGAUAUCGAGGAAUAUUUUAGAGACA---- (((.((((.((((((((..-.-----------.)))))))).......))))(((((((.....)))))))............(--((((((......))))))).)))...---- ( -23.60) >DroAna_CAF1 35046 92 - 1 CUGAAGCAGUUAUGGUAU----UAG-A---UAU----CCUG---C--UUAUCACUACGAAAUAACUGUUGGCGUAUAAAGAUAA--GAUUAUCGAGGAUUG-A---G-GAUUGUUU ...((((((((.....((----(.(-(---((.----....---(--(((((..((((.(((....)))..))))....)))))--)..))))...)))..-.---.-)))))))) ( -16.20) >consensus UUCAGGCAACAGGUGGAUU_ACUAG_GGCCUAUUCCACCUG__AU__UUGCCUGUACUAAAUAACGGUAUGCGUAUAAAAACAA__GGAUAUCGAGGAAUAGA___GAGAAU____ (((.(((((((((((((................))))))))......))))).(((((.......))))).......................))).................... (-12.47 = -13.47 + 1.00)


| Location | 14,859,648 – 14,859,749 |
|---|---|
| Length | 101 |
| Sequences | 6 |
| Columns | 106 |
| Reading direction | reverse |
| Mean pairwise identity | 74.77 |
| Mean single sequence MFE | -26.76 |
| Consensus MFE | -14.60 |
| Energy contribution | -15.57 |
| Covariance contribution | 0.97 |
| Combinations/Pair | 1.30 |
| Mean z-score | -1.54 |
| Structure conservation index | 0.55 |
| SVM decision value | 0.05 |
| SVM RNA-class probability | 0.558260 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 14859648 101 - 20766785 CGAAAUAGCAUUGUCUUGGCCAGCGUUUCAGGCAACAGGUGGAUUUUCCAG-AGCCUAUUCCACCUG--AA--UUGCCUGUACUAAAUAACGGUAUGCGUAUAAAA .......((((.((....))...((((.(((((((((((((((........-.......))))))))--..--)))))))........))))..))))........ ( -27.66) >DroSec_CAF1 31908 100 - 1 CGAAAUAGCUUUGUCUUGGCCAGCGUUUCAGGCAACAGGUGGAUU-ACUAG-GGCCUAUUCCACCUG--AU--UUGCCUGUACUAAAUAACGGUAUGCGUAUAAAA .(((((.(((..((....)).))))))))((((((((((((((.(-(....-....)).))))))))--..--))))))(((((.......))))).......... ( -29.30) >DroSim_CAF1 33509 100 - 1 CGAAAUAGCAUUGUCUUGGCCAGCGUUUCAGGCAACAGGUGGAUU-ACUAG-GGCCUAUUCCACCUG--AU--UUGCCUGCACUAAAUAACGGUAUGCAUAUAAAA .......((((.......(((...(((((((((((((((((((.(-(....-....)).))))))))--..--)))))))....))))...)))))))........ ( -27.91) >DroEre_CAF1 31532 103 - 1 CGAAAUCCCAUUGACUUGGCCAGCGUUUCAGGCAUCAGGUGGAUU-CCCGGCGACCUACUCCACCUGGCAC--UUGCCCGUACGAAACAAUUGUAUGCGUAUAAAA .(((((.(((......))).....))))).(((((((((((((..-...(....)....)))))))))...--.))))(((((((.....)))))))......... ( -27.30) >DroYak_CAF1 32980 94 - 1 CGAAAUCCCAUUAUCUUGGCCAGCGUUUCAGGCAUCAGGUGGAUU-U-----------UUCCACCUGCUAUUUUUGCCCGUACAAAACAAUUGUAUGCGUAUAAAA .(((((.(((......))).....))))).((((.((((((((..-.-----------.)))))))).......))))(((((((.....)))))))......... ( -26.20) >DroAna_CAF1 35075 89 - 1 CGACGGGUCGGCGUCUUGGCCAGCGUCUGAAGCAGUUAUGGUAU----UAG-A---UAU----CCUG---C--UUAUCACUACGAAAUAACUGUUGGCGUAUAAAG .....((((((....)))))).(((((...(((((((((.(((.----..(-(---((.----....---.--.))))..)))...))))))))))))))...... ( -22.20) >consensus CGAAAUAGCAUUGUCUUGGCCAGCGUUUCAGGCAACAGGUGGAUU_ACUAG_GGCCUAUUCCACCUG__AU__UUGCCUGUACUAAAUAACGGUAUGCGUAUAAAA ......................((......(((((((((((((................))))))))......))))).(((((.......)))))))........ (-14.60 = -15.57 + 0.97)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:22:41 2006