
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 14,818,378 – 14,818,538 |
| Length | 160 |
| Max. P | 0.999921 |

| Location | 14,818,378 – 14,818,498 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 93.65 |
| Mean single sequence MFE | -52.76 |
| Consensus MFE | -49.32 |
| Energy contribution | -50.12 |
| Covariance contribution | 0.80 |
| Combinations/Pair | 1.07 |
| Mean z-score | -2.58 |
| Structure conservation index | 0.93 |
| SVM decision value | 4.56 |
| SVM RNA-class probability | 0.999921 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
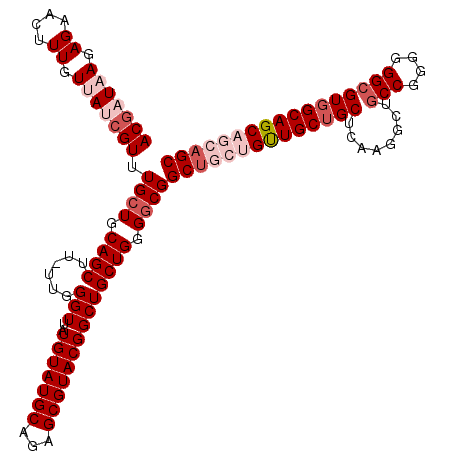
>2R_DroMel_CAF1 14818378 120 + 20766785 GGGUAUGAUACGCAUUGUUCCCAGUGGAGGCGGGGCCGCUGCUGCUGCUGCCACGCCACCCGGCAGCAUUGAGCAGCAACAGCAGCCGUCCCAGCAGCCGUACGCUCUGCAUACAUAACC ..(((((....((((((....))))((..(((((((.(((((((.((((((((.(((....))).....)).)))))).))))))).))))).))..))....))....)))))...... ( -52.60) >DroSec_CAF1 20265 120 + 1 GGGUAUGAUACGCAUUGUUCCCAGCGGAAGCGGGGCCGCUGCUGCUGCUGCGACGCCCCCCGGCAGCCUUGAGCAGCAACAGCAGCCGCCCCAGCAGCCGUACGCUCUGCAUACAUAACC ((.((((.((.(((..((.....((((..(((((((.(((((((.((((((...(((....)))........)))))).))))))).))))).))..))))..))..))).)))))).)) ( -55.20) >DroSim_CAF1 14501 120 + 1 GGGUAUGAUACGCAUUGUUCCCAGCGGAAGCGGGGCCGCUGCUGCUGCUGCCACGCCCCCCGGCAGCCUUGAGCAGCAACAGCAGCCGCCCCAGCAGCCGUACGCUCUGCAUACAUAACC ((.((((.((.(((..((.....((((..(((((((.(((((((.((((((((.(((....))).....)).)))))).))))))).))))).))..))))..))..))).)))))).)) ( -56.10) >DroEre_CAF1 13188 117 + 1 GGGUAUGAUACGCAUUGUUCCCAGCGGAGGCGGGGCCGCUGCA---GCUGCCACUCCCCCAGGGACCCUUGAGCAGCAACAGCAGCCGCCCCAGCAGCCGUACGCUCUGCAUACAUAACC ((.((((.((.(((..((.....((((..(((((((.(((((.---(((((((.((((...))))....)).)))))....))))).))))).))..))))..))..))).)))))).)) ( -50.00) >DroYak_CAF1 14452 117 + 1 GGGUAUGAUACGCAUUGUUCCCAGCGGAGGCGGGUCCGCUGCU---GCUGCCACGCCCCCCGGCACCAUUGAGCAGCAGCAGCAGCCGCCCCAGCAGCCGUACGCUCUGCAUACAUAACC (((.(..((....))..).))).((((.(((((.(..((((((---(((((((.(((....))).....)).)))))))))))).)))))))((.(((.....))))))).......... ( -49.90) >consensus GGGUAUGAUACGCAUUGUUCCCAGCGGAGGCGGGGCCGCUGCUGCUGCUGCCACGCCCCCCGGCAGCCUUGAGCAGCAACAGCAGCCGCCCCAGCAGCCGUACGCUCUGCAUACAUAACC ((.((((.((.(((..((.....((((..(((((((.(((((((.((((((((.(((....))).....)).)))))).))))))).))))).))..))))..))..))).)))))).)) (-49.32 = -50.12 + 0.80)


| Location | 14,818,378 – 14,818,498 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 93.65 |
| Mean single sequence MFE | -64.20 |
| Consensus MFE | -56.22 |
| Energy contribution | -58.62 |
| Covariance contribution | 2.40 |
| Combinations/Pair | 1.00 |
| Mean z-score | -3.53 |
| Structure conservation index | 0.88 |
| SVM decision value | 4.40 |
| SVM RNA-class probability | 0.999888 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 14818378 120 - 20766785 GGUUAUGUAUGCAGAGCGUACGGCUGCUGGGACGGCUGCUGUUGCUGCUCAAUGCUGCCGGGUGGCGUGGCAGCAGCAGCAGCGGCCCCGCCUCCACUGGGAACAAUGCGUAUCAUACCC (((.(((((((((........((..((.(((.(.(((((((((((((((..((((..(...)..))))))))))))))))))).)))).))..))..((....)).)))))).)))))). ( -61.10) >DroSec_CAF1 20265 120 - 1 GGUUAUGUAUGCAGAGCGUACGGCUGCUGGGGCGGCUGCUGUUGCUGCUCAAGGCUGCCGGGGGGCGUCGCAGCAGCAGCAGCGGCCCCGCUUCCGCUGGGAACAAUGCGUAUCAUACCC (((.(((((((((...(.(((((..((.(((((.((((((((((((((....(((.(((....)))))))))))))))))))).)))))))..))).)).).....)))))).)))))). ( -68.80) >DroSim_CAF1 14501 120 - 1 GGUUAUGUAUGCAGAGCGUACGGCUGCUGGGGCGGCUGCUGUUGCUGCUCAAGGCUGCCGGGGGGCGUGGCAGCAGCAGCAGCGGCCCCGCUUCCGCUGGGAACAAUGCGUAUCAUACCC (((.(((((((((...(.(((((..((.(((((.(((((((((((((((....((.(((....)))))))))))))))))))).)))))))..))).)).).....)))))).)))))). ( -68.40) >DroEre_CAF1 13188 117 - 1 GGUUAUGUAUGCAGAGCGUACGGCUGCUGGGGCGGCUGCUGUUGCUGCUCAAGGGUCCCUGGGGGAGUGGCAGC---UGCAGCGGCCCCGCCUCCGCUGGGAACAAUGCGUAUCAUACCC (((.(((((((((.((((...(((....(((((.(((((.(((((..(((.(((...)))....)))..)))))---.))))).))))))))..))))(....)..)))))).)))))). ( -61.00) >DroYak_CAF1 14452 117 - 1 GGUUAUGUAUGCAGAGCGUACGGCUGCUGGGGCGGCUGCUGCUGCUGCUCAAUGGUGCCGGGGGGCGUGGCAGC---AGCAGCGGACCCGCCUCCGCUGGGAACAAUGCGUAUCAUACCC (((.(((((((((.(((.....)))((.((((((((((((((((((((.((....((((....)))))))))))---))))))))..))))))).))((....)).)))))).)))))). ( -61.70) >consensus GGUUAUGUAUGCAGAGCGUACGGCUGCUGGGGCGGCUGCUGUUGCUGCUCAAGGCUGCCGGGGGGCGUGGCAGCAGCAGCAGCGGCCCCGCCUCCGCUGGGAACAAUGCGUAUCAUACCC (((.(((((((((...(.(((((..((.(((((.((((((((((((((.((....((((....)))))))))))))))))))).)))))))..))).)).).....)))))).)))))). (-56.22 = -58.62 + 2.40)


| Location | 14,818,418 – 14,818,538 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 92.44 |
| Mean single sequence MFE | -47.54 |
| Consensus MFE | -37.28 |
| Energy contribution | -40.12 |
| Covariance contribution | 2.84 |
| Combinations/Pair | 1.02 |
| Mean z-score | -1.86 |
| Structure conservation index | 0.78 |
| SVM decision value | 0.73 |
| SVM RNA-class probability | 0.834166 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 14818418 120 - 20766785 ACGAUAAGAGAACUUUGUUAUCGUUUGGUGCAGCUUUUUGGGUUAUGUAUGCAGAGCGUACGGCUGCUGGGACGGCUGCUGUUGCUGCUCAAUGCUGCCGGGUGGCGUGGCAGCAGCAGC (((((((.((....)).))))))).....((((((.(((.(((..(((((((...)))))))...))).))).))))))((((((((((..((((..(...)..)))))))))))))).. ( -50.40) >DroSec_CAF1 20305 119 - 1 ACGAUAAGAGAACUUUGUUAUCGUUUGCUGCAGCUG-UUGGGUUAUGUAUGCAGAGCGUACGGCUGCUGGGGCGGCUGCUGUUGCUGCUCAAGGCUGCCGGGGGGCGUCGCAGCAGCAGC (((((((.((....)).)))))))..(((((.((((-(.(.(((..((((((...))))))(((.(((.(((((((.......)))))))..))).)))....))).).))))).))))) ( -50.90) >DroSim_CAF1 14541 119 - 1 ACGAUAAGAGAACUUUGUUAUCGUUUGCUGCAGCUU-UUGGGUUAUGUAUGCAGAGCGUACGGCUGCUGGGGCGGCUGCUGUUGCUGCUCAAGGCUGCCGGGGGGCGUGGCAGCAGCAGC (((((((.((....)).))))))).((((.((((..-...(((..(((((((...)))))))))))))).))))((((((((((((((((..((...))...))))..)))))))))))) ( -50.40) >DroEre_CAF1 13228 116 - 1 ACGUUAAGAGAACUUUGUAAUCGUUUGCUGCAGCUU-UUGGGUUAUGUAUGCAGAGCGUACGGCUGCUGGGGCGGCUGCUGUUGCUGCUCAAGGGUCCCUGGGGGAGUGGCAGC---UGC ...(((((((......((((....)))).....)))-)))).....((((((...))))))((((((....))))))(((((..((.(((.(((...))).))).))..)))))---... ( -40.80) >DroYak_CAF1 14492 116 - 1 ACAUUAAGAGAACUUUGUAAUCGUUUGCUGCAGCUU-UUGGGUUAUGUAUGCAGAGCGUACGGCUGCUGGGGCGGCUGCUGCUGCUGCUCAAUGGUGCCGGGGGGCGUGGCAGC---AGC .........................((((.((((..-...(((..(((((((...)))))))))))))).))))((((((((..(.((((..((....))..)))))..)))))---))) ( -45.20) >consensus ACGAUAAGAGAACUUUGUUAUCGUUUGCUGCAGCUU_UUGGGUUAUGUAUGCAGAGCGUACGGCUGCUGGGGCGGCUGCUGUUGCUGCUCAAGGCUGCCGGGGGGCGUGGCAGCAGCAGC (((((((.((....)).))))))).((((.((((......(((..(((((((...)))))))))))))).))))((((((((((((((........(((....))))))))))))))))) (-37.28 = -40.12 + 2.84)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:22:17 2006