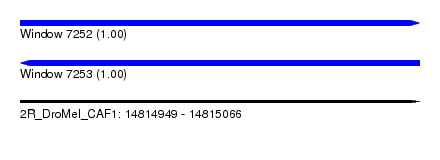
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 14,814,949 – 14,815,066 |
| Length | 117 |
| Max. P | 0.999844 |

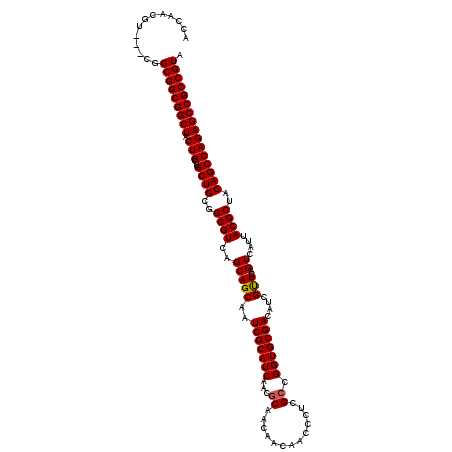
| Location | 14,814,949 – 14,815,066 |
|---|---|
| Length | 117 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 96.11 |
| Mean single sequence MFE | -44.90 |
| Consensus MFE | -43.44 |
| Energy contribution | -43.40 |
| Covariance contribution | -0.04 |
| Combinations/Pair | 1.03 |
| Mean z-score | -2.35 |
| Structure conservation index | 0.97 |
| SVM decision value | 4.23 |
| SVM RNA-class probability | 0.999844 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 14814949 117 + 20766785 ACCAACGU---CGGCGGCGGCUACUGCUGCUGCGGCGUCAGCAGCAAUCGCAUCAACGCAACAACAACCCUCGCCGGUGCGACAUCGUUCAGUCAUAACGCUACAGCCAGAGCCGCCGUA ........---..(((((((((.(((..((((..((((...........(((((...((.............)).)))))(((........)))...))))..)))))))))))))))). ( -44.22) >DroSec_CAF1 16796 117 + 1 ACCAACAU---CGGCGGCGGCUACUGCUGCUGCGGCGUCAGCAGCAAUCGCAUCAACCCAACAACAACCCUCGCCGGUGCGACAUCGUUCAGUCAUUACGCUACAGCCAGAGCCGCCGUA ........---..(((((((((.(((..((((..((((..((.((....))...............(((......)))))(((........)))...))))..)))))))))))))))). ( -43.40) >DroSim_CAF1 11069 117 + 1 ACCAACAU---CGGCGGCGGCUACUGCUGCUGCGGCGUCAGCAGCAAUCGCAUCAACGCAACAACAACCCUCGCCGGUGCGACAUCGUUCAGUCAUUACGCUACAGCCAGAGCCGCCGUA ........---..(((((((((.(((..((((..((((...........(((((...((.............)).)))))(((........)))...))))..)))))))))))))))). ( -44.12) >DroEre_CAF1 9673 117 + 1 ACCAACGU---CGGCGGCGGCUACUGGUGCUGCGGCGUCAGCAGCAAUCGCAUCAACGCAACAACAACCCUCGCCGGUGCGACAUCGCUCAGUCGUUACGCUACAGCCAGAGCCGCCGUU ........---.((((((((((.(((..((((..((((.((((((..(((((((...((.............)).)))))))....))).....)))))))..))))))))))))))))) ( -46.82) >DroYak_CAF1 10943 120 + 1 ACCAACGUCGUCGGCGGCGGCUACUGGUGCUGCGGCGUCAGCAGCAAUCGCAUCAACGCAACAACAACCCUCGCCGGUGCGACAUCGCUCAGUCAUUACGCUACAGCCAGAGCCGCCGUA .....((....))(((((((((.((((((((((.......)))))..(((((((...((.............)).))))))).......................)))))))))))))). ( -45.92) >consensus ACCAACGU___CGGCGGCGGCUACUGCUGCUGCGGCGUCAGCAGCAAUCGCAUCAACGCAACAACAACCCUCGCCGGUGCGACAUCGUUCAGUCAUUACGCUACAGCCAGAGCCGCCGUA .............(((((((((.(((..((((..((((..(((((..(((((((...((.............)).)))))))....)))..))....))))..)))))))))))))))). (-43.44 = -43.40 + -0.04)

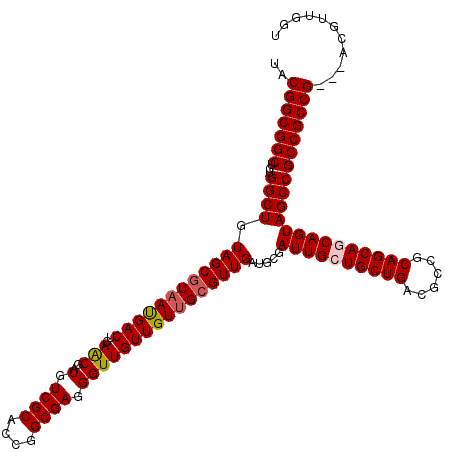
| Location | 14,814,949 – 14,815,066 |
|---|---|
| Length | 117 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 96.11 |
| Mean single sequence MFE | -54.18 |
| Consensus MFE | -49.56 |
| Energy contribution | -49.96 |
| Covariance contribution | 0.40 |
| Combinations/Pair | 1.05 |
| Mean z-score | -2.09 |
| Structure conservation index | 0.91 |
| SVM decision value | 2.93 |
| SVM RNA-class probability | 0.997771 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 14814949 117 - 20766785 UACGGCGGCUCUGGCUGUAGCGUUAUGACUGAACGAUGUCGCACCGGCGAGGGUUGUUGUUGCGUUGAUGCGAUUGCUGCUGACGCCGCAGCAGCAGUAGCCGCCGCCG---ACGUUGGU ..(((((((...((((...((((((.(((........)))(((.((((((...)))))).)))..)))))).((((((((((......)))))))))))))))))))))---........ ( -53.10) >DroSec_CAF1 16796 117 - 1 UACGGCGGCUCUGGCUGUAGCGUAAUGACUGAACGAUGUCGCACCGGCGAGGGUUGUUGUUGGGUUGAUGCGAUUGCUGCUGACGCCGCAGCAGCAGUAGCCGCCGCCG---AUGUUGGU ..(((((((...((((....((((.(((((.(((((((((((....))))....))))))).))))).))))((((((((((......)))))))))))))))))))))---........ ( -54.50) >DroSim_CAF1 11069 117 - 1 UACGGCGGCUCUGGCUGUAGCGUAAUGACUGAACGAUGUCGCACCGGCGAGGGUUGUUGUUGCGUUGAUGCGAUUGCUGCUGACGCCGCAGCAGCAGUAGCCGCCGCCG---AUGUUGGU ..(((((((...((((.((((((((..((.......(.((((....)))).)...))..)))))))).....((((((((((......)))))))))))))))))))))---........ ( -54.10) >DroEre_CAF1 9673 117 - 1 AACGGCGGCUCUGGCUGUAGCGUAACGACUGAGCGAUGUCGCACCGGCGAGGGUUGUUGUUGCGUUGAUGCGAUUGCUGCUGACGCCGCAGCACCAGUAGCCGCCGCCG---ACGUUGGU ...((((((((((((..((((((((((((...(((....))).((.....))...))))))))))))..))...((((((.......)))))).))).)))))))((((---....)))) ( -54.00) >DroYak_CAF1 10943 120 - 1 UACGGCGGCUCUGGCUGUAGCGUAAUGACUGAGCGAUGUCGCACCGGCGAGGGUUGUUGUUGCGUUGAUGCGAUUGCUGCUGACGCCGCAGCACCAGUAGCCGCCGCCGACGACGUUGGU ...(((((((((((.(((.((...........(((....)))...((((..(((.((.((((((....)))))).)).)))..)))))).))))))).)))))))((((((...)))))) ( -55.20) >consensus UACGGCGGCUCUGGCUGUAGCGUAAUGACUGAACGAUGUCGCACCGGCGAGGGUUGUUGUUGCGUUGAUGCGAUUGCUGCUGACGCCGCAGCAGCAGUAGCCGCCGCCG___ACGUUGGU ..(((((((...((((.((((((((((((..(((..(.((((....)))).)))))))))))))))).....((((((((((......)))))))))))))))))))))........... (-49.56 = -49.96 + 0.40)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:22:10 2006