
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 14,808,120 – 14,808,276 |
| Length | 156 |
| Max. P | 0.999729 |

| Location | 14,808,120 – 14,808,236 |
|---|---|
| Length | 116 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 96.09 |
| Mean single sequence MFE | -37.96 |
| Consensus MFE | -34.30 |
| Energy contribution | -34.94 |
| Covariance contribution | 0.64 |
| Combinations/Pair | 1.03 |
| Mean z-score | -2.76 |
| Structure conservation index | 0.90 |
| SVM decision value | 3.96 |
| SVM RNA-class probability | 0.999729 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 14808120 116 + 20766785 UGGCUGUAGCUGGUUUUUAUCUUGUGCCGUUAACACAGCCAUUACGUGUCUCUUUCGCACUCGCACUGCACAUUGGUGUUCGUGUGUCUGUGCAAGACUGCACGU----AACUGUAUUAC ((((((((((.(((...........))))))...)))))))((((((((.((((.((((..(((((.((((....))))..)))))..)))).))))..))))))----))......... ( -40.80) >DroSec_CAF1 9974 116 + 1 UGGCCGUAGCUGGUUUUUAUCUUGUGCCGUUAACACAGCCAUUACGUGUCUCUUUCGCACUCGCAAUGCACAUUGGUGUUCCUGUGUCUGUGCAAGACUGCACGU----AACUGUAUUAC ((((..((((.(((...........))))))).....))))((((((((.((((.((((..((((..((((....))))...))))..)))).))))..))))))----))......... ( -33.90) >DroSim_CAF1 4247 116 + 1 UGGCCGUAGCUGGUUUUUAUCUUGUGCCGUUAACACAGCCAUUACGUGUCUCUUUCGCACUCGCACUGCACAUUGGUGUUCGUGUGUCUGUGCAAGACUGCACGU----AACUGUAUUAC ((((..((((.(((...........))))))).....))))((((((((.((((.((((..(((((.((((....))))..)))))..)))).))))..))))))----))......... ( -36.60) >DroEre_CAF1 2883 120 + 1 UGGCUGUAGCUGGUUUUUAUCUUGUGCCGUUAACACAGCCAUUACGUGUCUCUUUCGCACUCGCACUGCACAUCGGUGCUUGUGUGUCUGUGCAAGACUGCACGUACAUAACUGUAUUAC ((((((((((.(((...........))))))...))))))).(((((((.((((.((((..(((((.((((....))))..)))))..)))).))))..))))))).............. ( -42.00) >DroYak_CAF1 4050 116 + 1 UGGCUGUAGCUGGUUUUUAUCUUGUGCCGUUAACACAGCCAUUACGUGUCUCUUUCGCACUCGCACUGCACAUUGGUAUUCGUGUGUCUGUGCAAGACUGCACGU----AACUGUAUUAC ((((((((((.(((...........))))))...)))))))((((((((.((((.((((..((((((((......)))...)))))..)))).))))..))))))----))......... ( -36.50) >consensus UGGCUGUAGCUGGUUUUUAUCUUGUGCCGUUAACACAGCCAUUACGUGUCUCUUUCGCACUCGCACUGCACAUUGGUGUUCGUGUGUCUGUGCAAGACUGCACGU____AACUGUAUUAC ((((((((((.(((...........))))))...)))))))..((((((.((((.((((..(((((.((((....))))..)))))..)))).))))..))))))............... (-34.30 = -34.94 + 0.64)

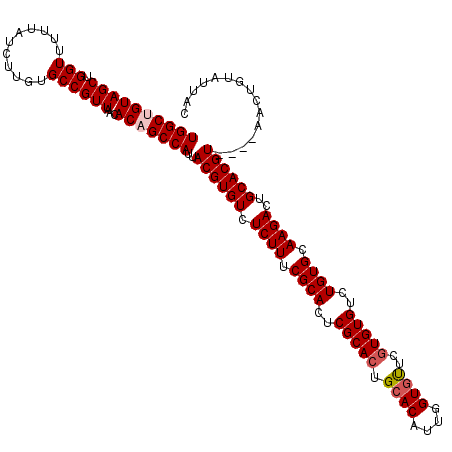
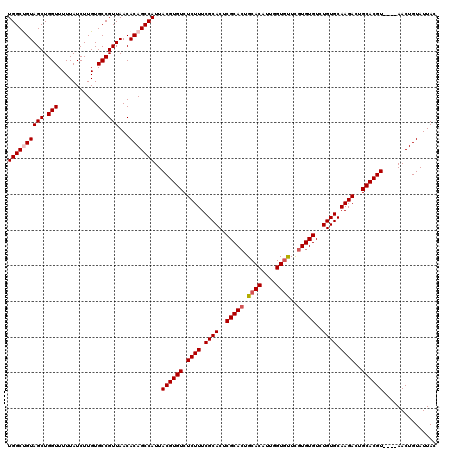
| Location | 14,808,120 – 14,808,236 |
|---|---|
| Length | 116 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 96.09 |
| Mean single sequence MFE | -34.13 |
| Consensus MFE | -32.72 |
| Energy contribution | -32.92 |
| Covariance contribution | 0.20 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.25 |
| Structure conservation index | 0.96 |
| SVM decision value | 3.91 |
| SVM RNA-class probability | 0.999700 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 14808120 116 - 20766785 GUAAUACAGUU----ACGUGCAGUCUUGCACAGACACACGAACACCAAUGUGCAGUGCGAGUGCGAAAGAGACACGUAAUGGCUGUGUUAACGGCACAAGAUAAAAACCAGCUACAGCCA ........(((----((((((...(((((((.(.((((..........))))).)))))))..(....).).))))))))(((((((((...((.............))))).)))))). ( -32.62) >DroSec_CAF1 9974 116 - 1 GUAAUACAGUU----ACGUGCAGUCUUGCACAGACACAGGAACACCAAUGUGCAUUGCGAGUGCGAAAGAGACACGUAAUGGCUGUGUUAACGGCACAAGAUAAAAACCAGCUACGGCCA (((((...)))----))((((.((...((((((.((((((....))..))))(((((((.((.(....)..)).))))))).))))))..)).)))).............((....)).. ( -34.70) >DroSim_CAF1 4247 116 - 1 GUAAUACAGUU----ACGUGCAGUCUUGCACAGACACACGAACACCAAUGUGCAGUGCGAGUGCGAAAGAGACACGUAAUGGCUGUGUUAACGGCACAAGAUAAAAACCAGCUACGGCCA .((((((((((----((((((...(((((((.(.((((..........))))).)))))))..(....).).))))))...)))))))))..(((...((...........))...))). ( -33.40) >DroEre_CAF1 2883 120 - 1 GUAAUACAGUUAUGUACGUGCAGUCUUGCACAGACACACAAGCACCGAUGUGCAGUGCGAGUGCGAAAGAGACACGUAAUGGCUGUGUUAACGGCACAAGAUAAAAACCAGCUACAGCCA .((((((((((((.(((((((...(((((((.(.....)..((((....)))).)))))))..(....).).))))))))))))))))))..(((.....................))). ( -37.30) >DroYak_CAF1 4050 116 - 1 GUAAUACAGUU----ACGUGCAGUCUUGCACAGACACACGAAUACCAAUGUGCAGUGCGAGUGCGAAAGAGACACGUAAUGGCUGUGUUAACGGCACAAGAUAAAAACCAGCUACAGCCA ........(((----((((((...(((((((.(.((((..........))))).)))))))..(....).).))))))))(((((((((...((.............))))).)))))). ( -32.62) >consensus GUAAUACAGUU____ACGUGCAGUCUUGCACAGACACACGAACACCAAUGUGCAGUGCGAGUGCGAAAGAGACACGUAAUGGCUGUGUUAACGGCACAAGAUAAAAACCAGCUACAGCCA .((((((((((....((((((...(((((((.(.((((..........))))).)))))))..(....).).)))))...))))))))))..(((...((...........))...))). (-32.72 = -32.92 + 0.20)

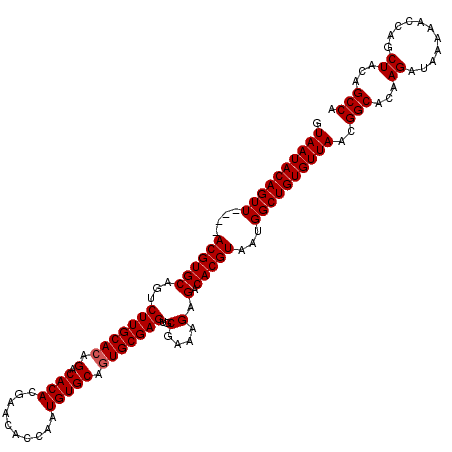
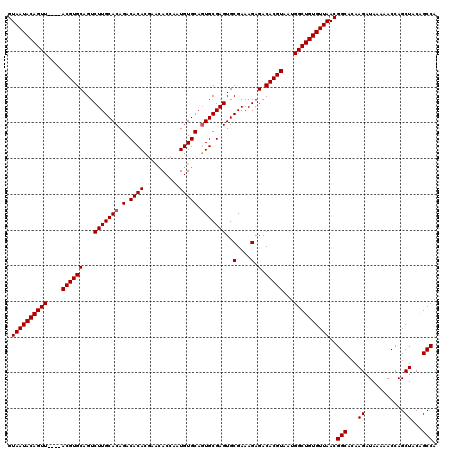
| Location | 14,808,160 – 14,808,276 |
|---|---|
| Length | 116 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 95.92 |
| Mean single sequence MFE | -31.38 |
| Consensus MFE | -28.12 |
| Energy contribution | -28.36 |
| Covariance contribution | 0.24 |
| Combinations/Pair | 1.03 |
| Mean z-score | -2.09 |
| Structure conservation index | 0.90 |
| SVM decision value | 2.69 |
| SVM RNA-class probability | 0.996405 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 14808160 116 + 20766785 AUUACGUGUCUCUUUCGCACUCGCACUGCACAUUGGUGUUCGUGUGUCUGUGCAAGACUGCACGU----AACUGUAUUACCCAUUCGUACAUGCGUAUACGUAUUUCCGCCUAUUGAUAA .((((((((.((((.((((..(((((.((((....))))..)))))..)))).))))..))))))----))...(((((......((...(((((....)))))...)).....))))). ( -32.40) >DroSec_CAF1 10014 116 + 1 AUUACGUGUCUCUUUCGCACUCGCAAUGCACAUUGGUGUUCCUGUGUCUGUGCAAGACUGCACGU----AACUGUAUUACCCAUUCGUACAUGCGUAUACGUAUUUCCGCCUAUUGAUAA .((((((((.((((.((((..((((..((((....))))...))))..)))).))))..))))))----))...(((((......((...(((((....)))))...)).....))))). ( -29.70) >DroSim_CAF1 4287 116 + 1 AUUACGUGUCUCUUUCGCACUCGCACUGCACAUUGGUGUUCGUGUGUCUGUGCAAGACUGCACGU----AACUGUAUUACCCAUUCGUACAUGCGUAUACGUAUUUCCGCCUAUUGAUAA .((((((((.((((.((((..(((((.((((....))))..)))))..)))).))))..))))))----))...(((((......((...(((((....)))))...)).....))))). ( -32.40) >DroEre_CAF1 2923 120 + 1 AUUACGUGUCUCUUUCGCACUCGCACUGCACAUCGGUGCUUGUGUGUCUGUGCAAGACUGCACGUACAUAACUGUAUUACCCGUUCGUACAUGCGUAUACGUAUUUCCGUCUAUUGAUAA ..(((((((.((((.((((..(((((.((((....))))..)))))..)))).))))..)))))))........(((((......((...(((((....)))))...)).....))))). ( -34.30) >DroYak_CAF1 4090 116 + 1 AUUACGUGUCUCUUUCGCACUCGCACUGCACAUUGGUAUUCGUGUGUCUGUGCAAGACUGCACGU----AACUGUAUUACCCAUUCGUACAUGCGUAUACGUAUUUCCGCCUAUUGAUAA .((((((((.((((.((((..((((((((......)))...)))))..)))).))))..))))))----))...(((((......((...(((((....)))))...)).....))))). ( -28.10) >consensus AUUACGUGUCUCUUUCGCACUCGCACUGCACAUUGGUGUUCGUGUGUCUGUGCAAGACUGCACGU____AACUGUAUUACCCAUUCGUACAUGCGUAUACGUAUUUCCGCCUAUUGAUAA ...((((((.((((.((((..(((((.((((....))))..)))))..)))).))))..)))))).........(((((......((...(((((....)))))...)).....))))). (-28.12 = -28.36 + 0.24)


| Location | 14,808,160 – 14,808,276 |
|---|---|
| Length | 116 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 95.92 |
| Mean single sequence MFE | -30.39 |
| Consensus MFE | -27.10 |
| Energy contribution | -26.30 |
| Covariance contribution | -0.80 |
| Combinations/Pair | 1.14 |
| Mean z-score | -1.32 |
| Structure conservation index | 0.89 |
| SVM decision value | 0.44 |
| SVM RNA-class probability | 0.736009 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
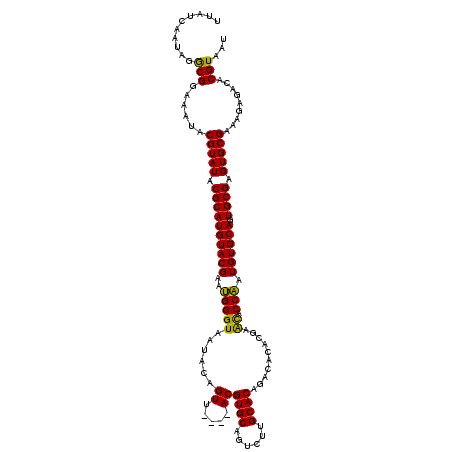
>2R_DroMel_CAF1 14808160 116 - 20766785 UUAUCAAUAGGCGGAAAUACGUAUACGCAUGUACGAAUGGGUAAUACAGUU----ACGUGCAGUCUUGCACAGACACACGAACACCAAUGUGCAGUGCGAGUGCGAAAGAGACACGUAAU ..........(((......))).(((.(((......))).))).....(((----((((((...(((((((.(.((((..........))))).)))))))..(....).).)))))))) ( -29.30) >DroSec_CAF1 10014 116 - 1 UUAUCAAUAGGCGGAAAUACGUAUACGCAUGUACGAAUGGGUAAUACAGUU----ACGUGCAGUCUUGCACAGACACAGGAACACCAAUGUGCAUUGCGAGUGCGAAAGAGACACGUAAU ..........(((......(((((.((((((((((..((((((((...)))----))((((......)))).............))).)))))).)))).))))).........)))... ( -28.86) >DroSim_CAF1 4287 116 - 1 UUAUCAAUAGGCGGAAAUACGUAUACGCAUGUACGAAUGGGUAAUACAGUU----ACGUGCAGUCUUGCACAGACACACGAACACCAAUGUGCAGUGCGAGUGCGAAAGAGACACGUAAU ..........(((......))).(((.(((......))).))).....(((----((((((...(((((((.(.((((..........))))).)))))))..(....).).)))))))) ( -29.30) >DroEre_CAF1 2923 120 - 1 UUAUCAAUAGACGGAAAUACGUAUACGCAUGUACGAACGGGUAAUACAGUUAUGUACGUGCAGUCUUGCACAGACACACAAGCACCGAUGUGCAGUGCGAGUGCGAAAGAGACACGUAAU .................(((((....((((((((((((.(......).))).))))))))).(((((((((.(.(((....((((....)))).))))..))))....)))))))))).. ( -35.20) >DroYak_CAF1 4090 116 - 1 UUAUCAAUAGGCGGAAAUACGUAUACGCAUGUACGAAUGGGUAAUACAGUU----ACGUGCAGUCUUGCACAGACACACGAAUACCAAUGUGCAGUGCGAGUGCGAAAGAGACACGUAAU ..........(((......))).(((.(((......))).))).....(((----((((((...(((((((.(.((((..........))))).)))))))..(....).).)))))))) ( -29.30) >consensus UUAUCAAUAGGCGGAAAUACGUAUACGCAUGUACGAAUGGGUAAUACAGUU____ACGUGCAGUCUUGCACAGACACACGAACACCAAUGUGCAGUGCGAGUGCGAAAGAGACACGUAAU ..........(((......(((((.((((((((((..(((((......((.....))((((......))))..........)).))).)))))).)))).))))).........)))... (-27.10 = -26.30 + -0.80)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:21:41 2006