
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 14,807,769 – 14,807,927 |
| Length | 158 |
| Max. P | 0.999992 |

| Location | 14,807,769 – 14,807,887 |
|---|---|
| Length | 118 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 91.79 |
| Mean single sequence MFE | -40.26 |
| Consensus MFE | -34.96 |
| Energy contribution | -36.36 |
| Covariance contribution | 1.40 |
| Combinations/Pair | 1.10 |
| Mean z-score | -4.50 |
| Structure conservation index | 0.87 |
| SVM decision value | 4.25 |
| SVM RNA-class probability | 0.999851 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 14807769 118 + 20766785 CGCACACAUCCACAUACAAUUGGGAAGUGCACAGGUGUGGGUGGAUCUGCUCUGUGUGCAAAUAACAAAUUGUGAAAAACAACUCGCACACA--UGCAGAUCACCUAGUGCACCAAAUAA .((((...(((.((......))))).))))...((((((((((.((((((..(((((((.....((.....))((........)))))))))--.)))))))))))...)))))...... ( -43.40) >DroSec_CAF1 9628 118 + 1 CGCACACAUCGCAAUACUUUUGGGAAGUGCGCAAGUGUGGGUGGAUCUGCUCUGUGUGCAAAUAACAAAUUAUGAAAAACAACUCGCACACA--UGCAGAUCACCUAGUGCACCAAAUAA .((.(((.((.(((.....))).)).))).))..(((((((((.((((((..(((((((..((((....))))((........)))))))))--.)))))))))))...))))....... ( -40.90) >DroSim_CAF1 3900 118 + 1 CGCACACAUCCACAUACUUUUGGGAAGUGCGCAGGUGUGGGUGGAUCUGCUCUGUGUGCAAAUAACAAAUUGUGAAAAACAACUCGCACACA--UGCAGAUCACCUAGUGCACCAAAUAA .((.(((.(((.((......))))).))).)).((((((((((.((((((..(((((((.....((.....))((........)))))))))--.)))))))))))...)))))...... ( -46.10) >DroEre_CAF1 2541 119 + 1 UGCACACAAUCAUAUACCAUUGGGAACUGCGCAGGUGUGGGUGGAUCUGCUCUGUGUGCAAAUAACAAAUUGUGAAAAACAACUCGCACACACAAGC-GAUUACCUAGUGCACCAAAUAA (((.((...(((........)))....)).)))((((((((((.(((.(((.(((((((.....((.....))((........)))))))))..)))-))))))))...)))))...... ( -31.60) >DroYak_CAF1 3709 120 + 1 UGCACACAGCCACAUACCUUUGAGAACUGCGCAGGUGUGGGUGGAUCUGCUCUGUGUGCAAAUAACAAAUUGUGAAAAACAACUCGCACACACAAGCAGAUCACCUAAUGCACCAAAUAA ((((....(((.(((((((.((.......)).)))))))))).((((((((.(((((((.....((.....))((........)))))))))..))))))))......))))........ ( -39.30) >consensus CGCACACAUCCACAUACCUUUGGGAAGUGCGCAGGUGUGGGUGGAUCUGCUCUGUGUGCAAAUAACAAAUUGUGAAAAACAACUCGCACACA__UGCAGAUCACCUAGUGCACCAAAUAA .((.(((.(((.((......))))).))).)).((((((((((.((((((..(((((((..((((....))))((........)))))))))...)))))))))))...)))))...... (-34.96 = -36.36 + 1.40)

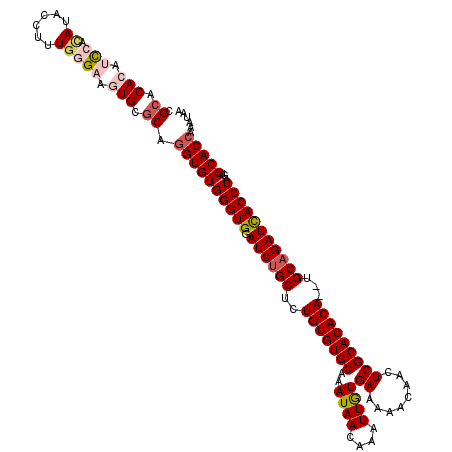
| Location | 14,807,769 – 14,807,887 |
|---|---|
| Length | 118 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 91.79 |
| Mean single sequence MFE | -42.77 |
| Consensus MFE | -36.63 |
| Energy contribution | -37.59 |
| Covariance contribution | 0.96 |
| Combinations/Pair | 1.09 |
| Mean z-score | -3.87 |
| Structure conservation index | 0.86 |
| SVM decision value | 4.24 |
| SVM RNA-class probability | 0.999846 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
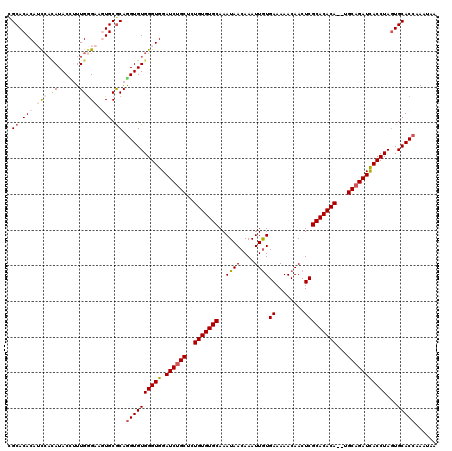
>2R_DroMel_CAF1 14807769 118 - 20766785 UUAUUUGGUGCACUAGGUGAUCUGCA--UGUGUGCGAGUUGUUUUUCACAAUUUGUUAUUUGCACACAGAGCAGAUCCACCCACACCUGUGCACUUCCCAAUUGUAUGUGGAUGUGUGCG ......((((.....((((((((((.--(((((((((((..................)))))))))))..)))))).))))..)))).(..(((.(((((......)).))).)))..). ( -45.07) >DroSec_CAF1 9628 118 - 1 UUAUUUGGUGCACUAGGUGAUCUGCA--UGUGUGCGAGUUGUUUUUCAUAAUUUGUUAUUUGCACACAGAGCAGAUCCACCCACACUUGCGCACUUCCCAAAAGUAUUGCGAUGUGUGCG ......((((.....((((((((((.--(((((((((((.(((......))).....)))))))))))..)))))).))))..)))).((((((.((.(((.....))).)).)))))). ( -42.30) >DroSim_CAF1 3900 118 - 1 UUAUUUGGUGCACUAGGUGAUCUGCA--UGUGUGCGAGUUGUUUUUCACAAUUUGUUAUUUGCACACAGAGCAGAUCCACCCACACCUGCGCACUUCCCAAAAGUAUGUGGAUGUGUGCG ......((((.....((((((((((.--(((((((((((..................)))))))))))..)))))).))))..)))).((((((.(((((......)).))).)))))). ( -47.47) >DroEre_CAF1 2541 119 - 1 UUAUUUGGUGCACUAGGUAAUC-GCUUGUGUGUGCGAGUUGUUUUUCACAAUUUGUUAUUUGCACACAGAGCAGAUCCACCCACACCUGCGCAGUUCCCAAUGGUAUAUGAUUGUGUGCA ......((((.....(((.(((-(((((((((((((((((((.....))))))))).....))))))).))).)))..)))..)))).((((((((((....)).....))))))))... ( -33.40) >DroYak_CAF1 3709 120 - 1 UUAUUUGGUGCAUUAGGUGAUCUGCUUGUGUGUGCGAGUUGUUUUUCACAAUUUGUUAUUUGCACACAGAGCAGAUCCACCCACACCUGCGCAGUUCUCAAAGGUAUGUGGCUGUGUGCA ......((((.....(((((((((((((((((((((((((((.....))))))))).....))))))).))))))).))))..)))).((((((((..((......)).))))))))... ( -45.60) >consensus UUAUUUGGUGCACUAGGUGAUCUGCA__UGUGUGCGAGUUGUUUUUCACAAUUUGUUAUUUGCACACAGAGCAGAUCCACCCACACCUGCGCACUUCCCAAAAGUAUGUGGAUGUGUGCG ......((((.....((((((((((...(((((((((((..................)))))))))))..)))))).))))..)))).((((((...(((........)))..)))))). (-36.63 = -37.59 + 0.96)


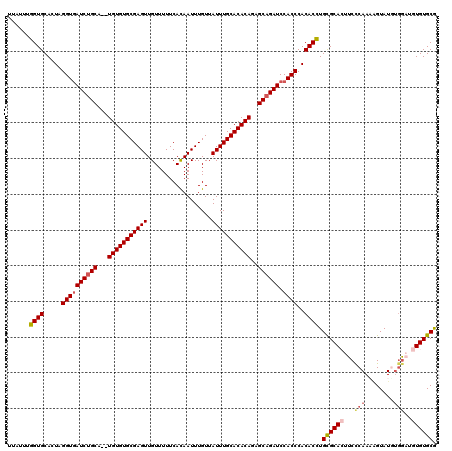
| Location | 14,807,809 – 14,807,927 |
|---|---|
| Length | 118 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 93.80 |
| Mean single sequence MFE | -27.04 |
| Consensus MFE | -25.88 |
| Energy contribution | -25.76 |
| Covariance contribution | -0.12 |
| Combinations/Pair | 1.07 |
| Mean z-score | -3.40 |
| Structure conservation index | 0.96 |
| SVM decision value | 5.69 |
| SVM RNA-class probability | 0.999992 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 14807809 118 + 20766785 GUGGAUCUGCUCUGUGUGCAAAUAACAAAUUGUGAAAAACAACUCGCACACA--UGCAGAUCACCUAGUGCACCAAAUAAAAGCAAUAAAUAAACACAUUUCCAAACGGGCAACAAGUUG ((((((((((..(((((((.....((.....))((........)))))))))--.)))))))......(((...........))).........))).....(((...(....)...))) ( -28.30) >DroSec_CAF1 9668 118 + 1 GUGGAUCUGCUCUGUGUGCAAAUAACAAAUUAUGAAAAACAACUCGCACACA--UGCAGAUCACCUAGUGCACCAAAUAAAAGCAAUAAAUAAACACAUUUCCACACGGGCAACAAGUUG ((((((((((..(((((((..((((....))))((........)))))))))--.)))))))......(((...........))).........)))...........(....)...... ( -26.60) >DroSim_CAF1 3940 118 + 1 GUGGAUCUGCUCUGUGUGCAAAUAACAAAUUGUGAAAAACAACUCGCACACA--UGCAGAUCACCUAGUGCACCAAAUAAAAGCAAUAAAUAAACACAUUUCCACACGGGCAACAAGUUG ((((((((((..(((((((.....((.....))((........)))))))))--.)))))))......(((...........))).........)))...........(....)...... ( -27.20) >DroEre_CAF1 2581 119 + 1 GUGGAUCUGCUCUGUGUGCAAAUAACAAAUUGUGAAAAACAACUCGCACACACAAGC-GAUUACCUAGUGCACCAAAUAAAAGCAAUAAAUAAACACAUUCACACACAGGCAACAUGCCU (((((((.(((.(((((((.....((.....))((........)))))))))..)))-))))......(((...........))).........)))..........((((.....)))) ( -24.10) >DroYak_CAF1 3749 120 + 1 GUGGAUCUGCUCUGUGUGCAAAUAACAAAUUGUGAAAAACAACUCGCACACACAAGCAGAUCACCUAAUGCACCAAAUAAAAGCAAUAAAUAAACACAUUCACACACAGGCAACAUGUUG (((((((((((.(((((((.....((.....))((........)))))))))..))))))))......(((...........))).........)))........((((....).))).. ( -29.00) >consensus GUGGAUCUGCUCUGUGUGCAAAUAACAAAUUGUGAAAAACAACUCGCACACA__UGCAGAUCACCUAGUGCACCAAAUAAAAGCAAUAAAUAAACACAUUUCCACACGGGCAACAAGUUG ((((((((((..(((((((..((((....))))((........)))))))))...)))))))......(((...........))).........)))...........(....)...... (-25.88 = -25.76 + -0.12)

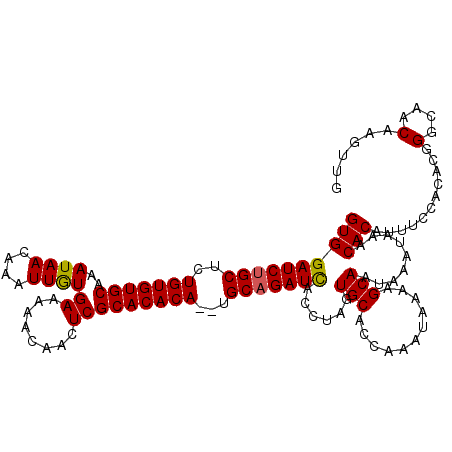
| Location | 14,807,809 – 14,807,927 |
|---|---|
| Length | 118 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 93.80 |
| Mean single sequence MFE | -37.97 |
| Consensus MFE | -31.75 |
| Energy contribution | -32.51 |
| Covariance contribution | 0.76 |
| Combinations/Pair | 1.08 |
| Mean z-score | -3.86 |
| Structure conservation index | 0.84 |
| SVM decision value | 3.61 |
| SVM RNA-class probability | 0.999450 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 14807809 118 - 20766785 CAACUUGUUGCCCGUUUGGAAAUGUGUUUAUUUAUUGCUUUUAUUUGGUGCACUAGGUGAUCUGCA--UGUGUGCGAGUUGUUUUUCACAAUUUGUUAUUUGCACACAGAGCAGAUCCAC ..(((((.((((((..(((((..(((......)))...)))))..))).))).)))))(((((((.--(((((((((((..................)))))))))))..)))))))... ( -37.57) >DroSec_CAF1 9668 118 - 1 CAACUUGUUGCCCGUGUGGAAAUGUGUUUAUUUAUUGCUUUUAUUUGGUGCACUAGGUGAUCUGCA--UGUGUGCGAGUUGUUUUUCAUAAUUUGUUAUUUGCACACAGAGCAGAUCCAC ..(((((.((((((.((((((..(((......)))...)))))).))).))).)))))(((((((.--(((((((((((.(((......))).....)))))))))))..)))))))... ( -37.90) >DroSim_CAF1 3940 118 - 1 CAACUUGUUGCCCGUGUGGAAAUGUGUUUAUUUAUUGCUUUUAUUUGGUGCACUAGGUGAUCUGCA--UGUGUGCGAGUUGUUUUUCACAAUUUGUUAUUUGCACACAGAGCAGAUCCAC ..(((((.((((((.((((((..(((......)))...)))))).))).))).)))))(((((((.--(((((((((((..................)))))))))))..)))))))... ( -37.87) >DroEre_CAF1 2581 119 - 1 AGGCAUGUUGCCUGUGUGUGAAUGUGUUUAUUUAUUGCUUUUAUUUGGUGCACUAGGUAAUC-GCUUGUGUGUGCGAGUUGUUUUUCACAAUUUGUUAUUUGCACACAGAGCAGAUCCAC .((....(((((((((((..((((((.(((((((.(((...........))).))))))).)-))........(((((((((.....))))))))).)))..))))))).))))..)).. ( -38.70) >DroYak_CAF1 3749 120 - 1 CAACAUGUUGCCUGUGUGUGAAUGUGUUUAUUUAUUGCUUUUAUUUGGUGCAUUAGGUGAUCUGCUUGUGUGUGCGAGUUGUUUUUCACAAUUUGUUAUUUGCACACAGAGCAGAUCCAC .........(((((((((((((((....))))))).(((.......)))))).)))))((((((((((((((((((((((((.....))))))))).....))))))).))))))))... ( -37.80) >consensus CAACUUGUUGCCCGUGUGGAAAUGUGUUUAUUUAUUGCUUUUAUUUGGUGCACUAGGUGAUCUGCA__UGUGUGCGAGUUGUUUUUCACAAUUUGUUAUUUGCACACAGAGCAGAUCCAC ..(((((.((((((.((((((..(((......)))...)))))).))).))).)))))(((((((...(((((((((((..................)))))))))))..)))))))... (-31.75 = -32.51 + 0.76)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:21:38 2006