
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 14,675,431 – 14,675,624 |
| Length | 193 |
| Max. P | 0.993837 |

| Location | 14,675,431 – 14,675,546 |
|---|---|
| Length | 115 |
| Sequences | 6 |
| Columns | 115 |
| Reading direction | forward |
| Mean pairwise identity | 74.28 |
| Mean single sequence MFE | -51.53 |
| Consensus MFE | -30.92 |
| Energy contribution | -34.17 |
| Covariance contribution | 3.25 |
| Combinations/Pair | 1.21 |
| Mean z-score | -2.56 |
| Structure conservation index | 0.60 |
| SVM decision value | 2.43 |
| SVM RNA-class probability | 0.993837 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
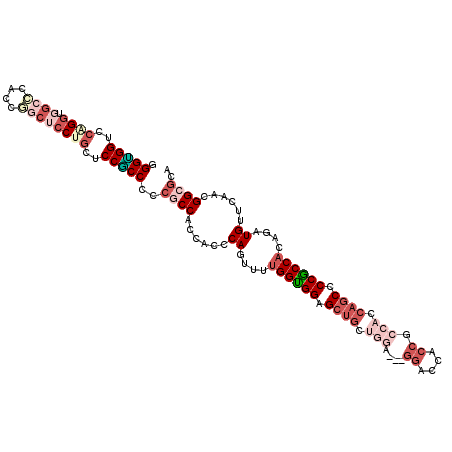
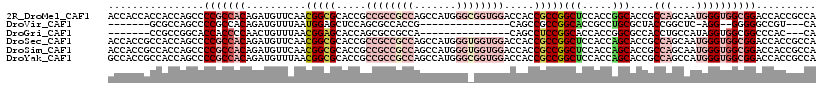
>2R_DroMel_CAF1 14675431 115 + 20766785 GGGUGGUCCAGGUGGCCCACCGGCUCCUGCUCCGCCCCCGCCACCACCCAGUUUUGGUGGAGCUGCUGGAGGAGGACCACCACCACCAGCCCCGCCACAGAUGUUCAACGGCGCA ((((((..((((.((((....))))))))..)))))).((((........((((((((((.((((.(((.((.......)).))).)))).)))))).)))).......)))).. ( -53.16) >DroGri_CAF1 5819 97 + 1 GGGUGGUCCCGGGCAAGUAGGUGCUCCAGCGCCGCCGCCACCGCCACCCACAUUCGGUGGUGCA------------------CCGCCGGCACCACCCCAACUGUUUAACGGAGCA (((((((.((((.(.....(((((....)))))((.(((((((...........))))))))).------------------..))))).)))))))...(((.....))).... ( -44.80) >DroSec_CAF1 6711 115 + 1 GGGCGGUCCAGGUGGCCCACCGGCUCCUGCUCCGCCCCCGCCACCACCCAGUUUUGGUGGAGCUGCUGGAGGAGGACCACCGCCACCAGCCCCGCCACAGAUGUUCAACGGCGCA ((((((..((((.((((....))))))))..)))))).((((........((((((((((.((((.(((.((.......)).))).)))).)))))).)))).......)))).. ( -55.56) >DroSim_CAF1 1788 115 + 1 GGGCGGUCCAGGUGGCCCACCGGCUCCUGCUCCGCCCCCGCCACCACCCAGUUUUGGUGGAGCUGCUGGAGGAGGACCACCGCCACCAGCCCCGCCACAGAUGUUCAACGGCGCA ((((((..((((.((((....))))))))..)))))).((((........((((((((((.((((.(((.((.......)).))).)))).)))))).)))).......)))).. ( -55.56) >DroWil_CAF1 8074 103 + 1 AGGAGGCGGCGUAGGAGUAGGAGCACCAGCACCUCCCCCACCACCGCCCACAUUUGGUGGUGGUG------------CACCGCCACCAGCGCCACCACAAAUGUUUAAUGGAAUU ..(.(((((.(..((.(.(((.((....)).))).).))..).))))))(((((((.((((((((------------(..........))))))))))))))))........... ( -42.40) >DroYak_CAF1 5292 112 + 1 AGGUGGUCCGGGUGGCCCACCAGCUCCUGCUCCACCGCCGCCACCACCCAGUUUUGGCGGAGCUGCUGGA---GGGCCACCGCCACCAGCCCCGCCACAGAUGUUUAACGGCGCA .((((((...((((((((.(((((....(((((...((((..((......))..))))))))).))))).---)))))))))))))).((.(((..((....))....))).)). ( -57.70) >consensus GGGUGGUCCAGGUGGCCCACCGGCUCCUGCUCCGCCCCCGCCACCACCCAGUUUUGGUGGAGCUGCUGGA___GGACCACCGCCACCAGCCCCGCCACAGAUGUUCAACGGCGCA .(((((..((((.((((....))))))))..)))))..((((......((....((((((.((((.(((....((....)).))).)))).))))))....))......)))).. (-30.92 = -34.17 + 3.25)

| Location | 14,675,431 – 14,675,546 |
|---|---|
| Length | 115 |
| Sequences | 6 |
| Columns | 115 |
| Reading direction | reverse |
| Mean pairwise identity | 74.28 |
| Mean single sequence MFE | -56.88 |
| Consensus MFE | -36.02 |
| Energy contribution | -38.47 |
| Covariance contribution | 2.45 |
| Combinations/Pair | 1.32 |
| Mean z-score | -1.95 |
| Structure conservation index | 0.63 |
| SVM decision value | 1.67 |
| SVM RNA-class probability | 0.970893 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 14675431 115 - 20766785 UGCGCCGUUGAACAUCUGUGGCGGGGCUGGUGGUGGUGGUCCUCCUCCAGCAGCUCCACCAAAACUGGGUGGUGGCGGGGGCGGAGCAGGAGCCGGUGGGCCACCUGGACCACCC ..(((((..(..((....(((.(((((((.(((.((.......)).))).))))))).)))....))..)..))))).(((.((..((((.(((....)))..))))..)).))) ( -57.00) >DroGri_CAF1 5819 97 - 1 UGCUCCGUUAAACAGUUGGGGUGGUGCCGGCGG------------------UGCACCACCGAAUGUGGGUGGCGGUGGCGGCGGCGCUGGAGCACCUACUUGCCCGGGACCACCC ..................(((((((.(((((((------------------((...)))))...(((((((.(..(((((....)))))..))))))))....)))).))))))) ( -44.90) >DroSec_CAF1 6711 115 - 1 UGCGCCGUUGAACAUCUGUGGCGGGGCUGGUGGCGGUGGUCCUCCUCCAGCAGCUCCACCAAAACUGGGUGGUGGCGGGGGCGGAGCAGGAGCCGGUGGGCCACCUGGACCGCCC ..(((((..(..((....(((.(((((((.(((.((.......)).))).))))))).)))....))..)..))))).((((((..((((.(((....)))..))))..)))))) ( -62.10) >DroSim_CAF1 1788 115 - 1 UGCGCCGUUGAACAUCUGUGGCGGGGCUGGUGGCGGUGGUCCUCCUCCAGCAGCUCCACCAAAACUGGGUGGUGGCGGGGGCGGAGCAGGAGCCGGUGGGCCACCUGGACCGCCC ..(((((..(..((....(((.(((((((.(((.((.......)).))).))))))).)))....))..)..))))).((((((..((((.(((....)))..))))..)))))) ( -62.10) >DroWil_CAF1 8074 103 - 1 AAUUCCAUUAAACAUUUGUGGUGGCGCUGGUGGCGGUG------------CACCACCACCAAAUGUGGGCGGUGGUGGGGGAGGUGCUGGUGCUCCUACUCCUACGCCGCCUCCU ...........((((((((((((((((((....)))))------------)..))))).)))))))((((((((.(((((((((.((....)).))).))))))))))))))... ( -52.40) >DroYak_CAF1 5292 112 - 1 UGCGCCGUUAAACAUCUGUGGCGGGGCUGGUGGCGGUGGCCC---UCCAGCAGCUCCGCCAAAACUGGGUGGUGGCGGCGGUGGAGCAGGAGCUGGUGGGCCACCCGGACCACCU .((.((((((........)))))).)).((((((((((((((---.(((((.((((((((....(((........))).))))))))....))))).))))))))..).))))). ( -62.80) >consensus UGCGCCGUUAAACAUCUGUGGCGGGGCUGGUGGCGGUGGUCC___UCCAGCAGCUCCACCAAAACUGGGUGGUGGCGGGGGCGGAGCAGGAGCCGGUGGGCCACCUGGACCACCC ..(((((..(..((....(((.(((((((.(((.............))).))))))).)))....))..)..))))).((((((..((((.(((....)))..))))..)))))) (-36.02 = -38.47 + 2.45)


| Location | 14,675,466 – 14,675,584 |
|---|---|
| Length | 118 |
| Sequences | 4 |
| Columns | 118 |
| Reading direction | forward |
| Mean pairwise identity | 96.05 |
| Mean single sequence MFE | -54.35 |
| Consensus MFE | -51.46 |
| Energy contribution | -51.02 |
| Covariance contribution | -0.44 |
| Combinations/Pair | 1.06 |
| Mean z-score | -1.58 |
| Structure conservation index | 0.95 |
| SVM decision value | 0.33 |
| SVM RNA-class probability | 0.692617 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 14675466 118 + 20766785 CCCCGCCACCACCCAGUUUUGGUGGAGCUGCUGGAGGAGGACCACCACCACCAGCCCCGCCACAGAUGUUCAACGGCGCACCGCCGCCGCCAGCCAUGGGCGGUGGACCACCGCCGGC ....(((........((((((((((.((((.(((.((.......)).))).)))).)))))).)))).......((((.....((((((((.......)))))))).....))))))) ( -52.40) >DroSec_CAF1 6746 118 + 1 CCCCGCCACCACCCAGUUUUGGUGGAGCUGCUGGAGGAGGACCACCGCCACCAGCCCCGCCACAGAUGUUCAACGGCGCACCGCCGCCGCCAGCCAUGGGUGGUGGACCACCGCCGGC ..(((((((((((((...(((((((.((.(((((.((.((....)).)).)))))...)).............(((((...))))))))))))...))))))))))........))). ( -54.30) >DroSim_CAF1 1823 118 + 1 CCCCGCCACCACCCAGUUUUGGUGGAGCUGCUGGAGGAGGACCACCGCCACCAGCCCCGCCACAGAUGUUCAACGGCGCACCGCCGCCGCCAGCCAUGGGUGGUGGACCACCGCCGGC ..(((((((((((((...(((((((.((.(((((.((.((....)).)).)))))...)).............(((((...))))))))))))...))))))))))........))). ( -54.30) >DroYak_CAF1 5327 115 + 1 CGCCGCCACCACCCAGUUUUGGCGGAGCUGCUGGA---GGGCCACCGCCACCAGCCCCGCCACAGAUGUUUAACGGCGCACCGCCGCCGCCAGCCAUGGGCGGUGGACCACCGCCGGC ....(((........((((((((((.((((.(((.---((....)).))).)))).)))))).)))).......((((.....((((((((.......)))))))).....))))))) ( -56.40) >consensus CCCCGCCACCACCCAGUUUUGGUGGAGCUGCUGGAGGAGGACCACCGCCACCAGCCCCGCCACAGAUGUUCAACGGCGCACCGCCGCCGCCAGCCAUGGGCGGUGGACCACCGCCGGC ....(((........((((((((((.((((.(((....((....)).))).)))).)))))).)))).......((((.....((((((((.......)))))))).....))))))) (-51.46 = -51.02 + -0.44)


| Location | 14,675,506 – 14,675,624 |
|---|---|
| Length | 118 |
| Sequences | 6 |
| Columns | 118 |
| Reading direction | forward |
| Mean pairwise identity | 74.56 |
| Mean single sequence MFE | -44.35 |
| Consensus MFE | -26.67 |
| Energy contribution | -29.73 |
| Covariance contribution | 3.06 |
| Combinations/Pair | 1.22 |
| Mean z-score | -1.28 |
| Structure conservation index | 0.60 |
| SVM decision value | -0.05 |
| SVM RNA-class probability | 0.508586 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 14675506 118 + 20766785 ACCACCACCACCAGCCCCGCCACAGAUGUUCAACGGCGCACCGCCGCCGCCAGCCAUGGGCGGUGGACCACCGCCGGCUCCACCGGCACCGCCAGCAAUGGGUGGCGGACCACCGCCA ......................((...(((...(((((......)))))..)))..))((((((((......(((((.....))))).((((((.(....).)))))).)))))))). ( -50.40) >DroVir_CAF1 4892 90 + 1 -------GCGCCAGCCCCGCCACAGAUGUUUAAUGGAGCUCCAGCGCCACCG---------------CAGCCGCCGGCACCGCCUGCGCUACCGGCUC-AGG--GGGGGCCGU---CA -------(((...(((((.((.((.........))(((((..(((((...((---------------..(((...)))..))...)))))...)))))-.))--.))))))))---.. ( -34.50) >DroGri_CAF1 5883 93 + 1 -------CCGCCGGCACCACCCCAACUGUUUAACGGAGCACCAGCGCCGCCA---------------CAGCCUCCGGCACCACCGGCGCCACCUGCCAUAGGUGGCGGCCCAC---CA -------..((((.((((.......(((.....))).(((...((((((...---------------..(((...))).....))))))....)))....)))).))))....---.. ( -35.10) >DroSec_CAF1 6786 118 + 1 ACCACCGCCACCAGCCCCGCCACAGAUGUUCAACGGCGCACCGCCGCCGCCAGCCAUGGGUGGUGGACCACCGCCGGCUCCACCAGCACCGCCAGCAAUGGGUGGCGGACCACCGCCA ....((((((((....(((((((..........(((((......)))))(((....))))))))))......((.(((............))).))....)))))))).......... ( -48.50) >DroSim_CAF1 1863 118 + 1 ACCACCGCCACCAGCCCCGCCACAGAUGUUCAACGGCGCACCGCCGCCGCCAGCCAUGGGUGGUGGACCACCGCCGGCUCCACCAGCACCGCCAGCAAUGGGUGGCGGACCACCGCCA ....((((((((....(((((((..........(((((......)))))(((....))))))))))......((.(((............))).))....)))))))).......... ( -48.50) >DroYak_CAF1 5364 118 + 1 GCCACCGCCACCAGCCCCGCCACAGAUGUUUAACGGCGCACCGCCGCCGCCAGCCAUGGGCGGUGGACCACCGCCGGCUCCACCAGCACCGCCAGCCAUGGGUGGCGGACCACCGCCA ....((((((((.((...((.............(((((.....((((((((.......)))))))).....)))))(((.....)))...))..))....)))))))).......... ( -49.10) >consensus ACCACCGCCACCAGCCCCGCCACAGAUGUUCAACGGCGCACCGCCGCCGCCAGCCAUGGG_GGUGGACCACCGCCGGCUCCACCAGCACCGCCAGCAAUGGGUGGCGGACCACCGCCA ................(((((((..........(((((.....((((((((.......)))))))).....)))))(((.....)))..............))))))).......... (-26.67 = -29.73 + 3.06)
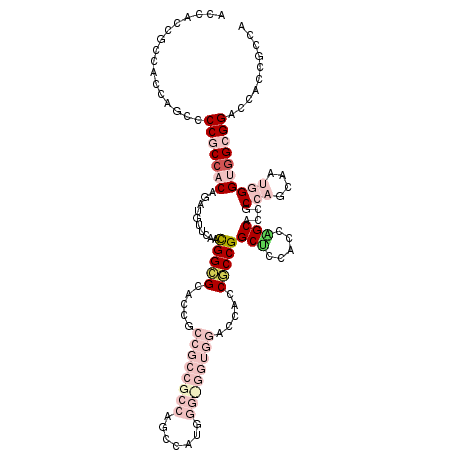
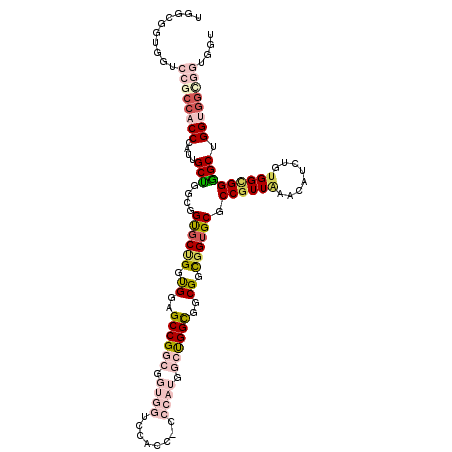
| Location | 14,675,506 – 14,675,624 |
|---|---|
| Length | 118 |
| Sequences | 6 |
| Columns | 118 |
| Reading direction | reverse |
| Mean pairwise identity | 74.56 |
| Mean single sequence MFE | -58.43 |
| Consensus MFE | -37.57 |
| Energy contribution | -40.13 |
| Covariance contribution | 2.56 |
| Combinations/Pair | 1.31 |
| Mean z-score | -1.19 |
| Structure conservation index | 0.64 |
| SVM decision value | 0.16 |
| SVM RNA-class probability | 0.610315 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 14675506 118 - 20766785 UGGCGGUGGUCCGCCACCCAUUGCUGGCGGUGCCGGUGGAGCCGGCGGUGGUCCACCGCCCAUGGCUGGCGGCGGCGGUGCGCCGUUGAACAUCUGUGGCGGGGCUGGUGGUGGUGGU ((((((....)))))).((((..(((.((((.((((((((.(((....)))))))))(((.((((.((.((((((((...))))))))..)).))))))))).)))).)))..)))). ( -65.30) >DroVir_CAF1 4892 90 - 1 UG---ACGGCCCCC--CCU-GAGCCGGUAGCGCAGGCGGUGCCGGCGGCUG---------------CGGUGGCGCUGGAGCUCCAUUAAACAUCUGUGGCGGGGCUGGCGC------- ..---.(((((((.--...-..(((((((.((....)).))))))).((..---------------(((((....(((....))).....)).)))..)))))))))....------- ( -46.00) >DroGri_CAF1 5883 93 - 1 UG---GUGGGCCGCCACCUAUGGCAGGUGGCGCCGGUGGUGCCGGAGGCUG---------------UGGCGGCGCUGGUGCUCCGUUAAACAGUUGGGGUGGUGCCGGCGG------- .(---(....))(((((((.....)))))))(((((((.((((...(((((---------------(.((((.((....)).))))...))))))..)))).)))))))..------- ( -49.70) >DroSec_CAF1 6786 118 - 1 UGGCGGUGGUCCGCCACCCAUUGCUGGCGGUGCUGGUGGAGCCGGCGGUGGUCCACCACCCAUGGCUGGCGGCGGCGGUGCGCCGUUGAACAUCUGUGGCGGGGCUGGUGGCGGUGGU ((((((....)))))).(((((((((.((((.((((..(((((((((((((....)))))....))))))(((((((...))))))).....))..)..))).)))).))))))))). ( -63.30) >DroSim_CAF1 1863 118 - 1 UGGCGGUGGUCCGCCACCCAUUGCUGGCGGUGCUGGUGGAGCCGGCGGUGGUCCACCACCCAUGGCUGGCGGCGGCGGUGCGCCGUUGAACAUCUGUGGCGGGGCUGGUGGCGGUGGU ((((((....)))))).(((((((((.((((.((((..(((((((((((((....)))))....))))))(((((((...))))))).....))..)..))).)))).))))))))). ( -63.30) >DroYak_CAF1 5364 118 - 1 UGGCGGUGGUCCGCCACCCAUGGCUGGCGGUGCUGGUGGAGCCGGCGGUGGUCCACCGCCCAUGGCUGGCGGCGGCGGUGCGCCGUUAAACAUCUGUGGCGGGGCUGGUGGCGGUGGC ((((((....)))))).((((.((((.((((.((((((((.(((....)))))))))(((((((..((((((((......))))))))..))..)).))))).)))).)))).)))). ( -63.00) >consensus UGGCGGUGGUCCGCCACCCAUUGCUGGCGGUGCUGGUGGAGCCGGCGGUGGUCCACC_CCCAUGGCUGGCGGCGGCGGUGCGCCGUUAAACAUCUGUGGCGGGGCUGGUGGCGGUGGU ..........((((((((....(((....((((((.((..((((((.((((........)))).))))))..)).)))))).((((((........))))))))).)))))))).... (-37.57 = -40.13 + 2.56)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:20:19 2006