
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
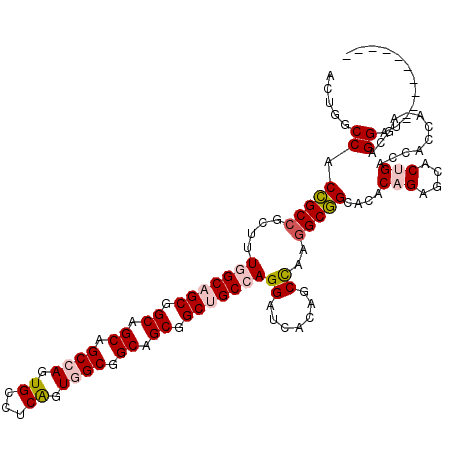
| Location | 14,605,911 – 14,606,020 |
| Length | 109 |
| Max. P | 0.877448 |

| Location | 14,605,911 – 14,606,020 |
|---|---|
| Length | 109 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 81.78 |
| Mean single sequence MFE | -51.12 |
| Consensus MFE | -34.75 |
| Energy contribution | -35.42 |
| Covariance contribution | 0.67 |
| Combinations/Pair | 1.10 |
| Mean z-score | -1.41 |
| Structure conservation index | 0.68 |
| SVM decision value | 0.00 |
| SVM RNA-class probability | 0.534868 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
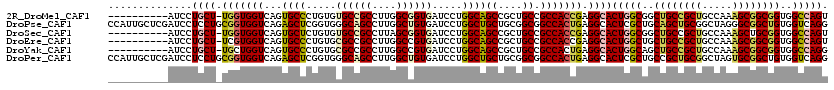
>2R_DroMel_CAF1 14605911 109 + 20766785 ACUGGCCACCGCCGCUUUGGCAGCGGCAGCCGCCAGUGCCUCGGUGGCGGCAGCGGCUGCCAGGAUCACCGCCAAGGCGGCACACAGGGCACUGACCACCA-AGCAGGAU---------- .(((......(((((((((((...(((((((((...((((.....))))...))))))))).((....)))))))))))))...(((....))).......-..)))...---------- ( -57.70) >DroPse_CAF1 233671 120 + 1 CCUGACCACAGCCGCCCUAGCCGCAGCUGCAGCGAGUGCCUCAGUGGCCGCCGCAGCAGCCAGGAUCACAGCCAAGGCUGCCCACCGAGCUCUGACCACCGCAGGAGGAUCGAGCAAUGG ((((......((.((....)).)).(((((.(((.(((((.....)).)))))).)))))))))....((((....))))....(((.((((.((((.((...)).)).))))))..))) ( -44.10) >DroSec_CAF1 237879 109 + 1 ACUGGCCACCGCAGCUUUGGCAGCGGCAGCCGCCAGUGCCUCGGUGGCGGCAGCGGCUGCCAGGAUCACCGCUAAGGCGGCACACAGAGCACUGACCACCA-AGCAGGAU---------- .((((((.((..(((..(((((((.((.(((((((.((...)).))))))).)).)))))))((....)))))..)).)))...(((....))).......-..)))...---------- ( -53.00) >DroEre_CAF1 245736 109 + 1 ACUGGCCACCGCCGCUUUGGCAGCGGCAGCAGCCAGUGCCUCGGUGGCGGCAGCGGCUGCCAGGAUCACGGCCAAGGCGGCGCACAGGGCACUGACCACGA-AGCAGGAU---------- .((((((..((((((((((((...((((((.((...((((.....))))...)).)))))).(.....).)))))))))))).....))).((........-)))))...---------- ( -51.70) >DroYak_CAF1 253891 109 + 1 CCUGGCCACCGCCGCUUUGGCAGCGGCAGCUGCCAGUGCCUCAGUGGCGGCAGCGGCUGCCAGGAUCACGGCCAAGGCGGCGCACAGGGCACUGACCAGCA-AGCAGGAU---------- (((((((..((((((((((((.(((((.((((((...(((.....))))))))).)))))..(.....).)))))))))))).....))).((........-))))))..---------- ( -58.30) >DroPer_CAF1 236168 120 + 1 CCUGACCACAGCCGCACUAGCCGCAGCGGCAGCGAGUGCCUCAGUGGCCGCCGCAGCAGCCAGGAUCACAGCCAAGGCUGCCCACCGAGCUCUGACCACCGCAGGAGGAUCGAGCAAUGG .....(((..(((((..........))))).(((.(((((.....)).)))))).((((((.((.......))..)))))).......((((.((((.((...)).)).))))))..))) ( -41.90) >consensus ACUGGCCACCGCCGCUUUGGCAGCGGCAGCAGCCAGUGCCUCAGUGGCGGCAGCGGCUGCCAGGAUCACAGCCAAGGCGGCACACAGAGCACUGACCACCA_AGCAGGAU__________ .....((.(((((....(((((((.((.((.((((.((...)).)))).)).)).)))))))((.......))..)))))....(((....)))............))............ (-34.75 = -35.42 + 0.67)

| Location | 14,605,911 – 14,606,020 |
|---|---|
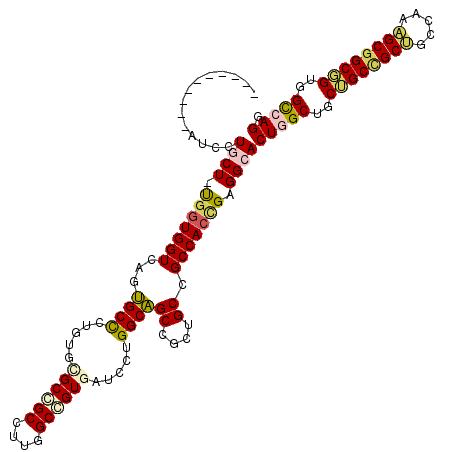
| Length | 109 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 81.78 |
| Mean single sequence MFE | -58.80 |
| Consensus MFE | -48.78 |
| Energy contribution | -48.70 |
| Covariance contribution | -0.08 |
| Combinations/Pair | 1.28 |
| Mean z-score | -1.99 |
| Structure conservation index | 0.83 |
| SVM decision value | 0.90 |
| SVM RNA-class probability | 0.877448 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 14605911 109 - 20766785 ----------AUCCUGCU-UGGUGGUCAGUGCCCUGUGUGCCGCCUUGGCGGUGAUCCUGGCAGCCGCUGCCGCCACCGAGGCACUGGCGGCUGCCGCUGCCAAAGCGGCGGUGGCCAGU ----------......((-((((((((((((..((((.((((((....))))))......)))).)))))..)))))))))..((((((.(((((((((.....))))))))).)))))) ( -62.60) >DroPse_CAF1 233671 120 - 1 CCAUUGCUCGAUCCUCCUGCGGUGGUCAGAGCUCGGUGGGCAGCCUUGGCUGUGAUCCUGGCUGCUGCGGCGGCCACUGAGGCACUCGCUGCAGCUGCGGCUAGGGCGGCUGUGGUCAGG ((((.((((...((.((...)).))...))))...))))(((((....)))))...(((((((((((((((((((.....)))...))))))))).((((((.....))))))))))))) ( -59.30) >DroSec_CAF1 237879 109 - 1 ----------AUCCUGCU-UGGUGGUCAGUGCUCUGUGUGCCGCCUUAGCGGUGAUCCUGGCAGCCGCUGCCGCCACCGAGGCACUGGCGGCUGCCGCUGCCAAAGCUGCGGUGGCCAGU ----------((((((((-.(((((((((....)))...))))))..))))).)))...(((((...)))))(((((((.(((..(((((((....)))))))..))).))))))).... ( -57.70) >DroEre_CAF1 245736 109 - 1 ----------AUCCUGCU-UCGUGGUCAGUGCCCUGUGCGCCGCCUUGGCCGUGAUCCUGGCAGCCGCUGCCGCCACCGAGGCACUGGCUGCUGCCGCUGCCAAAGCGGCGGUGGCCAGU ----------.....(((-((((((((((((..((((.(((.((....)).)))......)))).)))))..)))).))))))((((((..((((((((.....))))))))..)))))) ( -59.00) >DroYak_CAF1 253891 109 - 1 ----------AUCCUGCU-UGCUGGUCAGUGCCCUGUGCGCCGCCUUGGCCGUGAUCCUGGCAGCCGCUGCCGCCACUGAGGCACUGGCAGCUGCCGCUGCCAAAGCGGCGGUGGCCAGG ----------..((((..-.((((.(((((((((.(((.((.((..((((.((.......)).))))..)).))))).).)))))))))))).(((((((((.....))))))))))))) ( -57.50) >DroPer_CAF1 236168 120 - 1 CCAUUGCUCGAUCCUCCUGCGGUGGUCAGAGCUCGGUGGGCAGCCUUGGCUGUGAUCCUGGCUGCUGCGGCGGCCACUGAGGCACUCGCUGCCGCUGCGGCUAGUGCGGCUGUGGUCAGG (((((((..(.....)..)))))))...((.(.((((.((((((....)))))....((((((((.((((((((....(((...))))))))))).))))))))..).)))).).))... ( -56.70) >consensus __________AUCCUGCU_UGGUGGUCAGUGCCCUGUGCGCCGCCUUGGCCGUGAUCCUGGCAGCCGCUGCCGCCACCGAGGCACUGGCUGCUGCCGCUGCCAAAGCGGCGGUGGCCAGG ..............((((.(((((((...((((.....((((((....)))))).....))))((....)).))))))).))))(((((..((((((((.....))))))))..))))). (-48.78 = -48.70 + -0.08)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:19:45 2006