
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 14,578,318 – 14,578,428 |
| Length | 110 |
| Max. P | 0.611068 |

| Location | 14,578,318 – 14,578,428 |
|---|---|
| Length | 110 |
| Sequences | 6 |
| Columns | 115 |
| Reading direction | reverse |
| Mean pairwise identity | 73.03 |
| Mean single sequence MFE | -34.37 |
| Consensus MFE | -12.51 |
| Energy contribution | -13.43 |
| Covariance contribution | 0.92 |
| Combinations/Pair | 1.35 |
| Mean z-score | -2.00 |
| Structure conservation index | 0.36 |
| SVM decision value | 0.16 |
| SVM RNA-class probability | 0.611068 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
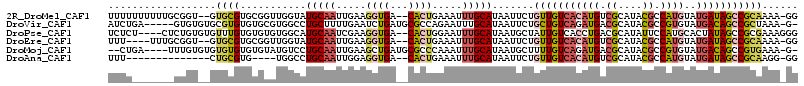
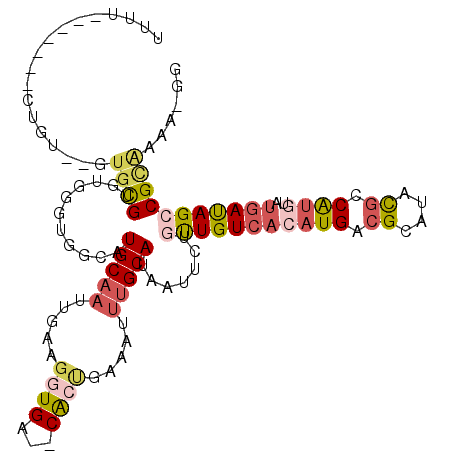
>2R_DroMel_CAF1 14578318 110 - 20766785 UUUUUUUUUUGCGGU--GUGCGUGCGGUUGGUAUGCAAUUGAAGGUGA--CACUGAAAUUUGCAUAAUUCUGUUGUCACAUGUCGCAUACGCCAUGUAUGAUAGCCGCAAAA-GG .....((((((((((--...((((((((..((((((....((..((((--((.(..((((.....))))..).))))))...)))))))))))..)))))...)))))))))-). ( -37.70) >DroVir_CAF1 287221 108 - 1 AUCUGA-----GUGUGUGCGUGUGUGCGUGGCCUGCUUUUGAAUCUGAUGCGCCAGAAUUUGCAUAAUUCUGCUGUCAGAUGACGCAUACGCCGUGUAUGACAGCCGCUAAA-G- ..(((.-----(((..((.((((((((((((....)).....((((((((.((.((((((.....)))))))))))))))).))))))))))))..)))..)))........-.- ( -35.90) >DroPse_CAF1 206704 109 - 1 UCUCU----CUCUGUGUGUUUGUGUGUGUGGCAUGCAAUCGAAGGUGA--CACUGGAAUUUGCAUAAUGCUAUUGUCACCUGACGCAUAUUCCAUGCACUAUAGCCGCGAAAGGG .((((----.((.(((.(((...((((((((.((((......((((((--((.(((.(((.....))).))).))))))))...))))...))))))))...)))))))).)))) ( -37.20) >DroEre_CAF1 217382 106 - 1 UUU----UUUGCGGU--GUGCGUGCGGUUGGUAUGCAAUUGAAGGUGA--CACUGAAAUUUGCAUAAUUCUGUUGUCACAUGUCGCAUACGCCAUGUAUGAUAGCCGCAAAA-GG .((----((((((((--...((((((((..((((((....((..((((--((.(..((((.....))))..).))))))...)))))))))))..)))))...)))))))))-). ( -37.70) >DroMoj_CAF1 349173 107 - 1 --CUGA----UUUGUGUGUGUGUGUGUAUGUCCUGCAAUUGAAGCUGAUGCGCCCAAAUUUGCAUAAUGCUUUUGUCAGAUGACGCAUACGCCGUGUAUGACAGCCGUGAAA-G- --(((.----..((..((.((((((((..((((((((...(((((..(((((........)))))...))))))).)))..)))))))))))))..))...)))........-.- ( -29.00) >DroAna_CAF1 249456 94 - 1 UUU--------------CUGCGUG----UGGCCUGCAAUUGGAGGUGA--CACUGAAAUUUGCAUAAUUCUGUUGUCACAUGUCGCAUACGCCAUGUAUGAUAGCCGCAAGG-GG (((--------------(...(((----(.((((.(....).)))).)--))).))))...........((((..(.(((((.((....)).))))))..))))((....))-.. ( -28.70) >consensus UUUU_______CUGU__GUGCGUGUGGGUGGCAUGCAAUUGAAGGUGA__CACUGAAAUUUGCAUAAUUCUGUUGUCACAUGACGCAUACGCCAUGUAUGAUAGCCGCAAAA_GG ..................((((...........(((((.....((((...)))).....))))).......(((((((((((.((....)).))))..)))))))))))...... (-12.51 = -13.43 + 0.92)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:19:35 2006