
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 14,543,620 – 14,543,764 |
| Length | 144 |
| Max. P | 0.698913 |

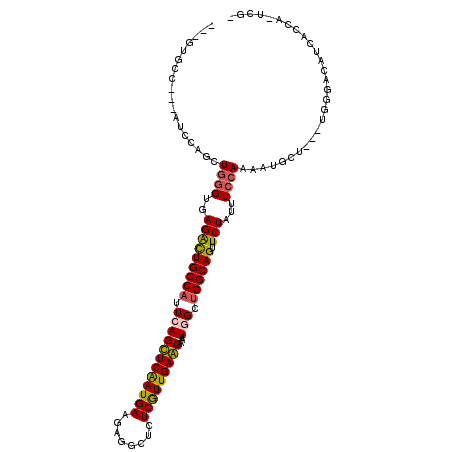
| Location | 14,543,620 – 14,543,726 |
|---|---|
| Length | 106 |
| Sequences | 6 |
| Columns | 115 |
| Reading direction | forward |
| Mean pairwise identity | 82.45 |
| Mean single sequence MFE | -38.40 |
| Consensus MFE | -25.09 |
| Energy contribution | -24.78 |
| Covariance contribution | -0.30 |
| Combinations/Pair | 1.20 |
| Mean z-score | -2.16 |
| Structure conservation index | 0.65 |
| SVM decision value | 0.35 |
| SVM RNA-class probability | 0.698913 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
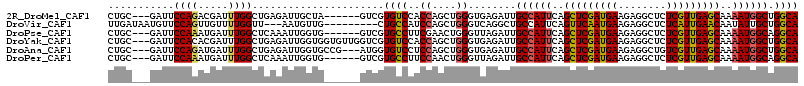
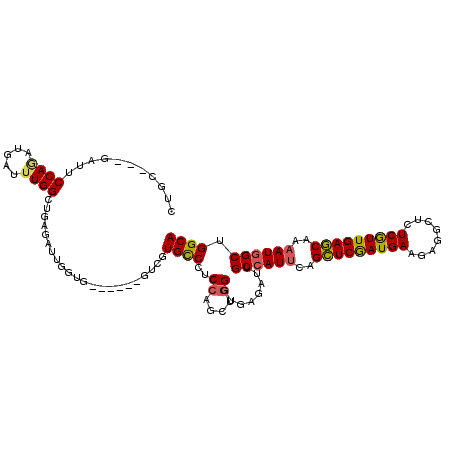
>2R_DroMel_CAF1 14543620 106 + 20766785 CUGC---GAUUCCAGACGAUUUGGCUGAGAUUGCUA------GUCGUGUCCACCAGCUGGGUGAGAUUGCCAUUCAGCUCGAUGAAGAGGCUCUCGUUGAGCAAAAUGGCUGGCA ..((---(((..(((.(......))))..)))))..------(((..(((((((.....)))).))).((((((..(((((((((........)))))))))..)))))).))). ( -40.00) >DroVir_CAF1 225505 103 + 1 UUGAUAAUGUUCCAGUUGUUUUGGUU---AAUGUUG---------CUGCCAUCCAGCUGGGUCAGGCUGCCAUUCAGUUCAAUGAAGAGGCUCUCAUUGAACAAUAUUGCUGGCA .......((..(((((((...((((.---.......---------..))))..)))))))..)).(((((.((...(((((((((........)))))))))...)).)).))). ( -31.00) >DroPse_CAF1 171381 106 + 1 CUGC---GAUUCCAAAUGAUUUGGCUCAAAUUGGUG------GUCGUGCCUUCGAACUGGGUUAGAUUGCCAUUCAGCUCGAUGAAGAGGCUCUCGUUGAGCAAAAUGGCAGGCA ..((---..........(((((((((((..((((.(------(.....)).))))..)))))))))))((((((..(((((((((........)))))))))..))))))..)). ( -38.60) >DroYak_CAF1 189116 112 + 1 CUGC---GAUUCCACACGAUUUGGCUGAGAUUGGUGGUGUUGGUCGUGUCCACCAGCUGGGUGAGAUUGCCAUUCAGCUCGAUGAAGAGGCUCUCGUUGAGCAAAAUGGCUGGCA ...(---(((.((((.((((((.....)))))))))).))))(((..(((((((.....)))).))).((((((..(((((((((........)))))))))..)))))).))). ( -42.80) >DroAna_CAF1 207197 109 + 1 CUGC---GAUUCCAGAUGAUUUGGCUGAGAUUGGUGCCG---AUGGUGUCCUCCAGCUGGGUGAGAUUGCCAUUCAGCUCGAUGAAGAGGCUGUCGUUGAGCAAAAUGGCUGGCA ..((---.........(.(((..((((.((..((..((.---..))..)).))))))..))).)....((((((..(((((((((........)))))))))..))))))..)). ( -38.60) >DroPer_CAF1 173681 106 + 1 CUGC---GAUUCCAAAUGAUUUGGCUCAAAUUGGUG------GUCGUGCCUUCCAACUGGGUUAGAUUGCCAUUCAGCUCGAUGAAGAGGCUCUCGUUGAGCAAAAUGGCAGGCA ..((---..........(((((((((((..((((.(------(.....))..)))).)))))))))))((((((..(((((((((........)))))))))..))))))..)). ( -39.40) >consensus CUGC___GAUUCCAGAUGAUUUGGCUGAGAUUGGUG______GUCGUGCCCUCCAGCUGGGUGAGAUUGCCAUUCAGCUCGAUGAAGAGGCUCUCGUUGAGCAAAAUGGCUGGCA ...........((((.....))))......................((((..((....))........((((((..(((((((((........)))))))))..)))))).)))) (-25.09 = -24.78 + -0.30)
| Location | 14,543,653 – 14,543,764 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 116 |
| Reading direction | forward |
| Mean pairwise identity | 82.37 |
| Mean single sequence MFE | -35.40 |
| Consensus MFE | -24.09 |
| Energy contribution | -23.53 |
| Covariance contribution | -0.55 |
| Combinations/Pair | 1.20 |
| Mean z-score | -1.88 |
| Structure conservation index | 0.68 |
| SVM decision value | 0.28 |
| SVM RNA-class probability | 0.664206 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
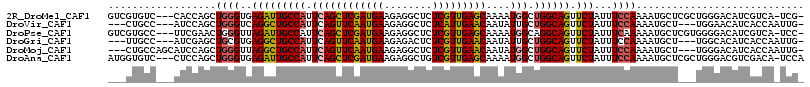
>2R_DroMel_CAF1 14543653 111 + 20766785 GUCGUGUC---CACCAGCUGGGUGAGAUUGCCAUUCAGCUCGAUGAAGAGGCUCUCGUUGAGCAAAAUGGCUGGCAGUUCUAUUUCCAAAAUGCUCGCUGGGACAUCGUCA-UCG- (.((((((---(..((((.((((......((((((..(((((((((........)))))))))..))))))(((.((......)))))....))))))))))))).)).).-...- ( -40.30) >DroVir_CAF1 225538 106 + 1 ---CUGCC---AUCCAGCUGGGUCAGGCUGCCAUUCAGUUCAAUGAAGAGGCUCUCAUUGAACAAUAUUGCUGGCAGUUCUAUUUCCAAAAUGCU---UGGAACAUCACCAAUUG- ---(((((---(..(((((......))))).......(((((((((........)))))))))........))))))......((((((.....)---)))))............- ( -30.60) >DroPse_CAF1 171414 111 + 1 GUCGUGCC---UUCGAACUGGGUUAGAUUGCCAUUCAGCUCGAUGAAGAGGCUCUCGUUGAGCAAAAUGGCAGGCAGUUCUAUUUCAAAAAUGCUCGUGGGGACAUCGUCA-UCC- ...((.((---(.(((.(((((.((((((((((((..(((((((((........)))))))))..)))))))).....)))).)))).....).))).))).)).......-...- ( -34.50) >DroGri_CAF1 202376 106 + 1 ---UUGCC---AUCGAGCUGCGUGAGGCUGCCAUUCAGUUCAAUGAAGAGACUCUCGUUGAACAAUAUUGCUGGCAGUUCUAUUUCCAAAAUGCU---UGGCACAUCACCAAUUG- ---.((((---(...(((..(....)(((((((..(((((((((((........))))))))).....)).)))))))..............)))---)))))............- ( -28.80) >DroMoj_CAF1 283835 109 + 1 ---CUGCCAGCAUCCAGCUGGGUUAGGCUGCCAUUCAGUUCAAUGAAGAGGCUCUCGUUGAACAAUAUGGCUGGCAGUUCUAUUUCCAAAAUGCU---UGGGACAUCACCAAUUG- ---((((((((...(((((......))))).(((...(((((((((........)))))))))...)))))))))))......((((((.....)---)))))............- ( -37.60) >DroAna_CAF1 207233 112 + 1 AUGGUGUC---CUCCAGCUGGGUGAGAUUGCCAUUCAGCUCGAUGAAGAGGCUGUCGUUGAGCAAAAUGGCUGGCAGUUCUAUUUCCAAAAUGCUCGCUGGGACGUCGACA-UCCA ..((((((---..(...((.((((((...((((((..(((((((((........)))))))))..))))))(((.((......))))).....)))))).))..)..))))-)).. ( -40.60) >consensus ___GUGCC___AUCCAGCUGGGUGAGACUGCCAUUCAGCUCAAUGAAGAGGCUCUCGUUGAACAAAAUGGCUGGCAGUUCUAUUUCCAAAAUGCU___UGGGACAUCACCA_UCG_ ..................((((..(((((((((.((((((((((((........)))))))))....))).)))))).)))...))))............................ (-24.09 = -23.53 + -0.55)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:19:23 2006