
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 14,524,173 – 14,524,306 |
| Length | 133 |
| Max. P | 0.991679 |

| Location | 14,524,173 – 14,524,267 |
|---|---|
| Length | 94 |
| Sequences | 6 |
| Columns | 100 |
| Reading direction | forward |
| Mean pairwise identity | 81.97 |
| Mean single sequence MFE | -29.80 |
| Consensus MFE | -25.43 |
| Energy contribution | -25.10 |
| Covariance contribution | -0.33 |
| Combinations/Pair | 1.12 |
| Mean z-score | -2.85 |
| Structure conservation index | 0.85 |
| SVM decision value | 1.54 |
| SVM RNA-class probability | 0.962743 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
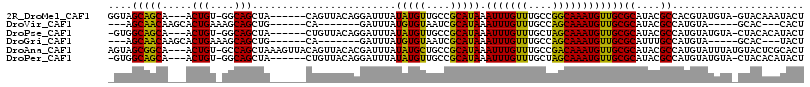
>2R_DroMel_CAF1 14524173 94 + 20766785 AA------CAGCAAACCUCAGUGGCAACCGAAUUGGUAGCAGCAACUGUGGCAGCUACAGUUACAGGAUUUAUAUGUUGCCGCAUAAAUUUGUUUGCCGG ..------.............((((((.((((((.(..((((((.(((((((.......)))))))........))))))..)...)))))).)))))). ( -28.50) >DroPse_CAF1 153259 77 + 1 ----------------------GGCAACUGCGUU-GUGGCAGCAACUGUGGCAGCUACUGUUACAGGAUUUAUAUGUUGCCGCAUAAAUUUGUUUGCUAG ----------------------((((((.....(-(((((((((.(((((((((...)))))))))........)))))))))).......).))))).. ( -28.00) >DroSec_CAF1 156468 100 + 1 AACAACAACGGCAAACCUCAGUGGCAACCGAAUUGGUAGCAGCAACUGUGGCAGCUACAGUUACAGGAUUUAUAUGUUGCCGCAUAAAUUUGUUUGCCGG ........((((((((....((.((.(((.....))).)).))...(((((((((...((((....)))).....))))))))).......)))))))). ( -32.90) >DroSim_CAF1 157591 100 + 1 AACAACAACGGCAAACCUCAGUGGCAACCGAAUUGGUAGCAGCAACUGUGGCAGCUACAGUUACAGGAUUUAUAUGUUGCCGCAUAAAUUUGUUUGCCGG ........((((((((....((.((.(((.....))).)).))...(((((((((...((((....)))).....))))))))).......)))))))). ( -32.90) >DroYak_CAF1 160526 94 + 1 --CAAAAAC----UACAUCAGUGGCAACCGAAUUGGUAGCAGCAACUGUGGCAGCUACAGUUACAGGAUUUAUAUGUUGCCGCAUAAAUUUGUUUGCCGG --.......----........((((((.((((((.(..((((((.(((((((.......)))))))........))))))..)...)))))).)))))). ( -28.50) >DroPer_CAF1 155465 77 + 1 ----------------------GGCAACUGCGUU-GUGGCAGCAACUGUGGCAGCUACUGUUACAGGAUUUAUAUGUUGCCGCAUAAAUUUGUUUGCUAG ----------------------((((((.....(-(((((((((.(((((((((...)))))))))........)))))))))).......).))))).. ( -28.00) >consensus ________C____AACCUCAGUGGCAACCGAAUUGGUAGCAGCAACUGUGGCAGCUACAGUUACAGGAUUUAUAUGUUGCCGCAUAAAUUUGUUUGCCGG ......................(((((.((((((.((.((((((.(((((((.......)))))))........)))))).))...)))))).))))).. (-25.43 = -25.10 + -0.33)


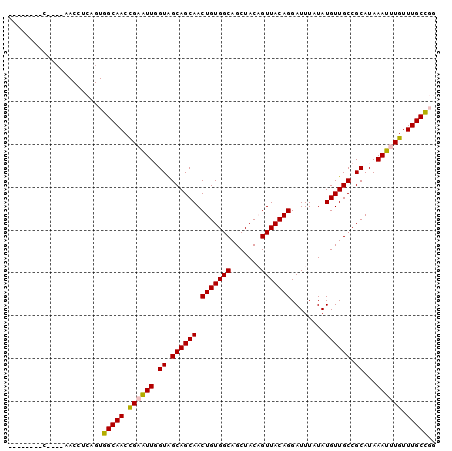
| Location | 14,524,173 – 14,524,267 |
|---|---|
| Length | 94 |
| Sequences | 6 |
| Columns | 100 |
| Reading direction | reverse |
| Mean pairwise identity | 81.97 |
| Mean single sequence MFE | -25.62 |
| Consensus MFE | -18.37 |
| Energy contribution | -18.70 |
| Covariance contribution | 0.33 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.42 |
| Structure conservation index | 0.72 |
| SVM decision value | 0.84 |
| SVM RNA-class probability | 0.865019 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 14524173 94 - 20766785 CCGGCAAACAAAUUUAUGCGGCAACAUAUAAAUCCUGUAACUGUAGCUGCCACAGUUGCUGCUACCAAUUCGGUUGCCACUGAGGUUUGCUG------UU .((((((((........(((((.(((((((.....))))..))).)))))..((((.((....(((.....))).)).))))..))))))))------.. ( -29.20) >DroPse_CAF1 153259 77 - 1 CUAGCAAACAAAUUUAUGCGGCAACAUAUAAAUCCUGUAACAGUAGCUGCCACAGUUGCUGCCAC-AACGCAGUUGCC---------------------- ...(((..........)))((((((.((((.....)))).(((((((((...)))))))))....-......))))))---------------------- ( -20.10) >DroSec_CAF1 156468 100 - 1 CCGGCAAACAAAUUUAUGCGGCAACAUAUAAAUCCUGUAACUGUAGCUGCCACAGUUGCUGCUACCAAUUCGGUUGCCACUGAGGUUUGCCGUUGUUGUU .((((((((........(((((.(((((((.....))))..))).)))))..((((.((....(((.....))).)).))))..))))))))........ ( -31.40) >DroSim_CAF1 157591 100 - 1 CCGGCAAACAAAUUUAUGCGGCAACAUAUAAAUCCUGUAACUGUAGCUGCCACAGUUGCUGCUACCAAUUCGGUUGCCACUGAGGUUUGCCGUUGUUGUU .((((((((........(((((.(((((((.....))))..))).)))))..((((.((....(((.....))).)).))))..))))))))........ ( -31.40) >DroYak_CAF1 160526 94 - 1 CCGGCAAACAAAUUUAUGCGGCAACAUAUAAAUCCUGUAACUGUAGCUGCCACAGUUGCUGCUACCAAUUCGGUUGCCACUGAUGUA----GUUUUUG-- ..(((((....(((((((.(....).))))))).(((.....(((((.((.......)).))))).....)))))))).........----.......-- ( -21.50) >DroPer_CAF1 155465 77 - 1 CUAGCAAACAAAUUUAUGCGGCAACAUAUAAAUCCUGUAACAGUAGCUGCCACAGUUGCUGCCAC-AACGCAGUUGCC---------------------- ...(((..........)))((((((.((((.....)))).(((((((((...)))))))))....-......))))))---------------------- ( -20.10) >consensus CCGGCAAACAAAUUUAUGCGGCAACAUAUAAAUCCUGUAACUGUAGCUGCCACAGUUGCUGCUACCAAUUCGGUUGCCACUGAGGUU____G________ ..((((((((.(((((((.(....).)))))))..)))....(((((.((....)).)))))...........)))))...................... (-18.37 = -18.70 + 0.33)

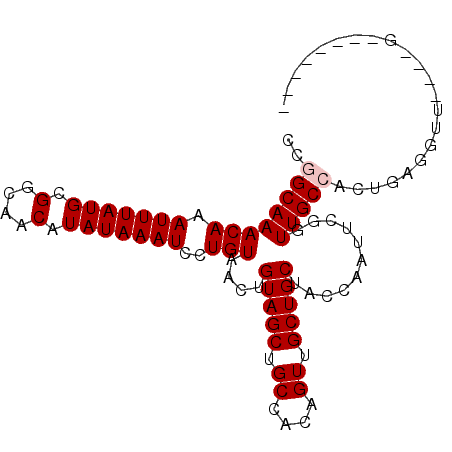
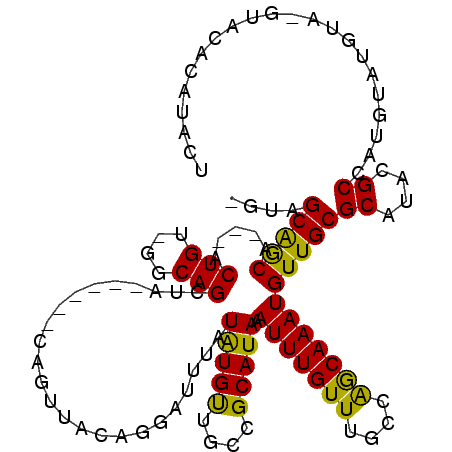
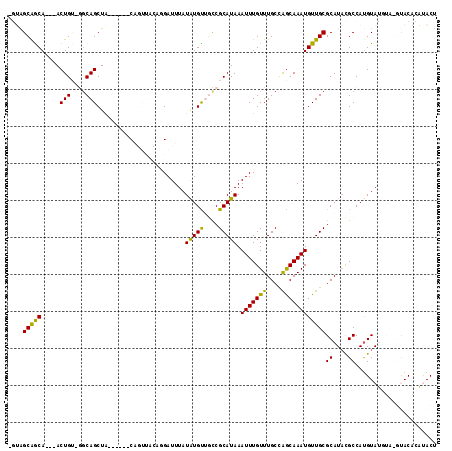
| Location | 14,524,201 – 14,524,306 |
|---|---|
| Length | 105 |
| Sequences | 6 |
| Columns | 116 |
| Reading direction | forward |
| Mean pairwise identity | 76.97 |
| Mean single sequence MFE | -31.75 |
| Consensus MFE | -15.65 |
| Energy contribution | -14.57 |
| Covariance contribution | -1.08 |
| Combinations/Pair | 1.27 |
| Mean z-score | -3.05 |
| Structure conservation index | 0.49 |
| SVM decision value | 2.28 |
| SVM RNA-class probability | 0.991679 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 14524201 105 + 20766785 GGUAGCAGCA---ACUGU-GGCAGCUA------CAGUUACAGGAUUUAUAUGUUGCCGCAUAAAUUUGUUUGCCGGCAAAUGUUGCGCAUACGCCACGUAUGUA-GUACAAAUACU .(((((((((---.((((-(((.....------..)))))))..........((((((((..........)).)))))).))))))(((((((...))))))).-.)))....... ( -32.80) >DroVir_CAF1 200303 92 + 1 ---AGCAACAAGCACUGAAAGCAGCUG------CA-------GAUUUAUGUGUAAUCGCAUAAAUUUGUUUGCCAGCAAAUGUUGCGCAUACGCCAUGUA-----GCAC---CACU ---.((((((....(((...((((..(------((-------((((((((((....))))))))))))))))))))....))))))((.(((.....)))-----))..---.... ( -29.30) >DroPse_CAF1 153271 104 + 1 -GUGGCAGCA---ACUGU-GGCAGCUA------CUGUUACAGGAUUUAUAUGUUGCCGCAUAAAUUUGUUUGCUAGCAAAUGUUGCGCAUACGCCAUGUAUGUA-CUACACAUACU -(((((....---.((((-(((((...------))))))))).........(((((.(((...(((((((....)))))))..)))))).)))))))((((((.-....)))))). ( -35.30) >DroGri_CAF1 178535 92 + 1 ---AGCAACAAGCACUGAAAGCAGCUG------CA-------GAUUUAUGUGUAAUCGCAUAAAUUUGUUUGCCAGCAAAUGUUGCGCAUUUGCCAUGUA-----GCAC---UACU ---.((.(((..........((((..(------((-------((((((((((....)))))))))))))))))..(((((((.....)))))))..))).-----))..---.... ( -29.60) >DroAna_CAF1 173695 112 + 1 AGUAGCGGCA---ACUGU-GCCAGCUAAAGUUACAGUUACACGAUUUAUAUGCUGCCGCAUAAAUUUGUUUGCCGACAAAUGUUGCGCAUACGCCAUGUAUUUAUGUACUCGCACU (((.((((((---(((((-(.(.......).)))))))..........(((((....))))).....((.(((((((....)))).))).)))))..((((....))))..))))) ( -28.20) >DroPer_CAF1 155477 104 + 1 -GUGGCAGCA---ACUGU-GGCAGCUA------CUGUUACAGGAUUUAUAUGUUGCCGCAUAAAUUUGUUUGCUAGCAAAUGUUGCGCAUACGCCAUGUAUGUA-CUACACAUACU -(((((....---.((((-(((((...------))))))))).........(((((.(((...(((((((....)))))))..)))))).)))))))((((((.-....)))))). ( -35.30) >consensus _GUAGCAGCA___ACUGU_GGCAGCUA______CAGUUACAGGAUUUAUAUGUUGCCGCAUAAAUUUGUUUGCCAGCAAAUGUUGCGCAUACGCCAUGUAUGUA_GUACACAUACU ....(((((.....(((....)))........................(((((....))))).(((((((....))))))))))))((....))...................... (-15.65 = -14.57 + -1.08)
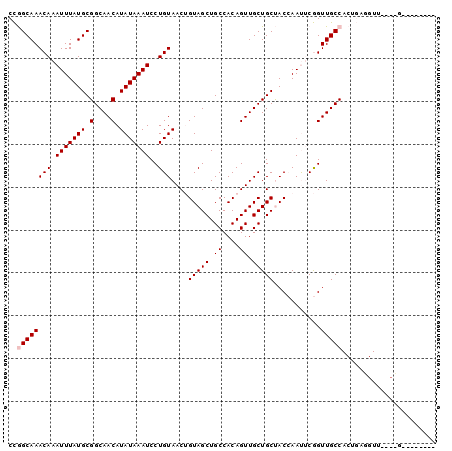
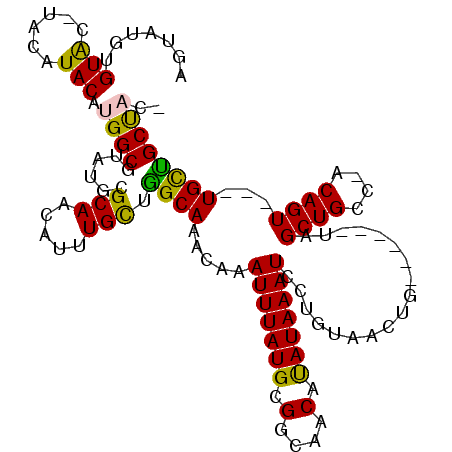
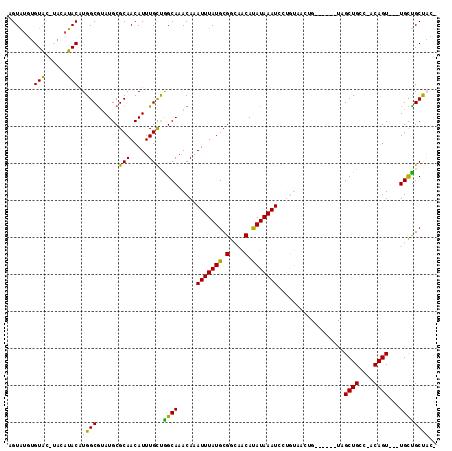
| Location | 14,524,201 – 14,524,306 |
|---|---|
| Length | 105 |
| Sequences | 6 |
| Columns | 116 |
| Reading direction | reverse |
| Mean pairwise identity | 76.97 |
| Mean single sequence MFE | -26.58 |
| Consensus MFE | -15.59 |
| Energy contribution | -14.53 |
| Covariance contribution | -1.05 |
| Combinations/Pair | 1.27 |
| Mean z-score | -1.67 |
| Structure conservation index | 0.59 |
| SVM decision value | 0.47 |
| SVM RNA-class probability | 0.749227 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 14524201 105 - 20766785 AGUAUUUGUAC-UACAUACGUGGCGUAUGCGCAACAUUUGCCGGCAAACAAAUUUAUGCGGCAACAUAUAAAUCCUGUAACUG------UAGCUGCC-ACAGU---UGCUGCUACC ((((....)))-)......(((((((.((((((.....)))..))).))..(((((((.(....).)))))))...(((((((------(.......-)))))---))).))))). ( -25.70) >DroVir_CAF1 200303 92 - 1 AGUG---GUGC-----UACAUGGCGUAUGCGCAACAUUUGCUGGCAAACAAAUUUAUGCGAUUACACAUAAAUC-------UG------CAGCUGCUUUCAGUGCUUGUUGCU--- ....---..((-----(((.....))).))((((((..((((((((..((.(((((((.(....).))))))).-------))------....)))...)))))..)))))).--- ( -22.30) >DroPse_CAF1 153271 104 - 1 AGUAUGUGUAG-UACAUACAUGGCGUAUGCGCAACAUUUGCUAGCAAACAAAUUUAUGCGGCAACAUAUAAAUCCUGUAACAG------UAGCUGCC-ACAGU---UGCUGCCAC- ((((.((((.(-..(((((.....)))))..).)))).)))).....(((.(((((((.(....).)))))))..)))..(((------((((((..-.))))---)))))....- ( -29.50) >DroGri_CAF1 178535 92 - 1 AGUA---GUGC-----UACAUGGCAAAUGCGCAACAUUUGCUGGCAAACAAAUUUAUGCGAUUACACAUAAAUC-------UG------CAGCUGCUUUCAGUGCUUGUUGCU--- .(((---.(((-----(....))))..)))((((((..((((((((..((.(((((((.(....).))))))).-------))------....)))...)))))..)))))).--- ( -24.20) >DroAna_CAF1 173695 112 - 1 AGUGCGAGUACAUAAAUACAUGGCGUAUGCGCAACAUUUGUCGGCAAACAAAUUUAUGCGGCAGCAUAUAAAUCGUGUAACUGUAACUUUAGCUGGC-ACAGU---UGCCGCUACU .....(((((((....(((((((.((((((((..(((((((......))))....)))..)).))))))...)))))))..))).))))((((.(((-(....---)))))))).. ( -28.30) >DroPer_CAF1 155477 104 - 1 AGUAUGUGUAG-UACAUACAUGGCGUAUGCGCAACAUUUGCUAGCAAACAAAUUUAUGCGGCAACAUAUAAAUCCUGUAACAG------UAGCUGCC-ACAGU---UGCUGCCAC- ((((.((((.(-..(((((.....)))))..).)))).)))).....(((.(((((((.(....).)))))))..)))..(((------((((((..-.))))---)))))....- ( -29.50) >consensus AGUAUGUGUAC_UACAUACAUGGCGUAUGCGCAACAUUUGCUGGCAAACAAAUUUAUGCGGCAACAUAUAAAUCCUGUAACUG______UAGCUGCC_ACAGU___UGCUGCUAC_ .......(((......))).((((......(((.....))).((((.....(((((((.(....).)))))))..................((((....))))...)))))))).. (-15.59 = -14.53 + -1.05)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:19:13 2006