
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 14,521,100 – 14,521,220 |
| Length | 120 |
| Max. P | 0.981077 |

| Location | 14,521,100 – 14,521,220 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 93.90 |
| Mean single sequence MFE | -51.40 |
| Consensus MFE | -44.86 |
| Energy contribution | -44.75 |
| Covariance contribution | -0.11 |
| Combinations/Pair | 1.06 |
| Mean z-score | -2.30 |
| Structure conservation index | 0.87 |
| SVM decision value | 1.88 |
| SVM RNA-class probability | 0.981077 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
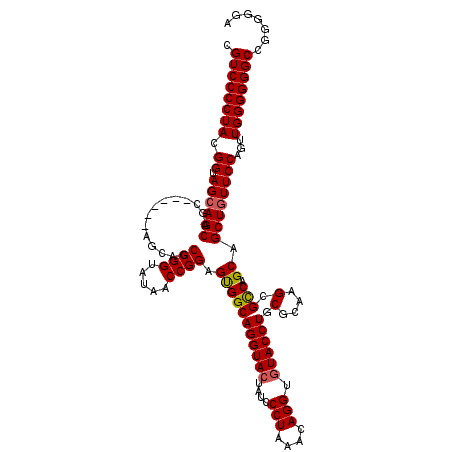
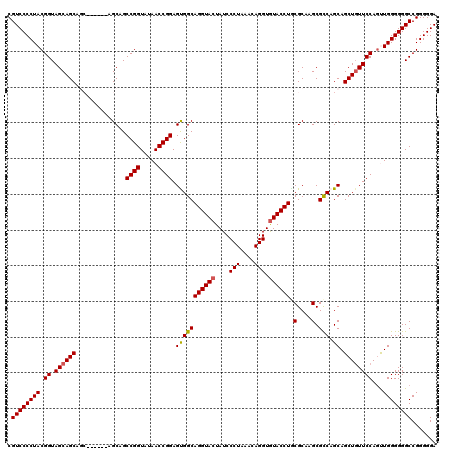
>2R_DroMel_CAF1 14521100 120 + 20766785 CGUCCCCUACGGUAGCAGCAGCAGUGGCAGCAGCCGGUAUAACCGGAGCGGCAGGUACUAUCCCUAAACAGGUGUACCUGCGCAAGCGCCAGCAGCUGUUCCAGCUGGGGGGCCGGGGGA .(((((((((((.((((((.((..((((.((..((((.....)))).(((.(((((((....(((....))).))))))))))..)))))))).)))))))).).))))))))....... ( -59.40) >DroVir_CAF1 196518 114 + 1 CGUCCCCUACGGUAGCAGCAGC------AGCAGCCGGUAUAACCGGAGUGGCAGGUACUAUCCCUAAACAGGUGUACCUGCGCAAGCGCCAGCAGCUGUUCCAGUUGGGGGGCCGGGGGA .(((((((((((.((((((.((------.....((((.....))))..((((((((((....(((....))).))))))((....)))))))).)))))))).).))))))))....... ( -54.40) >DroGri_CAF1 175446 111 + 1 CGUCCCCUACGGUAGCAGC---------AGCAGCCGGUAUAACCGGAGUGGCAGGUACUAUCCCUAAACAGGUGUACCUACGCAAGCGCCAGCAGCUGUUCCAGUUGGGGGGCCGGGGGA .(((((((((((.((((((---------.((..((((.....))))..((((((((((....(((....))).)))))).(....).)))))).)))))))).).))))))))....... ( -50.20) >DroWil_CAF1 232933 114 + 1 CGUCCCCUACGGUAGCAGCAGC------AGCAGCCGGUAUAACCGGAGUGGCAGGUACUAUCCCUAAACAGGUGUACCUACGCAAGCGUCAGCAGCUUUUCCAAUUGGGGGGCCGGGGGA ..(((((..(((((((.((...------.((..((((.....)))).(((..((((((....(((....))).)))))).)))..))....)).)))(..((....))..)))))))))) ( -43.60) >DroMoj_CAF1 243531 114 + 1 CGUCCCCUACGGUAGCAGCAGC------AGCAGCCGGUAUAACCGGAGUGGCAGGUACUAUCCCUAAACAGGUAUACCUGCGCAAGCGCCAGCAGCUGUUCCAGUUGGGGGGCCGGGGGA .(((((((((((.((((((.((------.....((((.....))))..(((((((((.((((........)))))))))((....)))))))).)))))))).).))))))))....... ( -51.60) >DroAna_CAF1 168452 111 + 1 CGUCCCCUACGGUAGCAGCA---------GCAGCCGGUAUAACCGGAGCGGCAGGUACUAUCCCUAAACAGGUGUACCUUCGCAAGCGUCAGCAGCUGUUCCAGCUGGGGGGCCGGGGGA .(((((((((((.((((((.---------((..((((.....)))).((((.((((((....(((....))).))))))))))........)).)))))))).).))))))))....... ( -49.20) >consensus CGUCCCCUACGGUAGCAGCAGC______AGCAGCCGGUAUAACCGGAGUGGCAGGUACUAUCCCUAAACAGGUGUACCUGCGCAAGCGCCAGCAGCUGUUCCAGUUGGGGGGCCGGGGGA .((((((((.((.((((((..............((((.....)))).(((((((((((....(((....))).)))))).(....).))).)).))))))))...))))))))....... (-44.86 = -44.75 + -0.11)

| Location | 14,521,100 – 14,521,220 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 93.90 |
| Mean single sequence MFE | -44.67 |
| Consensus MFE | -41.25 |
| Energy contribution | -40.75 |
| Covariance contribution | -0.50 |
| Combinations/Pair | 1.11 |
| Mean z-score | -0.97 |
| Structure conservation index | 0.92 |
| SVM decision value | 0.02 |
| SVM RNA-class probability | 0.544978 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
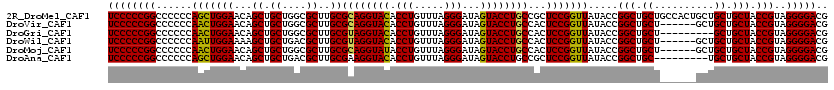
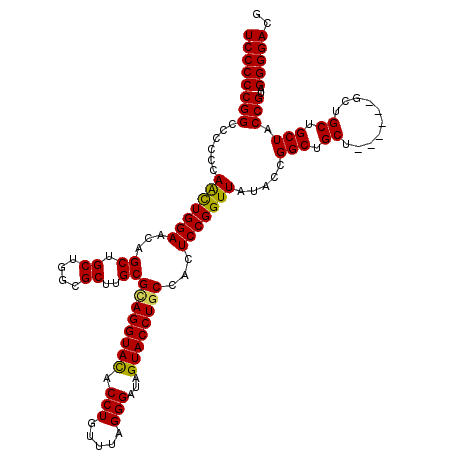
>2R_DroMel_CAF1 14521100 120 - 20766785 UCCCCCGGCCCCCCAGCUGGAACAGCUGCUGGCGCUUGCGCAGGUACACCUGUUUAGGGAUAGUACCUGCCGCUCCGGUUAUACCGGCUGCUGCCACUGCUGCUGCUACCGUAGGGGACG (((((((((......)))))..((((.(.((((((..((((((((((.(((.....)))...))))))).))).((((.....))))..)).))))).))))((((....)))))))).. ( -52.30) >DroVir_CAF1 196518 114 - 1 UCCCCCGGCCCCCCAACUGGAACAGCUGCUGGCGCUUGCGCAGGUACACCUGUUUAGGGAUAGUACCUGCCACUCCGGUUAUACCGGCUGCU------GCUGCUGCUACCGUAGGGGACG ((((((((....((....))..((((.((.(((...((.((((((((.(((.....)))...))))))))))..((((.....))))..)))------)).))))...)))..))))).. ( -46.20) >DroGri_CAF1 175446 111 - 1 UCCCCCGGCCCCCCAACUGGAACAGCUGCUGGCGCUUGCGUAGGUACACCUGUUUAGGGAUAGUACCUGCCACUCCGGUUAUACCGGCUGCU---------GCUGCUACCGUAGGGGACG ((((((((....((....))..((((.((.(((...((.((((((((.(((.....)))...))))))))))...(((.....)))))))).---------))))...)))..))))).. ( -42.00) >DroWil_CAF1 232933 114 - 1 UCCCCCGGCCCCCCAAUUGGAAAAGCUGCUGACGCUUGCGUAGGUACACCUGUUUAGGGAUAGUACCUGCCACUCCGGUUAUACCGGCUGCU------GCUGCUGCUACCGUAGGGGACG ((((((((....((....))...(((.((....((..((((((((((.(((.....)))...))))))))....((((.....))))..)).------)).)).))).)))..))))).. ( -40.90) >DroMoj_CAF1 243531 114 - 1 UCCCCCGGCCCCCCAACUGGAACAGCUGCUGGCGCUUGCGCAGGUAUACCUGUUUAGGGAUAGUACCUGCCACUCCGGUUAUACCGGCUGCU------GCUGCUGCUACCGUAGGGGACG ((((((((....((....))..((((.((.(((...((.((((((((.(((.....)))...))))))))))..((((.....))))..)))------)).))))...)))..))))).. ( -44.70) >DroAna_CAF1 168452 111 - 1 UCCCCCGGCCCCCCAGCUGGAACAGCUGCUGACGCUUGCGAAGGUACACCUGUUUAGGGAUAGUACCUGCCGCUCCGGUUAUACCGGCUGC---------UGCUGCUACCGUAGGGGACG (((((((((.....(((((...)))))))))(((...(((.((((((.(((.....)))...))))))((.((.((((.....))))..))---------.)))))...))).))))).. ( -41.90) >consensus UCCCCCGGCCCCCCAACUGGAACAGCUGCUGGCGCUUGCGCAGGUACACCUGUUUAGGGAUAGUACCUGCCACUCCGGUUAUACCGGCUGCU______GCUGCUGCUACCGUAGGGGACG ((((((((......(((((((...((.((....))..))((((((((.(((.....)))...))))))))...))))))).....(((.((..........)).))).)))..))))).. (-41.25 = -40.75 + -0.50)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:19:05 2006