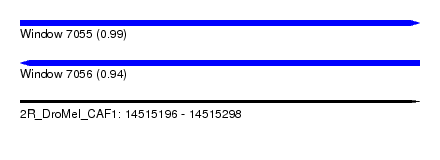
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 14,515,196 – 14,515,298 |
| Length | 102 |
| Max. P | 0.987145 |

| Location | 14,515,196 – 14,515,298 |
|---|---|
| Length | 102 |
| Sequences | 6 |
| Columns | 110 |
| Reading direction | forward |
| Mean pairwise identity | 72.89 |
| Mean single sequence MFE | -31.02 |
| Consensus MFE | -12.75 |
| Energy contribution | -14.78 |
| Covariance contribution | 2.03 |
| Combinations/Pair | 1.26 |
| Mean z-score | -2.58 |
| Structure conservation index | 0.41 |
| SVM decision value | 2.07 |
| SVM RNA-class probability | 0.987145 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
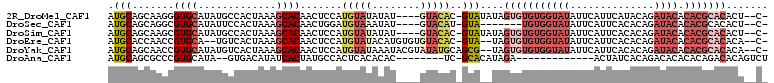
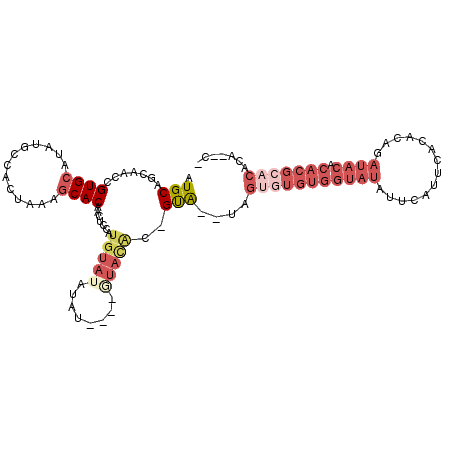
>2R_DroMel_CAF1 14515196 102 + 20766785 -G--AGUGUGCGUGUGUAUCUGUAUGAAUGAAUAUACCACACACUAUAUAC-GUGUAC----AUAUAUACAUGGAGUUGUGCUUUAGUGGCAUAUGCACCCUUGCUGCAU -(--((.((((((((((...(((((......))))).....(((((....(-(((((.----.....))))))(((.....)))))))))))))))))).)))....... ( -27.60) >DroSec_CAF1 147384 95 + 1 -G--AGUGUGCGUGUGUAUCUGUGUGAAUGAAUAUACCACA-------UAC-AUGUAC----AUAUUUACAUCCAGUUGUGCUUUAGUGGAAUAUGCACGCCUGCUGCAU -.--.((((((((((((((.((((((..((.......))))-------)))-).))))----)))).....((((.(........).))))....))))))......... ( -25.90) >DroSim_CAF1 148511 102 + 1 -G--AGUGUGCGUGUGUAUCUGUGUGAAUGAAUAUACCACACACUAUAUAC-GUGUAC----AUAUAUACAUGGAGUUGUGCUUUAGUGGCAUAUGCACGCUUGCUGCAU -(--(((((((((((((....(((((...........)))))((((....(-(((((.----.....))))))(((.....))))))).))))))))))))))....... ( -32.90) >DroEre_CAF1 152827 102 + 1 -G--UGUGUGCGUGUGUAUCUGUGUGAAUGAAUAUACCACACACUA--UAC-GUGUACACACAUGUAUACAUGGAGUUGUGCUUUAGUGACA--UGCACGGUUGGUGCAU -(--((((((((((((((..((((((...........)))))).))--)))-))))))))))((((.(((.(((((.....)))))))))))--)((((.....)))).. ( -37.40) >DroYak_CAF1 151088 105 + 1 -G--UGUGUGCGUGUGUAUCUGUGUGAAUGAAUAUACCACACACUA--CGCUGCAUAUACGUAUUUAUACAUGGAGUUGUGCUUUAGUGACAUAUGCACGGUUGCUGCAU -(--((((((((.(((((..((((((...........)))))).))--))))))))))))(((....)))(((.((((((((.............)))))...))).))) ( -32.42) >DroAna_CAF1 161632 86 + 1 AGACUGUGUCUGUGUGUGUCUGUGAUAGU-------------UCUAUGUGC-GA--------GUGUGUGAGUGGCAUAGUGAUAUGUCAC--UAUGCACGGGCGCUGCAU .(...(((((((((((((...((((((..-------------((..(((((-.(--------.(.....).).)))))..))..))))))--)))))))))))))..).. ( -29.90) >consensus _G__AGUGUGCGUGUGUAUCUGUGUGAAUGAAUAUACCACACACUA__UAC_GUGUAC____AUAUAUACAUGGAGUUGUGCUUUAGUGGCAUAUGCACGCUUGCUGCAU .......((((((((((....(((((...........))))).........................(((.(((((.....))))))))))))))))))........... (-12.75 = -14.78 + 2.03)


| Location | 14,515,196 – 14,515,298 |
|---|---|
| Length | 102 |
| Sequences | 6 |
| Columns | 110 |
| Reading direction | reverse |
| Mean pairwise identity | 72.89 |
| Mean single sequence MFE | -22.61 |
| Consensus MFE | -9.04 |
| Energy contribution | -11.71 |
| Covariance contribution | 2.67 |
| Combinations/Pair | 1.22 |
| Mean z-score | -2.30 |
| Structure conservation index | 0.40 |
| SVM decision value | 1.32 |
| SVM RNA-class probability | 0.941616 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 14515196 102 - 20766785 AUGCAGCAAGGGUGCAUAUGCCACUAAAGCACAACUCCAUGUAUAUAU----GUACAC-GUAUAUAGUGUGUGGUAUAUUCAUUCAUACAGAUACACACGCACACU--C- .(((.....(((((.(((((((((....((((.((.....))((((((----(....)-)))))).))))))))))))).))))).....(.....)..)))....--.- ( -24.90) >DroSec_CAF1 147384 95 - 1 AUGCAGCAGGCGUGCAUAUUCCACUAAAGCACAACUGGAUGUAAAUAU----GUACAU-GUA-------UGUGGUAUAUUCAUUCACACAGAUACACACGCACACU--C- .(((.......((((((((((((............)))).....))))----)))).(-(((-------(.((((..........)).)).)))))...)))....--.- ( -17.80) >DroSim_CAF1 148511 102 - 1 AUGCAGCAAGCGUGCAUAUGCCACUAAAGCACAACUCCAUGUAUAUAU----GUACAC-GUAUAUAGUGUGUGGUAUAUUCAUUCACACAGAUACACACGCACACU--C- .........(((((.(((((((((....((((.((.....))((((((----(....)-)))))).)))))))))))))....((.....))....))))).....--.- ( -24.80) >DroEre_CAF1 152827 102 - 1 AUGCACCAACCGUGCA--UGUCACUAAAGCACAACUCCAUGUAUACAUGUGUGUACAC-GUA--UAGUGUGUGGUAUAUUCAUUCACACAGAUACACACGCACACA--C- ((((((.....)))))--).........((.........(((((((....))))))).-(((--(..(((((((.........))))))).))))....)).....--.- ( -27.00) >DroYak_CAF1 151088 105 - 1 AUGCAGCAACCGUGCAUAUGUCACUAAAGCACAACUCCAUGUAUAAAUACGUAUAUGCAGCG--UAGUGUGUGGUAUAUUCAUUCACACAGAUACACACGCACACA--C- ((((.(((...((((.............))))......(((((....)))))...))).)))--).(((((((((((..............)))).)))))))...--.- ( -24.96) >DroAna_CAF1 161632 86 - 1 AUGCAGCGCCCGUGCAUA--GUGACAUAUCACUAUGCCACUCACACAC--------UC-GCACAUAGA-------------ACUAUCACAGACACACACAGACACAGUCU .(((((.(...(((((((--((((....)))))))).))).....).)--------).-)))......-------------........((((.............)))) ( -16.22) >consensus AUGCAGCAACCGUGCAUAUGCCACUAAAGCACAACUCCAUGUAUAUAU____GUACAC_GUA__UAGUGUGUGGUAUAUUCAUUCACACAGAUACACACGCACACA__C_ .(((.......((((.............)))).......(((((........)))))..)))....(((((((((((..............)))).)))))))....... ( -9.04 = -11.71 + 2.67)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:19:02 2006