
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 14,387,861 – 14,387,968 |
| Length | 107 |
| Max. P | 0.987842 |

| Location | 14,387,861 – 14,387,968 |
|---|---|
| Length | 107 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | forward |
| Mean pairwise identity | 77.74 |
| Mean single sequence MFE | -38.28 |
| Consensus MFE | -23.18 |
| Energy contribution | -23.52 |
| Covariance contribution | 0.33 |
| Combinations/Pair | 1.10 |
| Mean z-score | -2.88 |
| Structure conservation index | 0.61 |
| SVM decision value | 2.10 |
| SVM RNA-class probability | 0.987842 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
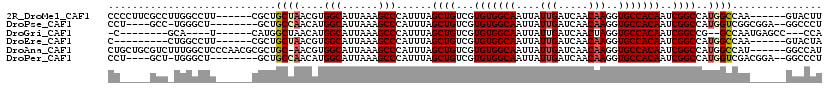
>2R_DroMel_CAF1 14387861 107 + 20766785 CCCCUUCGCCUUGGCCUU------CGCUGCUAACGUGGCAUUAAAGCCCAUUUAGCUGUCGUGUGGCAAUUAUUGAUCAACAAGGUGCCACAAUCGGCCAUGGCCAA------GUACUU ...(((.(((.(((((..------(((.(((((...(((......)))...))))).).))(((((((....(((.....)))..)))))))...))))).))).))------)..... ( -39.50) >DroPse_CAF1 18927 104 + 1 CCU----GCC-UGGGCU--------GCUGCCAACAUGGCAUUAAAGCCCAUUUAGCUGUCGUGUGGCAAUUAUUGAUCAACAAGGUGCCACAAUCGGCCAUGGUCGGCGGA--GGCCCU ...----...-.(((((--------.(((((..((((((.....(((.......)))....(((((((....(((.....)))..)))))))....))))))...))))).--))))). ( -43.40) >DroGri_CAF1 28946 95 + 1 -C--------GCA----U------CAUGGCUAACAUGGCAUUAAAGCCCAUUUAGCUGUCGUGUGGCAAUUAUUGAUCAACUAGGUGCCACAAUCGGCCG--GCCAAUGAGCC---CCA -.--------((.----(------(((((((.....(((......)))......((((...(((((((.(((.(.....).))).)))))))..)))).)--))).)))))).---... ( -31.20) >DroEre_CAF1 21787 98 + 1 C---------CUGGCCUU------CGCUGCUAACGUGGCAUUAAAGCCCAUUUAGCUGUCGUGUGGCAAUUAUUGAUCAACAAGGUGCCACAAUCGGCCAUGGCCAA------GUACUA .---------.(((((..------.((..(....)..))...............((((...(((((((....(((.....)))..)))))))..))))...))))).------...... ( -34.90) >DroAna_CAF1 24864 112 + 1 CUGCUGCGUCUUUGGCUCCCAACGCGCUGC-AACGUGGCAUUAAAGCCCAUUUAGCUGUCGUGUGGCAAUUAUUGAUCAACAAGGUGCCACAAUCGGCCAUGGCCAU------GGCCAU .(((.((((..((((...)))).)))).))-)..(.(((......)))).....((((...(((((((....(((.....)))..)))))))..)))).(((((...------.))))) ( -40.20) >DroPer_CAF1 18938 104 + 1 CCU----GCU-UGGGCU--------GCUGCCAACAUGGCAUUAAAGCCCAUUUAGCUGUCGUGUGGCAAUUAUUGAUCAACAAGGUGCCACAAUCGGCCAUGGUCGACGGA--GGCCCU ...----(((-((((((--------..((((.....))))....)))))......(((((.(((((((....(((.....)))..)))))))...(((....)))))))))--)))... ( -40.50) >consensus CC_____GCC_UGGGCUU______CGCUGCUAACAUGGCAUUAAAGCCCAUUUAGCUGUCGUGUGGCAAUUAUUGAUCAACAAGGUGCCACAAUCGGCCAUGGCCAA______GGCCCU ............................((((....(((......)))......((((...(((((((....(((.....)))..)))))))..))))..))))............... (-23.18 = -23.52 + 0.33)

| Location | 14,387,861 – 14,387,968 |
|---|---|
| Length | 107 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | reverse |
| Mean pairwise identity | 77.74 |
| Mean single sequence MFE | -38.88 |
| Consensus MFE | -26.29 |
| Energy contribution | -26.57 |
| Covariance contribution | 0.28 |
| Combinations/Pair | 1.08 |
| Mean z-score | -2.61 |
| Structure conservation index | 0.68 |
| SVM decision value | 1.91 |
| SVM RNA-class probability | 0.982241 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
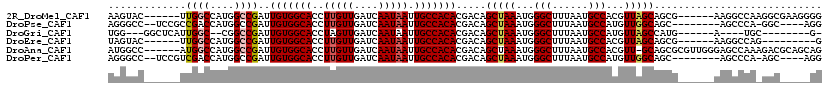
>2R_DroMel_CAF1 14387861 107 - 20766785 AAGUAC------UUGGCCAUGGCCGAUUGUGGCACCUUGUUGAUCAAUAAUUGCCACACGACAGCUAAAUGGGCUUUAAUGCCACGUUAGCAGCG------AAGGCCAAGGCGAAGGGG .....(------((.(((.(((((...(((((((..(((((....))))).)))))))((...(((((...(((......)))...)))))..))------..))))).))).)))... ( -45.60) >DroPse_CAF1 18927 104 - 1 AGGGCC--UCCGCCGACCAUGGCCGAUUGUGGCACCUUGUUGAUCAAUAAUUGCCACACGACAGCUAAAUGGGCUUUAAUGCCAUGUUGGCAGC--------AGCCCA-GGC----AGG .((((.--.(.((((((.(((((((..(((((((..(((((....))))).)))))))))..((((.....)))).....))))))))))).).--------.)))).-...----... ( -40.70) >DroGri_CAF1 28946 95 - 1 UGG---GGCUCAUUGGC--CGGCCGAUUGUGGCACCUAGUUGAUCAAUAAUUGCCACACGACAGCUAAAUGGGCUUUAAUGCCAUGUUAGCCAUG------A----UGC--------G- (((---(((((((((((--.(..((..(((((((....(((....)))...))))))))).).))).)))))))))))..((((((.....))))------.----.))--------.- ( -28.30) >DroEre_CAF1 21787 98 - 1 UAGUAC------UUGGCCAUGGCCGAUUGUGGCACCUUGUUGAUCAAUAAUUGCCACACGACAGCUAAAUGGGCUUUAAUGCCACGUUAGCAGCG------AAGGCCAG---------G .....(------((((((.(.((((..(((((((..(((((....))))).)))))))))...(((((...(((......)))...))))).)).------).))))))---------) ( -39.20) >DroAna_CAF1 24864 112 - 1 AUGGCC------AUGGCCAUGGCCGAUUGUGGCACCUUGUUGAUCAAUAAUUGCCACACGACAGCUAAAUGGGCUUUAAUGCCACGUU-GCAGCGCGUUGGGAGCCAAAGACGCAGCAG .(((((------((....)))))))..(((((((..(((((....))))).))))))).....(((.(((((((......))).))))-..)))(((((.(....)...)))))..... ( -41.50) >DroPer_CAF1 18938 104 - 1 AGGGCC--UCCGUCGACCAUGGCCGAUUGUGGCACCUUGUUGAUCAAUAAUUGCCACACGACAGCUAAAUGGGCUUUAAUGCCAUGUUGGCAGC--------AGCCCA-AGC----AGG .((((.--.(.((((((.(((((((..(((((((..(((((....))))).)))))))))..((((.....)))).....))))))))))).).--------.)))).-...----... ( -38.00) >consensus AGGGCC______UUGGCCAUGGCCGAUUGUGGCACCUUGUUGAUCAAUAAUUGCCACACGACAGCUAAAUGGGCUUUAAUGCCACGUUAGCAGCG______AAGCCCA_GGC_____GG .............((((....))))..(((((((..(((((....))))).))))))).....(((((...(((......)))...)))))............................ (-26.29 = -26.57 + 0.28)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:18:12 2006