
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 14,148,331 – 14,148,503 |
| Length | 172 |
| Max. P | 0.876357 |

| Location | 14,148,331 – 14,148,429 |
|---|---|
| Length | 98 |
| Sequences | 6 |
| Columns | 111 |
| Reading direction | forward |
| Mean pairwise identity | 81.72 |
| Mean single sequence MFE | -41.32 |
| Consensus MFE | -26.42 |
| Energy contribution | -26.32 |
| Covariance contribution | -0.11 |
| Combinations/Pair | 1.33 |
| Mean z-score | -2.51 |
| Structure conservation index | 0.64 |
| SVM decision value | 0.75 |
| SVM RNA-class probability | 0.840259 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 14148331 98 + 20766785 UAGCCACCGCC---------U---ACAGCGGUGUUAUUGCCAUACUUGGGUAGCGAUUGACCAGCUGACCGCUCAUAGCCACCGCGACUAUCU-UCUUUGGUGUCGCGGUG .(((((((((.---------.---...))))))..............((.((((.........)))).)))))......(((((((((.(((.-.....)))))))))))) ( -36.20) >DroVir_CAF1 912 96 + 1 UAGCCGG---C---------U---UGGGCCGAGUUAUUGCCGUAUUUGGGCAGCGCUAGUGCAGCUGACCGCUCAUAGCCACCGCGACUAUCUUUCUUUGGUGUCGCGGUG .....((---(---------(---((((((((((.........)))))((((((((....)).)))).))))))).))))((((((((.(((.......))))))))))). ( -40.90) >DroGri_CAF1 1106 95 + 1 UAGCCAG---C---------U---GGCGUCGAGUUAUUGCCAUAUUUGGCCAGCGCUUGGACCGCUGAGCGCUCAUAGCCACCGCGACUAUCU-UCUUUGGUGUCGCGGUG ..(((..---.---------.---))).....(((((.((((....)))).((((((..(....)..)))))).)))))(((((((((.(((.-.....)))))))))))) ( -42.70) >DroWil_CAF1 669 107 + 1 UAGCCGCCACCGCCAGCAGAU---UGGGCUGCAUUGUUGCCGUAUUUGGGCAGGGUUUGGGCAGCUGACCGCUCAUAGCCACCGCGACUAUCU-UCUUUGAUGUCGCGGUG ((((.(((...(((.((((..---....))))....(((((.......))))))))...))).))))...((.....))(((((((((.(((.-.....)))))))))))) ( -45.10) >DroMoj_CAF1 878 98 + 1 UAGCUGG---C---------UACCUGGGCCGAGUUAUUGCCGUAUUUGGGCAGCGCUUGUGCAGCUGACCGCUCAUAGCCACCGCGACUAUCU-UCUUUGGUGUCGCGGUG .....((---(---------((..((((((((((.........)))))((((((((....)).)))).))))))))))))((((((((.(((.-.....))))))))))). ( -41.50) >DroAna_CAF1 632 98 + 1 UAGCCACCGCC---------G---GCAGCCGAGUUAUUGCCGUACUUGGGCAGCGAUUGACCAGCCGAUCGCUCAUAGCCACCGCGACUAUCU-UCUUUGGUGUCGCGGUG ..(((.....(---------(---((((........))))))......)))((((((((......))))))))......(((((((((.(((.-.....)))))))))))) ( -41.50) >consensus UAGCCAC___C_________U___UGGGCCGAGUUAUUGCCGUAUUUGGGCAGCGCUUGAGCAGCUGACCGCUCAUAGCCACCGCGACUAUCU_UCUUUGGUGUCGCGGUG .........................(((((((((.........)))))((((((.........)))).)))))).....(((((((((.(((.......)))))))))))) (-26.42 = -26.32 + -0.11)


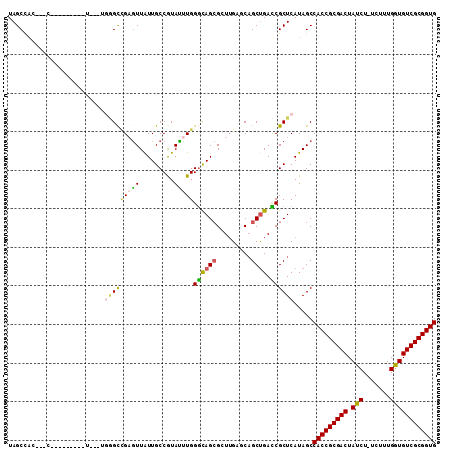
| Location | 14,148,331 – 14,148,429 |
|---|---|
| Length | 98 |
| Sequences | 6 |
| Columns | 111 |
| Reading direction | reverse |
| Mean pairwise identity | 81.72 |
| Mean single sequence MFE | -39.24 |
| Consensus MFE | -25.00 |
| Energy contribution | -25.47 |
| Covariance contribution | 0.47 |
| Combinations/Pair | 1.17 |
| Mean z-score | -2.74 |
| Structure conservation index | 0.64 |
| SVM decision value | 0.89 |
| SVM RNA-class probability | 0.876357 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 14148331 98 - 20766785 CACCGCGACACCAAAGA-AGAUAGUCGCGGUGGCUAUGAGCGGUCAGCUGGUCAAUCGCUACCCAAGUAUGGCAAUAACACCGCUGU---A---------GGCGGUGGCUA (((((((((........-.....)))))))))((((((...((..(((.........)))..))...)))))).....((((((...---.---------.)))))).... ( -39.42) >DroVir_CAF1 912 96 - 1 CACCGCGACACCAAAGAAAGAUAGUCGCGGUGGCUAUGAGCGGUCAGCUGCACUAGCGCUGCCCAAAUACGGCAAUAACUCGGCCCA---A---------G---CCGGCUA (((((((((..............)))))))))((((.(.((((....)))).)))))((((((.......))))......((((...---.---------)---))))).. ( -37.84) >DroGri_CAF1 1106 95 - 1 CACCGCGACACCAAAGA-AGAUAGUCGCGGUGGCUAUGAGCGCUCAGCGGUCCAAGCGCUGGCCAAAUAUGGCAAUAACUCGACGCC---A---------G---CUGGCUA (((((((((........-.....)))))))))((.....))((.((((((.(((.....))))).....((((...........)))---)---------)---))))).. ( -36.42) >DroWil_CAF1 669 107 - 1 CACCGCGACAUCAAAGA-AGAUAGUCGCGGUGGCUAUGAGCGGUCAGCUGCCCAAACCCUGCCCAAAUACGGCAACAAUGCAGCCCA---AUCUGCUGGCGGUGGCGGCUA ((((((((((((.....-.))).)))))))))......(((.((((.(((((.......((((.......)))).....((((....---..)))).))))))))).))). ( -46.10) >DroMoj_CAF1 878 98 - 1 CACCGCGACACCAAAGA-AGAUAGUCGCGGUGGCUAUGAGCGGUCAGCUGCACAAGCGCUGCCCAAAUACGGCAAUAACUCGGCCCAGGUA---------G---CCAGCUA .((((((((........-.....))))))))((((((..(.((((.(((.....)))((((........))))........)))))..)))---------)---))..... ( -37.12) >DroAna_CAF1 632 98 - 1 CACCGCGACACCAAAGA-AGAUAGUCGCGGUGGCUAUGAGCGAUCGGCUGGUCAAUCGCUGCCCAAGUACGGCAAUAACUCGGCUGC---C---------GGCGGUGGCUA (((((((((........-.....)))))))))......(((.((((.((((.((..((.((((.......))))......))..)))---)---------)))))).))). ( -38.52) >consensus CACCGCGACACCAAAGA_AGAUAGUCGCGGUGGCUAUGAGCGGUCAGCUGCACAAGCGCUGCCCAAAUACGGCAAUAACUCGGCCCA___A_________G___CCGGCUA (((((((((..............)))))))))......(((((.((((.((....)))))).))......(((.........)))......................))). (-25.00 = -25.47 + 0.47)


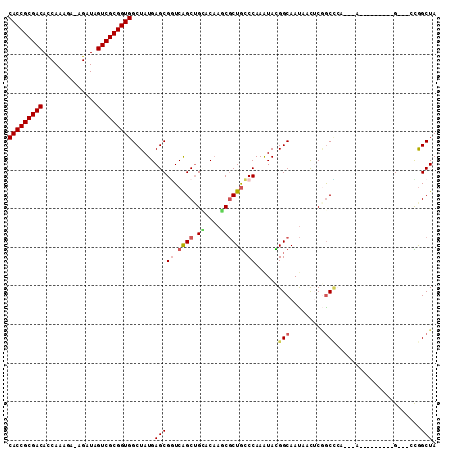
| Location | 14,148,350 – 14,148,466 |
|---|---|
| Length | 116 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 87.70 |
| Mean single sequence MFE | -47.24 |
| Consensus MFE | -35.93 |
| Energy contribution | -35.79 |
| Covariance contribution | -0.14 |
| Combinations/Pair | 1.17 |
| Mean z-score | -3.22 |
| Structure conservation index | 0.76 |
| SVM decision value | 0.62 |
| SVM RNA-class probability | 0.801818 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 14148350 116 - 20766785 AGGUGGUAC---UCAUCAACAACGGGAUUUUCGGCCUGGCCACCGCGACACCAAAGA-AGAUAGUCGCGGUGGCUAUGAGCGGUCAGCUGGUCAAUCGCUACCCAAGUAUGGCAAUAACA .((((((.(---((.........)))......(((((((((((((((((........-.....)))))))))))))..(((.....)))))))....))))))................. ( -42.02) >DroVir_CAF1 928 117 - 1 CGGCGGCAA---UCAUCAACAACGGGAUUUUCGGCAUGGCCACCGCGACACCAAAGAAAGAUAGUCGCGGUGGCUAUGAGCGGUCAGCUGCACUAGCGCUGCCCAAAUACGGCAAUAACU .((((((..---......................(((((((((((((((..............))))))))))))))).((((....))))......))))))................. ( -46.84) >DroGri_CAF1 1122 119 - 1 CGGCGGCAGCCACCAGCAACAACGGGAUUUUCGGCAUGGCCACCGCGACACCAAAGA-AGAUAGUCGCGGUGGCUAUGAGCGCUCAGCGGUCCAAGCGCUGGCCAAAUAUGGCAAUAACU .(((....)))..((((.......(((((.....(((((((((((((((........-.....))))))))))))))).((.....)))))))....))))((((....))))....... ( -50.92) >DroWil_CAF1 697 119 - 1 AGGUGGAGGCCAGCAUCAACAACGGGAUUUUCGGCCUGGCCACCGCGACAUCAAAGA-AGAUAGUCGCGGUGGCUAUGAGCGGUCAGCUGCCCAAACCCUGCCCAAAUACGGCAACAAUG .((..(.((((.((.(((....((((........))))((((((((((((((.....-.))).)))))))))))..)))))))))..)..)).......((((.......))))...... ( -49.40) >DroMoj_CAF1 897 116 - 1 CGGCGGUAA---UCAUCAACAACGGGAUUUUCGGCAUGGCCACCGCGACACCAAAGA-AGAUAGUCGCGGUGGCUAUGAGCGGUCAGCUGCACAAGCGCUGCCCAAAUACGGCAAUAACU ..((.(((.---......................(((((((((((((((........-.....)))))))))))))))...((.((((.((....)))))).))...))).))....... ( -45.82) >DroAna_CAF1 651 116 - 1 AGGCAGUGC---UCAUCAACAACGGGAUUUUCGGCCUGGCCACCGCGACACCAAAGA-AGAUAGUCGCGGUGGCUAUGAGCGAUCGGCUGGUCAAUCGCUGCCCAAGUACGGCAAUAACU .((((((((---((((......((((........))))(((((((((((........-.....))))))))))).))))))((((....))))....))))))................. ( -48.42) >consensus AGGCGGUAA___UCAUCAACAACGGGAUUUUCGGCAUGGCCACCGCGACACCAAAGA_AGAUAGUCGCGGUGGCUAUGAGCGGUCAGCUGCACAAGCGCUGCCCAAAUACGGCAAUAACU .((((((.......(((........)))...((((.(((((((((((((..............)))))))))))))(((....))))))).......))))))................. (-35.93 = -35.79 + -0.14)

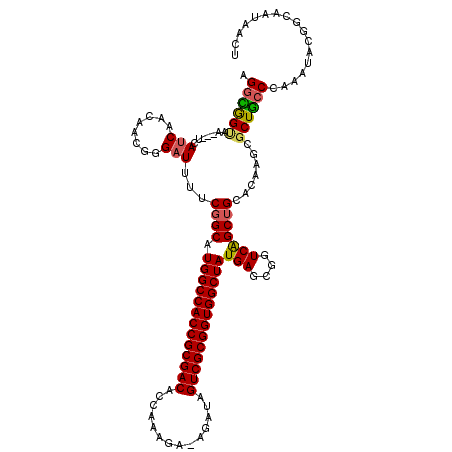
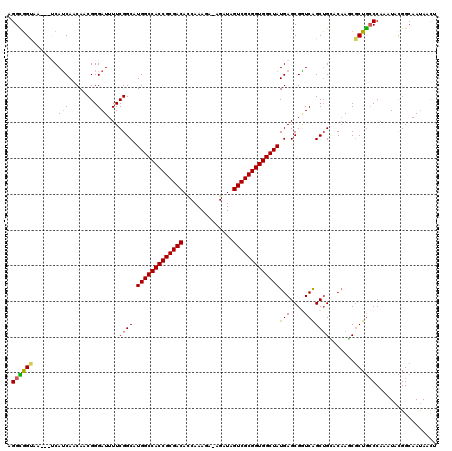
| Location | 14,148,390 – 14,148,503 |
|---|---|
| Length | 113 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 88.81 |
| Mean single sequence MFE | -47.94 |
| Consensus MFE | -35.96 |
| Energy contribution | -35.37 |
| Covariance contribution | -0.58 |
| Combinations/Pair | 1.15 |
| Mean z-score | -2.82 |
| Structure conservation index | 0.75 |
| SVM decision value | -0.01 |
| SVM RNA-class probability | 0.530692 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 14148390 113 - 20766785 GGAACCGGGGCAGCAGCCAGAGU---AGCUAUCGCUCGGGAGGUGGUAC---UCAUCAACAACGGGAUUUUCGGCCUGGCCACCGCGACACCAAAGA-AGAUAGUCGCGGUGGCUAUGAG ((..(((((((....))).((((---(.(((((........))))))))---))................))))))(((((((((((((........-.....))))))))))))).... ( -45.12) >DroVir_CAF1 968 114 - 1 GCAAUCGAGGCAGCAGCCAGAGC---AGCUAUCGCUCGGGCGGCGGCAA---UCAUCAACAACGGGAUUUUCGGCAUGGCCACCGCGACACCAAAGAAAGAUAGUCGCGGUGGCUAUGAG ....(((((((.((.(((.((((---.......)))).))).)).))((---((.((......)))))))))))(((((((((((((((..............))))))))))))))).. ( -50.14) >DroGri_CAF1 1162 116 - 1 GCAAUCGCGGCAACAGCCAGAGC---AGCUAUCGCUCCGGCGGCGGCAGCCACCAGCAACAACGGGAUUUUCGGCAUGGCCACCGCGACACCAAAGA-AGAUAGUCGCGGUGGCUAUGAG ..((((.((......(((.((((---.......)))).)))(((....)))...........)).)))).....(((((((((((((((........-.....))))))))))))))).. ( -52.02) >DroWil_CAF1 737 116 - 1 GAAAUCGGGGCAACAGCCAAAGC---AGCUAUCGCUCUGGAGGUGGAGGCCAGCAUCAACAACGGGAUUUUCGGCCUGGCCACCGCGACAUCAAAGA-AGAUAGUCGCGGUGGCUAUGAG ....(((((((...(((......---.)))...)))))))......(((((((.(((........))).)).)))))(((((((((((((((.....-.))).))))))))))))..... ( -47.20) >DroMoj_CAF1 937 116 - 1 GUAACCGAGGCAGCAGCCAAAGCAGCAGCUAUCGCUCCGGCGGCGGUAA---UCAUCAACAACGGGAUUUUCGGCAUGGCCACCGCGACACCAAAGA-AGAUAGUCGCGGUGGCUAUGAG ....(((((((.((.((....)).)).))(((((((.....))))))).---.................)))))(((((((((((((((........-.....))))))))))))))).. ( -46.52) >DroAna_CAF1 691 113 - 1 GUAACCGGGGCAGCAGCCAGAGC---AGUUAUCGCUCCGGAGGCAGUGC---UCAUCAACAACGGGAUUUUCGGCCUGGCCACCGCGACACCAAAGA-AGAUAGUCGCGGUGGCUAUGAG ((..((((((((((.((....))---.)))...)))))))..))....(---((((......((((........))))(((((((((((........-.....))))))))))).))))) ( -46.62) >consensus GCAACCGGGGCAGCAGCCAGAGC___AGCUAUCGCUCCGGAGGCGGUAA___UCAUCAACAACGGGAUUUUCGGCAUGGCCACCGCGACACCAAAGA_AGAUAGUCGCGGUGGCUAUGAG ....(((.(((....)))..(((....))).(((((.....)))))................)))...........(((((((((((((..............))))))))))))).... (-35.96 = -35.37 + -0.58)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:16:38 2006