
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 14,074,727 – 14,074,841 |
| Length | 114 |
| Max. P | 0.607548 |

| Location | 14,074,727 – 14,074,841 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 80.56 |
| Mean single sequence MFE | -57.97 |
| Consensus MFE | -32.93 |
| Energy contribution | -33.41 |
| Covariance contribution | 0.48 |
| Combinations/Pair | 1.24 |
| Mean z-score | -1.93 |
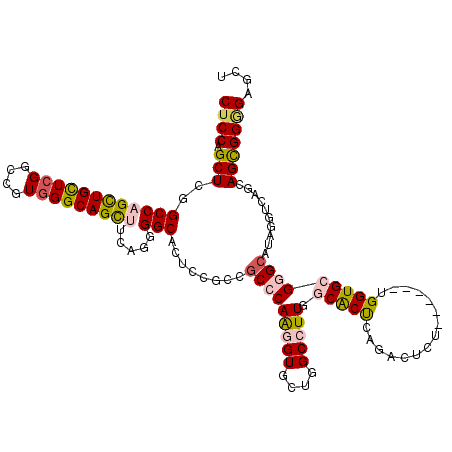
| Structure conservation index | 0.57 |
| SVM decision value | 0.15 |
| SVM RNA-class probability | 0.607548 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
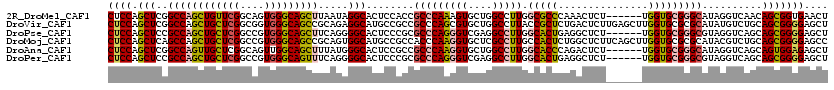
>2R_DroMel_CAF1 14074727 114 + 20766785 CUCCAGCUCGGCCAGCUGUUCGGCAGUGGGCAGCUUAAUAGGCACUCCACCGCCCAAAGUGCUGGCCUUGGCGCCCAAACUCU------UGGUGCGGGCAUAGGUCAACAGCGGUGAACU (.((.(((.(((((((..((.(((.(((((..(((.....)))..))))).))).))...)))))))(((((((((..((...------..))..))))....))))).))))).).... ( -46.00) >DroVir_CAF1 1867 120 + 1 CUCCAGCUCGGCCAGCUGCUCGGCGGUGGGCAGCCGCAGAGGCAUGCCGCCGCCCAGCGUGCUGGCCUUACCGCUCUGACUCUUGAGCUUGGUGCGCGCAUAUGUCUGCAGCGGGGAGCU ....((((((((((((((((.((((((((.(((((.....))).)))))))))).)))).)))))))...((((((.(((...((.((.....))...))...))).).))))).))))) ( -68.90) >DroPse_CAF1 1593 114 + 1 CUCCAGCUCCGCCAGCUGCUCGGCCGUGGGCAGCUUCAGGGGCACUCCCGCGCCCAGGGUCGAGGCCUUGGCACUGAGGCUCU------UGGUGCGGGCGUAGGUCAGCAGCGGGGAGCU ....((((((.((.((((((.(((((((((..(((.....)))...)))))(((((((((....))))).((((..((...))------..))))))))...)))))))))))))))))) ( -65.00) >DroMoj_CAF1 2094 120 + 1 CUCCAGCUCAGCCAGCUGCUCGGCCGUGGGCAGCCGCAGUGGCAUGCCGCCACCCAAGGUGCUCGCCUUGCCACUCUGGCUCUUCAGCUUGGUGCGCGCAUACGUCUGCAGCGGGGAGCC .....((((..((.(((((..(((.(((.(((((((.(((((((.((((((......))))...))..))))))).))))).....((.....)))).)))..))).))))))).)))). ( -53.00) >DroAna_CAF1 1475 114 + 1 CUCCAGCUCGGCCAGUUGCUCGGCAGUUGGCAGCUUUAUGGGCACUCCGCCGCCCAAGGUGCUGGCCUUGGCACCCAGACUCU------UGGUGCGGGCAUAGGUCAGCAGUGGAGAGCU ((((((((.((((...((((((((....((((.((...(((((........))))))).)))).)))...(((((.((...))------.))))))))))..)))))))..))))).... ( -50.80) >DroPer_CAF1 1491 114 + 1 CUCCAGCUCCGCCAGCUGCUCGGCCGUGGGCAGUUUCAGGGGCACUCCCGCGCCCAGGGUCGAGGCCUUGGCACUGAGGCUCU------UGGUGCGGGCGUAGGUCAGCAGCGGGGAGCU ....((((((.((.((((((.(((((((((.(((.(.....).))))))))(((((((((....))))).((((..((...))------..))))))))...)))))))))))))))))) ( -64.10) >consensus CUCCAGCUCGGCCAGCUGCUCGGCCGUGGGCAGCUUCAGGGGCACUCCGCCGCCCAAGGUGCUGGCCUUGGCACUCAGACUCU______UGGUGCGGGCAUAGGUCAGCAGCGGGGAGCU ((((.(((..((((((((((((....))))))))).....)))........(((((((((....))))).(((((...............)))))))))..........))))))).... (-32.93 = -33.41 + 0.48)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:15:35 2006