
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 14,073,438 – 14,073,548 |
| Length | 110 |
| Max. P | 0.500000 |

| Location | 14,073,438 – 14,073,548 |
|---|---|
| Length | 110 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 78.73 |
| Mean single sequence MFE | -42.05 |
| Consensus MFE | -25.08 |
| Energy contribution | -25.92 |
| Covariance contribution | 0.84 |
| Combinations/Pair | 1.17 |
| Mean z-score | -1.46 |
| Structure conservation index | 0.60 |
| SVM decision value | -0.07 |
| SVM RNA-class probability | 0.500000 |
| Prediction | OTHER |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 14073438 110 + 20766785 GAUGUUGGAGUAGCUUCUGCGUGGAGGCAAGCAGAGAGUACGUGUGCGUGGCCUGCAGCAACUUGGUCAGCAACGAUAUGGCUUCCGGGUAGCUUGUGCUGU------GGGGAAUC---- .((((....(((..((((((.((....)).))))))..)))....))))..((..(((((((((((..(((.........))).))))))......))))).------.)).....---- ( -34.70) >DroVir_CAF1 75 114 + 1 GAUGUUGCAGCAGCUUCUGCGUGGAGGCCAGCAGCGAGUUCGUGUGUUUCGCCUGCAGCAGCUUGGUCAGCAGCGAGAUGGCUUCCGGGUAGCUUGUGCUU------AAAAAAAGUGACA ..(((..(((((....((((.(((((((((((((((((.........)))).)))).((.(((.....))).))....))))))))).))))....)))).------.......)..))) ( -42.00) >DroGri_CAF1 75 114 + 1 GAUGCUGCAGCAGCUUCUGCGUGGAGGCAAGCAGCGAGUUCGUGUGCUUCGCCUGCAGCAGUUUGGUCAGCAGCGAGAUGGCUUCCGGGUAGCUUGUGCUG------CACAAAAACAAAA ..(((((((((.((((((....))))))((((((((....))).))))).).))))))))((((.(((((((.((((...(((....)))..)))))))))------.))..)))).... ( -45.00) >DroWil_CAF1 75 114 + 1 GAUGUUGCAACAGCUUCUGCGUGGAGGCCAAUAACGCAUUCGUGUGCGUUGCCUGCAGCAGUUUGGUCAACAGUGAAAUGGCUUCCGGGUAGCUUGUACUGC------AAUAAGCCCAAA ..(((((((((((...((((.(((((((((...((((((....))))((((((.((....))..)).)))).))....))))))))).)))).))))..)))------))))........ ( -41.50) >DroMoj_CAF1 75 120 + 1 GAUGCUGCAGCAGCGUCUGCGUGGAGGCCAGCAACGUAUUCGUAUGCUUCGCCUGCAGCAGUUUUGUCAACAGCGAGAUGGCUUCCGGGUAGCUUGUGCUGUAAGACAAAAGAAGAAAUA (((((((...)))))))(((.((.((((.(((((((....))).))))..)))).))))).(((((((.((((((....((((.......))))..))))))..)))))))......... ( -44.70) >DroAna_CAF1 75 113 + 1 GGUGCUGGAGGUGCUUCUGGGUGGAGGCCAGCAGGGAGUAGGUGUGCGUGGCCUGAAGCAGCUUGGUCAGCAGCGAGAUGGCUUCCGGGUAGCUUGUGCUGCAAU---GCCAAAAC---- (.((((((((.((((((.......(((((((((.(.......).))).)))))))))))).)))...))))).)....((((......(((((....)))))...---))))....---- ( -44.41) >consensus GAUGCUGCAGCAGCUUCUGCGUGGAGGCCAGCAGCGAGUUCGUGUGCGUCGCCUGCAGCAGCUUGGUCAGCAGCGAGAUGGCUUCCGGGUAGCUUGUGCUG_______AAAAAAAC_A_A ..((((((((..((((((....))))))..((((((....))).))).....))))))))..........(((((....((((.......))))..)))))................... (-25.08 = -25.92 + 0.84)

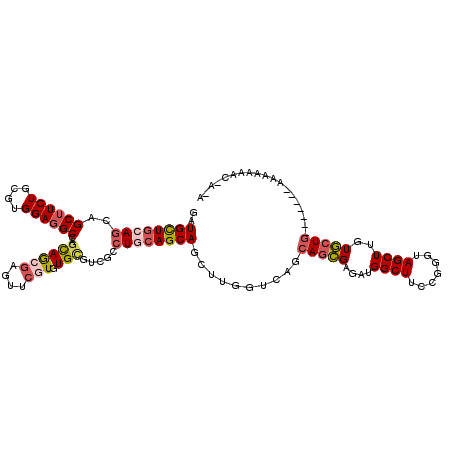
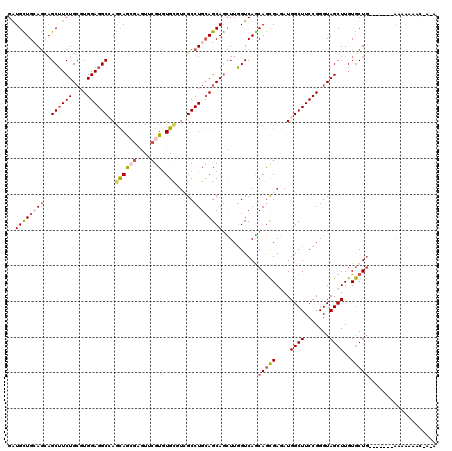
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:15:33 2006