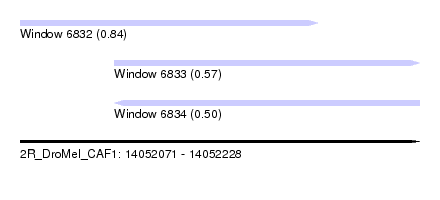
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 14,052,071 – 14,052,228 |
| Length | 157 |
| Max. P | 0.842561 |

| Location | 14,052,071 – 14,052,188 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 79.10 |
| Mean single sequence MFE | -31.98 |
| Consensus MFE | -20.60 |
| Energy contribution | -21.27 |
| Covariance contribution | 0.67 |
| Combinations/Pair | 1.16 |
| Mean z-score | -1.82 |
| Structure conservation index | 0.64 |
| SVM decision value | 0.76 |
| SVM RNA-class probability | 0.842561 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
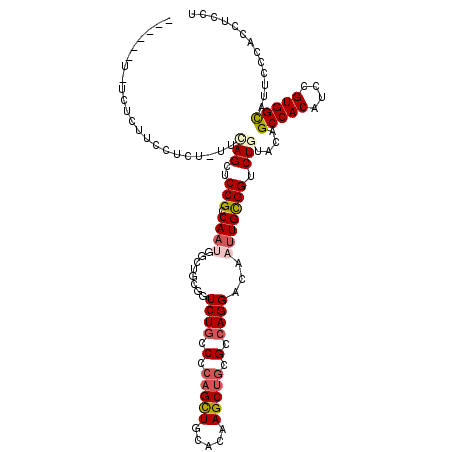
>2R_DroMel_CAF1 14052071 117 + 20766785 CUUUUAUCUCUCUCUUUCUCUUUAGCUCCGGCAAUGUCUGCGGUCUGCCCCAGUUGCACAAGCUGCGCCAGGACAAUUGCGGUCUGUACAGCCACAUCCGUGGCAUUCCCAUCUCCU ......................(((..(((.((((((((((((....)).(((((.....))))).))..)))).))))))).)))....(((((....)))))............. ( -27.60) >DroVir_CAF1 33732 109 + 1 ------U-UAUUUUCCACU-UACAGUUCCGCCAAUGGCUGCGGUCUGCCCCAGCUGCACAAGCUGCGCCAGGAUAAUUGUGGCCUGUACAGCCACAUCCGUGGCAUUCCCACCUCGU ------.-...........-..(((..((((........)))).)))...(((((.....))))).(((((((....((((((.......))))))))).))))............. ( -32.80) >DroGri_CAF1 25704 110 + 1 ------UCUCUCUUCCACU-UACAGCUCCGCCAAUGGUUGCGGUCUUCCCCAGCUGCACAAGCAGCGCCAGGACAAUUGUGGACUCUAUAGCCACAUUCGUGGUAUUCCCACCUCUU ------.......(((((.-....((.(((....)))..)).(((((.....(((((....)))))...)))))....))))).......(((((....)))))............. ( -29.60) >DroEre_CAF1 19593 103 + 1 ------U--------CUCCCCUCAGCUCCGGCAACGUGUGCGGUCUGCCCCAGUUGCACAAGCUGCGCCAGGACAAUUGCGGUCUGUACAGCCACAUCCGUGGCAUUCCCAUCUCCU ------.--------.......(((((.((....))(((((((.(((...))))))))))))))).(((((((....((.((.((....)))).))))).))))............. ( -30.80) >DroMoj_CAF1 26774 110 + 1 ------C-UUCUCUCCUCUUUUCAGUGCCGCCAACGGCUGUGCUCUGCCUCAGCUGAACAAGCUCCGCCAGGACAACUGCGGCCUGUACAGCCACAUUCGUGGCAUUCCCACCUCGU ------.-...............((((((((....((((((((...(((.(((((.....)))(((....)))....)).)))..))))))))......)))))))).......... ( -35.80) >DroAna_CAF1 31499 108 + 1 ------CCUCUCUCUGU---UGUAGCUCCGCCAAUGCCUGCGGUCUGCCCCAGCUGCACAAGCUGCGCCAGGACAAUUGCGGUCUCUACAGCCACAUCCGUGGCAUCCCCAUCUCCU ------...........---(((((..((((.(((.(((((((....)).(((((.....))))).).))))...)))))))...)))))(((((....)))))............. ( -35.30) >consensus ______U_UCUCUUCCUCU_UUCAGCUCCGCCAAUGGCUGCGGUCUGCCCCAGCUGCACAAGCUGCGCCAGGACAAUUGCGGUCUGUACAGCCACAUCCGUGGCAUUCCCACCUCCU ......................(((..(((.((((........((((.(.(((((.....))))).).))))...))))))).)))....(((((....)))))............. (-20.60 = -21.27 + 0.67)

| Location | 14,052,108 – 14,052,228 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 81.22 |
| Mean single sequence MFE | -43.30 |
| Consensus MFE | -25.63 |
| Energy contribution | -25.25 |
| Covariance contribution | -0.38 |
| Combinations/Pair | 1.52 |
| Mean z-score | -1.84 |
| Structure conservation index | 0.59 |
| SVM decision value | 0.07 |
| SVM RNA-class probability | 0.568899 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
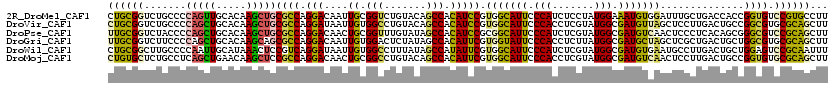
>2R_DroMel_CAF1 14052108 120 + 20766785 CUGCGGUCUGCCCCAGUUGCACAAGCUGCGCCAGGACAAUUGCGGUCUGUACAGCCACAUCCGUGGCAUUCCCAUCUCCUAUGGAAAUGUGGAUUUGCUGACCACCGGUGUCCGUGCCUU (((.((....)).)))..((((..((...))..(((((...(.((((.(((..(((((....)))))..((((((.(((...))).))).)))..))).)))).)...)))))))))... ( -41.90) >DroVir_CAF1 33761 120 + 1 CUGCGGUCUGCCCCAGCUGCACAAGCUGCGCCAGGAUAAUUGUGGCCUGUACAGCCACAUCCGUGGCAUUCCCACCUCGUAUGGCGAUGUUAGCUCCUUGACUGCCGGCGUGCGCAGCUU .(((((.(((...)))))))).((((((((((((((....((((((.......))))))))).))))...........(((((.((..(((((....)))))...)).)))))))))))) ( -47.70) >DroPse_CAF1 25865 120 + 1 UUGCGGUCUACCCCAGCUGCACAAGCUGCGCCAGGACAACUGCGGUUUGUAUAGCCACAUCCGCGGCAUUCCCAUCUCGUAUGGCGAUGUCAACUCCCUCACAGCGGGCGUCCGCAGCUU .((((((........)))))).((((((((((((((...((((((..(((......))).))))))...))).........))))((((((..((.......))..)))))).))))))) ( -43.10) >DroGri_CAF1 25734 120 + 1 UUGCGGUCUUCCCCAGCUGCACAAGCAGCGCCAGGACAAUUGUGGACUCUAUAGCCACAUUCGUGGUAUUCCCACCUCUUAUGGCGAUGCUAGCUCGCUGACUGCUGGCGUGCGCAGCUU ....((......))((((((...((((.((((((((....(((((.((....)))))))...((((.....)))).)))..))))).)))).((.((((.......)))).)))))))). ( -44.20) >DroWil_CAF1 31372 120 + 1 CUGCGGCUUGCCCCAAUUGCAUAAACUCCGUCAGGAUAAUUGUGGCCUUUAUAGCCAUAUUCGUGGCAUUCCCAUCUCGUAUGGCGAUGUGAAUGCCUUGACUGCUGGAGUCCGCAAUUU .(((((..(((.......)))...((((((.(((......((((((.......)))))).(((.(((((((.((((.(.....).)))).))))))).)))))).))))))))))).... ( -43.60) >DroMoj_CAF1 26804 120 + 1 CUGUGCUCUGCCUCAGCUGAACAAGCUCCGCCAGGACAACUGCGGCCUGUACAGCCACAUUCGUGGCAUUCCCACCUCGUAUGGCGAUGUCAACUCCUUGACUGCCGGUGUGCGCAGCUU ((((((.(.(((..((((.....))))((((.((.....))))))........(((((....)))))...............((((..(((((....))))))))))))).))))))... ( -39.30) >consensus CUGCGGUCUGCCCCAGCUGCACAAGCUGCGCCAGGACAAUUGCGGCCUGUACAGCCACAUCCGUGGCAUUCCCACCUCGUAUGGCGAUGUCAACUCCCUGACUGCCGGCGUCCGCAGCUU ((((((.......(((((.....)))))((((.(((....((.(((.......))).))))).(((((((.(((.......))).)))))))..............)))).))))))... (-25.63 = -25.25 + -0.38)
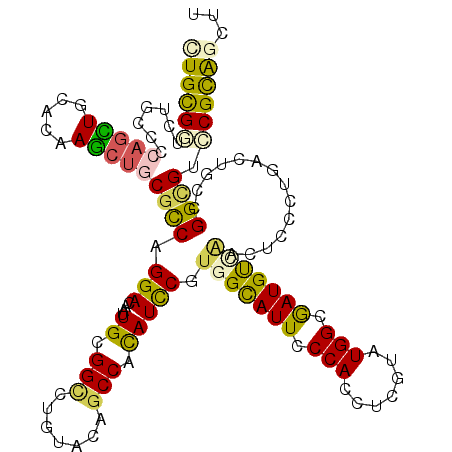
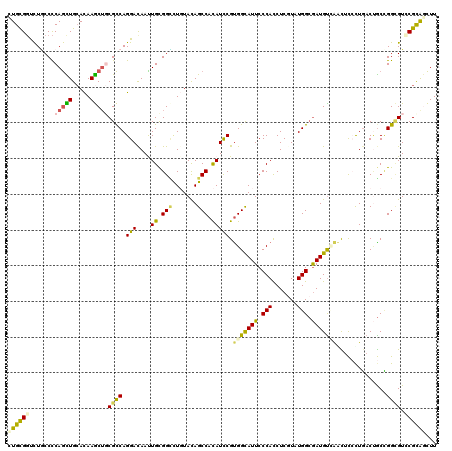
| Location | 14,052,108 – 14,052,228 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 81.22 |
| Mean single sequence MFE | -47.20 |
| Consensus MFE | -25.32 |
| Energy contribution | -25.60 |
| Covariance contribution | 0.28 |
| Combinations/Pair | 1.33 |
| Mean z-score | -2.07 |
| Structure conservation index | 0.54 |
| SVM decision value | -0.06 |
| SVM RNA-class probability | 0.500000 |
| Prediction | OTHER |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 14052108 120 - 20766785 AAGGCACGGACACCGGUGGUCAGCAAAUCCACAUUUCCAUAGGAGAUGGGAAUGCCACGGAUGUGGCUGUACAGACCGCAAUUGUCCUGGCGCAGCUUGUGCAACUGGGGCAGACCGCAG ...(((((((((...((((((......(((.(((((((...))))))))))..(((((....)))))......))))))...))))).(((...))).))))..(((.((....)).))) ( -48.90) >DroVir_CAF1 33761 120 - 1 AAGCUGCGCACGCCGGCAGUCAAGGAGCUAACAUCGCCAUACGAGGUGGGAAUGCCACGGAUGUGGCUGUACAGGCCACAAUUAUCCUGGCGCAGCUUGUGCAGCUGGGGCAGACCGCAG .(((((((((.((..((.((((.(((((.....(((((......)))))....))......(((((((.....)))))))....))))))))).)).)))))))))(.((....)).).. ( -49.00) >DroPse_CAF1 25865 120 - 1 AAGCUGCGGACGCCCGCUGUGAGGGAGUUGACAUCGCCAUACGAGAUGGGAAUGCCGCGGAUGUGGCUAUACAAACCGCAGUUGUCCUGGCGCAGCUUGUGCAGCUGGGGUAGACCGCAA ...((((((...(((((((((.((((.......)).)).))).)).))))....)))))).(((((((......(((.((((((..(.(((...))).)..)))))).))))).))))). ( -46.40) >DroGri_CAF1 25734 120 - 1 AAGCUGCGCACGCCAGCAGUCAGCGAGCUAGCAUCGCCAUAAGAGGUGGGAAUACCACGAAUGUGGCUAUAGAGUCCACAAUUGUCCUGGCGCUGCUUGUGCAGCUGGGGAAGACCGCAA .(((((((((.((..((.....))..)).((((.(((((.....((((....))))((((.(((((((....)).))))).))))..))))).)))))))))))))(.((....)).).. ( -47.70) >DroWil_CAF1 31372 120 - 1 AAAUUGCGGACUCCAGCAGUCAAGGCAUUCACAUCGCCAUACGAGAUGGGAAUGCCACGAAUAUGGCUAUAAAGGCCACAAUUAUCCUGACGGAGUUUAUGCAAUUGGGGCAAGCCGCAG .(((((((((((((..(((....(((((((.((((.........)))).))))))).......(((((.....)))))........)))..))))))..)))))))(.((....)).).. ( -43.50) >DroMoj_CAF1 26804 120 - 1 AAGCUGCGCACACCGGCAGUCAAGGAGUUGACAUCGCCAUACGAGGUGGGAAUGCCACGAAUGUGGCUGUACAGGCCGCAGUUGUCCUGGCGGAGCUUGUUCAGCUGAGGCAGAGCACAG (((((.(((.((..(((((((((....))))).(((((......)))))...))))((((.(((((((.....))))))).))))..))))).)))))((((.((....)).)))).... ( -47.70) >consensus AAGCUGCGCACGCCGGCAGUCAAGGAGUUAACAUCGCCAUACGAGAUGGGAAUGCCACGAAUGUGGCUAUACAGGCCACAAUUGUCCUGGCGCAGCUUGUGCAGCUGGGGCAGACCGCAG ....(((((.(.(((((.((..(.(((((....((.((((.....)))))).(((((.((.(((((((....)).)))))....)).))))).))))).))).))))).)....))))). (-25.32 = -25.60 + 0.28)

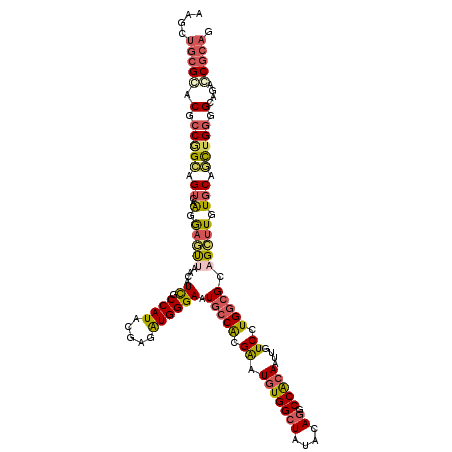
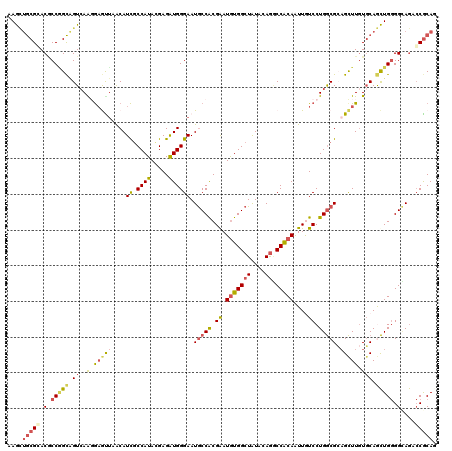
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:15:28 2006