
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 13,799,019 – 13,799,216 |
| Length | 197 |
| Max. P | 0.982405 |

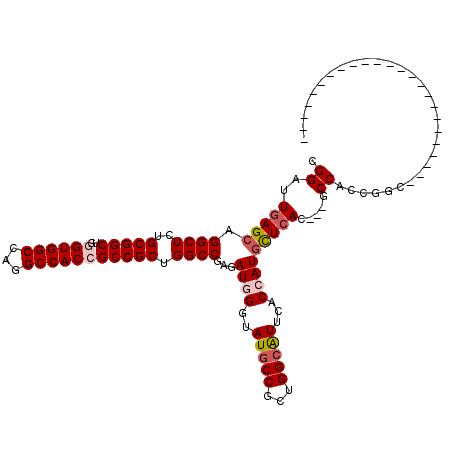
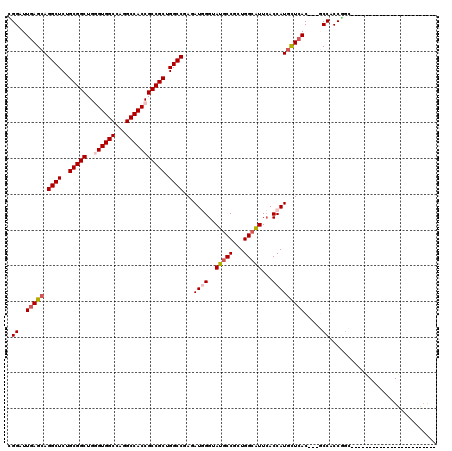
| Location | 13,799,019 – 13,799,136 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 76.09 |
| Mean single sequence MFE | -51.12 |
| Consensus MFE | -37.09 |
| Energy contribution | -37.78 |
| Covariance contribution | 0.70 |
| Combinations/Pair | 1.06 |
| Mean z-score | -2.17 |
| Structure conservation index | 0.73 |
| SVM decision value | 1.91 |
| SVM RNA-class probability | 0.982405 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 13799019 117 + 20766785 CGGAUUGAGCAGGCUCUGCGGCUGCGUGGCCAGGCCACCGCCGCUGGCCGAUAUGGGGAUGCCACUGGCGUUCACCAUGUUCAC---UCCACCGGCUCCACCAAUACCGCCAACAGACGC .(((.(((((.((((..(((((.(.(((((...))))))))))).))))...(((((((((((...))))))).))))))))).---)))...(((............)))......... ( -51.60) >DroVir_CAF1 122146 96 + 1 UGGGUUGAGCAGGCUCUGCGGCUGGGUGGCCAGGCCACCGCCGCUGGCCGAGAUGGGUAUGCCGCUGGCAUUCACCAUGCUCACCAUGCCCCCAGC------------------------ .(((((((((.((((..(((((..((((((...))))))))))).))))...((((..(((((...)))))...)))))))))....)))).....------------------------ ( -49.40) >DroGri_CAF1 118362 96 + 1 CGGAUUGAGCAGGCUCUGCGGCUGGGUGGCCAGGCCACCGCCGCUGGCCGAAAUUGGUAUGCCGCUGGCAUUGACCAUGCUCACAAUUCCACCGGC------------------------ .(((.((((((((((..(((((..((((((...))))))))))).)))).....(((((((((...)))))..))))))))))....)))......------------------------ ( -47.00) >DroWil_CAF1 123156 117 + 1 CGGAUUGAGCAGGCUCUGCGGCUGUGUGGCCAGGCCACCGCCGCUGGCCGAUAUGGGAAUUCCACUGGCAUUCACCAUAUUGAC---GCCCCCCACUCCGCCGGCACCGGCUCCGCCCGC ((((..(((..((....(((((.(.(((((...))))))))))).(((((((((((((((.((...)).)))).))))))))..---))).))..))).((((....))))))))..... ( -49.50) >DroMoj_CAF1 136065 96 + 1 UGGAUUGAGCAGGCUCUGCGGCUGGGUGGCCAGGCCACCGCCGCUGGCCGAGAUGGGAAUGCCGCUGGCAUUCACCAUGCUCACCAUGCCUCCGGC------------------------ (((..(((((.((((..(((((..((((((...))))))))))).))))...(((((((((((...))))))).)))))))))))).(((...)))------------------------ ( -51.30) >DroAna_CAF1 108612 117 + 1 CGGAUUGAGCAGGCUCUGCGGCUGGGUGGCCAGGCCACCGCCGCUGGCCGAGAUUGGUAUGCCGCUGGCGUUCACCAUGUUCAC---GCCGCCGGCACCAGCCACUCCGCCCGCUGCUCC ......((((((.....(((((..((((((...))))))))))).(((.(((..((((.(((((.((((((..((...))..))---)))).)))))...))))))).)))..)))))). ( -57.90) >consensus CGGAUUGAGCAGGCUCUGCGGCUGGGUGGCCAGGCCACCGCCGCUGGCCGAGAUGGGUAUGCCGCUGGCAUUCACCAUGCUCAC___GCCACCGGC________________________ .((..(((((.((((..(((((..((((((...))))))))))).))))...((((..(((((...)))))...))))))))).....)).............................. (-37.09 = -37.78 + 0.70)

| Location | 13,799,019 – 13,799,136 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 76.09 |
| Mean single sequence MFE | -53.43 |
| Consensus MFE | -33.35 |
| Energy contribution | -33.77 |
| Covariance contribution | 0.42 |
| Combinations/Pair | 1.08 |
| Mean z-score | -2.23 |
| Structure conservation index | 0.62 |
| SVM decision value | 1.78 |
| SVM RNA-class probability | 0.977054 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 13799019 117 - 20766785 GCGUCUGUUGGCGGUAUUGGUGGAGCCGGUGGA---GUGAACAUGGUGAACGCCAGUGGCAUCCCCAUAUCGGCCAGCGGCGGUGGCCUGGCCACGCAGCCGCAGAGCCUGCUCAAUCCG (((.(((((((((((((.((.(((((((.(((.---((..((...))..)).))).)))).))))))))))(((((.(...).)))))..)))).)))).))).(((....)))...... ( -53.00) >DroVir_CAF1 122146 96 - 1 ------------------------GCUGGGGGCAUGGUGAGCAUGGUGAAUGCCAGCGGCAUACCCAUCUCGGCCAGCGGCGGUGGCCUGGCCACCCAGCCGCAGAGCCUGCUCAACCCA ------------------------...(.((((.(((((((.((((.(.(((((...))))).)))))))).))))(((((((((((...))))))..)))))...)))).)........ ( -46.80) >DroGri_CAF1 118362 96 - 1 ------------------------GCCGGUGGAAUUGUGAGCAUGGUCAAUGCCAGCGGCAUACCAAUUUCGGCCAGCGGCGGUGGCCUGGCCACCCAGCCGCAGAGCCUGCUCAAUCCG ------------------------......(((....((((((((((..(((((...))))))))).....((((.(((((((((((...))))))..))))).).))))))))).))). ( -45.00) >DroWil_CAF1 123156 117 - 1 GCGGGCGGAGCCGGUGCCGGCGGAGUGGGGGGC---GUCAAUAUGGUGAAUGCCAGUGGAAUUCCCAUAUCGGCCAGCGGCGGUGGCCUGGCCACACAGCCGCAGAGCCUGCUCAAUCCG .((((..((((.(((..((((.(((((((((..---.(((...((((....)))).)))..))))))).)).))).(((((.(((((...)))))...))))).).))).))))..)))) ( -59.60) >DroMoj_CAF1 136065 96 - 1 ------------------------GCCGGAGGCAUGGUGAGCAUGGUGAAUGCCAGCGGCAUUCCCAUCUCGGCCAGCGGCGGUGGCCUGGCCACCCAGCCGCAGAGCCUGCUCAAUCCA ------------------------...(.((((.(((((((.((((.(((((((...)))))))))))))).))))(((((((((((...))))))..)))))...)))).)........ ( -51.50) >DroAna_CAF1 108612 117 - 1 GGAGCAGCGGGCGGAGUGGCUGGUGCCGGCGGC---GUGAACAUGGUGAACGCCAGCGGCAUACCAAUCUCGGCCAGCGGCGGUGGCCUGGCCACCCAGCCGCAGAGCCUGCUCAAUCCG (((..(((((((...(((((((((((((..(((---((..((...))..)))))..)))))..........(((((..(((....))))))))..))))))))...)))))))...))). ( -64.70) >consensus ________________________GCCGGGGGC___GUGAACAUGGUGAAUGCCAGCGGCAUACCCAUCUCGGCCAGCGGCGGUGGCCUGGCCACCCAGCCGCAGAGCCUGCUCAAUCCG ........................(((((.......(((....((((....))))....))).......)))))..(((((((((((...))))))..))))).(((....)))...... (-33.35 = -33.77 + 0.42)

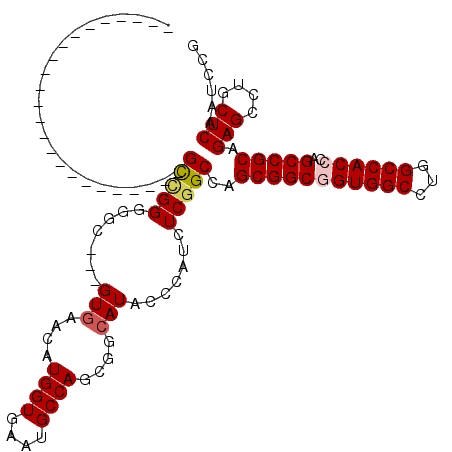
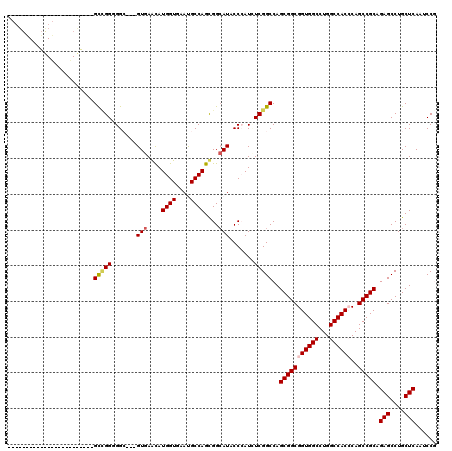
| Location | 13,799,059 – 13,799,176 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 80.62 |
| Mean single sequence MFE | -44.53 |
| Consensus MFE | -31.61 |
| Energy contribution | -32.00 |
| Covariance contribution | 0.39 |
| Combinations/Pair | 1.14 |
| Mean z-score | -1.39 |
| Structure conservation index | 0.71 |
| SVM decision value | 0.25 |
| SVM RNA-class probability | 0.652780 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 13799059 117 + 20766785 CCGCUGGCCGAUAUGGGGAUGCCACUGGCGUUCACCAUGUUCAC---UCCACCGGCUCCACCAAUACCGCCAACAGACGCUCCAUCCGAGGCGUCGCCGUUAAGCGGUGAAGGCGCCUCA (((.(((..((((((((((((((...))))))).)))))))...---.))).)))............((((....(((((..(....)..)))))((((.....))))...))))..... ( -45.20) >DroVir_CAF1 122186 96 + 1 CCGCUGGCCGAGAUGGGUAUGCCGCUGGCAUUCACCAUGCUCACCAUGCCCCCAGC------------------------GCCAUCGGCGGCAUCGCCGUUCAGUGGCGAGGGCGCCUCA .....(((.(((((((..(((((...)))))...)))).))).....((((....(------------------------((((((((((....)))))....)))))).)))))))... ( -44.10) >DroSec_CAF1 99836 117 + 1 CCGCUGGCCGAUAUGGGUAUGCCACUGGCGUUCACCAUGUUCAC---UCCACCGGCCCCACUAAUACCGCCACCAGAUGCUCCAUCCGAGGCGUCGCCGUUAAGCGGUGAAGGGGCCUCA ..(.(((..(((((((..(((((...)))))...)))))))...---.))).)((((((........((((.(..((((...)))).).))))((((((.....)))))).))))))... ( -47.20) >DroSim_CAF1 103709 117 + 1 CCGCUGGCCGAUAUGGGUAUGCCACUGGCGUUCACCAUGUUCAC---UCCACCGGCCCCACCAAUACCGCCACCAGAUGCUCCAUCCGAGGCGUCGCCGUUAAGCGGCGAAGGCGCCUCA ...((((..(((((((..(((((...)))))...)))))))...---......(((............))).))))...........((((((((((((.....)))))...))))))). ( -43.20) >DroYak_CAF1 104444 117 + 1 CCGCUGGCCGAUAUGGGGAUGCCACUGGCGUUCACCAUGUUCAC---UCCACCAGCCCCACCAAUACCGCCACCAGCUGCUCCAUCCGAGGCGUCGCCAUUAAGCGGUGAGGGCGCCUCG .....(((.((((((((((((((...))))))).)))))))...---.......((((((((.....((((..................))))..((......)))))).)))))))... ( -42.37) >DroAna_CAF1 108652 117 + 1 CCGCUGGCCGAGAUUGGUAUGCCGCUGGCGUUCACCAUGUUCAC---GCCGCCGGCACCAGCCACUCCGCCCGCUGCUCCUCCGUCAGAUGCAUCGCCGUUAAGUGGCGAUGGCGCCUCG ..((.(((.(((..((((.(((((.((((((..((...))..))---)))).)))))...))))))).))).))............((.((((((((((.....))))))).))).)).. ( -45.10) >consensus CCGCUGGCCGAUAUGGGUAUGCCACUGGCGUUCACCAUGUUCAC___UCCACCGGCCCCACCAAUACCGCCACCAGAUGCUCCAUCCGAGGCGUCGCCGUUAAGCGGCGAAGGCGCCUCA ..(((((..(((((((..(((((...)))))...)))))))..........)))))...............................(((((.((((((.....))))))....))))). (-31.61 = -32.00 + 0.39)
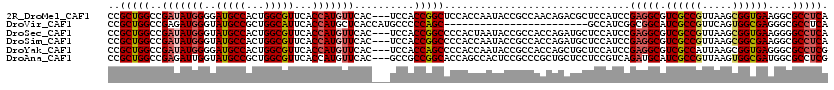


| Location | 13,799,099 – 13,799,216 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 78.76 |
| Mean single sequence MFE | -51.02 |
| Consensus MFE | -30.99 |
| Energy contribution | -32.13 |
| Covariance contribution | 1.15 |
| Combinations/Pair | 1.36 |
| Mean z-score | -1.77 |
| Structure conservation index | 0.61 |
| SVM decision value | 0.53 |
| SVM RNA-class probability | 0.769944 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 13799099 117 - 20766785 CAUUUUUGCCGCCACGGCCAGAGAAGCGCAAGUCGAAGGAUGAGGCGCCUUCACCGCUUAACGGCGACGCCUCGGAUGGAGCGUCUGUUGGCGGUAUUGGUGGAGCCGGUGGA---GUGA ((((.(..(((.(.(.(((((.(((((((..(((....)))...))).))))((((((.((((..(((((..(....)..))))))))))))))).))))).).).)))..))---))). ( -50.90) >DroGri_CAF1 118442 96 - 1 CAUUUUUGCCGCCACGGCCUGAGAAGCGCAAAUCAAAGGACGAGGCGCCGUCACCGCUCAACGGCGAUGGCGCCGAUGGU------------------------GCCGGUGGAAUUGUGA ((.(((..(((.((((.(((..((........))..))).)..(((((((((.(((.....))).)))))))))....))------------------------).)))..))).))... ( -41.90) >DroSim_CAF1 103749 117 - 1 CAUUUUUACCGCCUCGGCCUGAGAAGCGCAAGUCGAAGGAUGAGGCGCCUUCGCCGCUUAACGGCGACGCCUCGGAUGGAGCAUCUGGUGGCGGUAUUGGUGGGGCCGGUGGA---GUGA ((((((.(((((((((.(((..(..((....)))..))).))))))((((((((((.....))))))(((((((((((...))))))).)))).........)))).))))))---))). ( -53.80) >DroEre_CAF1 100312 117 - 1 CAUUUUUGCCUCCGCGGCCGGAGAAGCGCAAGUCAAAGGACGAGGCGCCUUCACCGCUUAACGGCGACGCCUCAGAUGGAGCAUCUGGUGGCGGUAUUGGCGGUGCUGGUGGA---GUGA ((((.(..((..(((.(((((.(((((((..(((....)))...))).)))).))(((....)))..(((((((((((...))))))).)))).....))).)))..))..))---))). ( -51.10) >DroYak_CAF1 104484 117 - 1 CAUUUUUGCCACCUCGGCCGGAGAAGCGCAAGUCGAAGGACGAGGCGCCCUCACCGCUUAAUGGCGACGCCUCGGAUGGAGCAGCUGGUGGCGGUAUUGGUGGGGCUGGUGGA---GUGA ((((.(..(((.(((.(((((((..((((..(((....)))...)))).)))((((((...((((..(.((......)).)..))))..))))))..)))).))).)))..))---))). ( -52.30) >DroAna_CAF1 108692 117 - 1 CCUUCCUGCCGCCGCGAGCGGAGAAGCGCAAGUCCAAGGACGAGGCGCCAUCGCCACUUAACGGCGAUGCAUCUGACGGAGGAGCAGCGGGCGGAGUGGCUGGUGCCGGCGGC---GUGA ...((.(((((((....(((......)))..(((....)))..(((((((..(((((((..(.((..(((.(((......)))))))).)...))))))))))))))))))))---).)) ( -56.10) >consensus CAUUUUUGCCGCCACGGCCGGAGAAGCGCAAGUCGAAGGACGAGGCGCCUUCACCGCUUAACGGCGACGCCUCGGAUGGAGCAUCUGGUGGCGGUAUUGGUGGGGCCGGUGGA___GUGA (((.(((((((.(((.(((.((...((((..(((....)))...))))..))((((((...((....)).(((.....)))........))))))...))).))).)))))))...))). (-30.99 = -32.13 + 1.15)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:14:01 2006