
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 13,678,215 – 13,678,350 |
| Length | 135 |
| Max. P | 0.775591 |

| Location | 13,678,215 – 13,678,335 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 83.33 |
| Mean single sequence MFE | -48.87 |
| Consensus MFE | -33.29 |
| Energy contribution | -34.02 |
| Covariance contribution | 0.72 |
| Combinations/Pair | 1.29 |
| Mean z-score | -1.79 |
| Structure conservation index | 0.68 |
| SVM decision value | 0.00 |
| SVM RNA-class probability | 0.534546 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
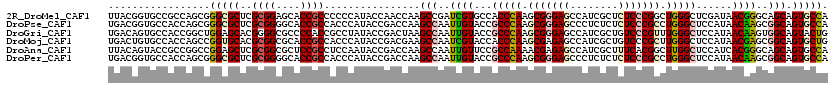
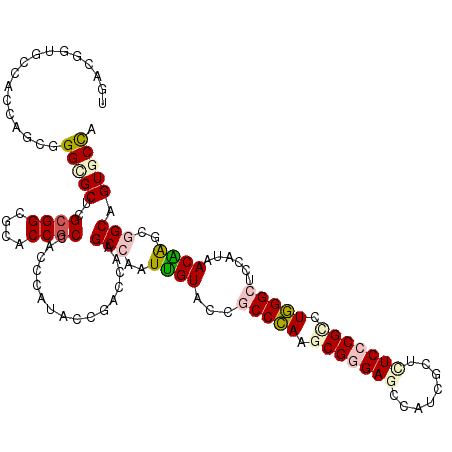
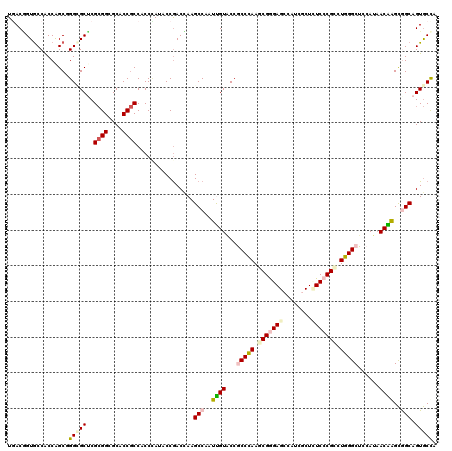
>2R_DroMel_CAF1 13678215 120 + 20766785 UUACGGUGCCGCCAGCGGGCGCUCGCGGAGCACCGCCCCCCAUACCAACCAAGCCGAUCGUGCCACCCAAGCGGGAGCCAUCGCUCUCCCGGCUGGGCUCGAUAACGGGCAGCAGUGCCA ....((..(.((..((((..((((...)))).))))................(((.((((.(((.....((((((((........))))).))).))).))))....))).)).)..)). ( -47.50) >DroPse_CAF1 2310 120 + 1 UGACGGUGCCACCAGCGGGCGCUCGCGGGGCACCGCCACCCAUACCGACCAAGCCAAUUGUACCGCCCAAGCGGGAGCCCUCUCUCUCCCGCCUGGGCUCCAUAACAAGCGGCAGUGCCA ((.(((((((.((.((((....))))))))))))).))..............(((..((((...(((((.(((((((........))))))).)))))......))))..)))....... ( -53.50) >DroGri_CAF1 2483 120 + 1 UGACAGUGCCACCGGCUGGAGCACGGGGCGCCCCACCGCCUAUACCGACUAAGCCAAUUGUACCGCCCAAGCGGGAGCCAUCGCUGUCCCGUUUGGGCUCCAUAACAAGUGGCAGUACUG ...((((((..(((((....)).)))((((......))))............((((.((((...((((((((((((((....))..))))))))))))......)))).)))).)))))) ( -51.00) >DroMoj_CAF1 2719 120 + 1 UGACUGUGCCACCAGCCGGUGCACGCGGCGCACCGCCACCCAUACCGACGAAGCCAAUCGUACCACCCAAGCGAGAGCCAUCGCUGUCCCGCUUGGGCUCCAUAACGAGCGGCAGUGCUG .(.(((((.((((....))))..))))))((((.(((..........((((......)))).....((((((((.(((....))).))..))))))((((......))))))).)))).. ( -44.30) >DroAna_CAF1 2409 120 + 1 UUACAGUACCGCCGGCCGGAGCUCGCGGCGCUCCGCCUCCAAUACCGACCAAGCCAAUUGUUCCGCCAAAACGAGAGCCAUCGCUUUCACGGCUUGGCUCCAUCACGGGCAGCAGUGCCA .....((((.((.(.((((((...((((....))))))))......(((((((((..(((......)))...((((((....))))))..))))))).))......)).).)).)))).. ( -43.40) >DroPer_CAF1 2311 120 + 1 UGACGGUGCCACCAGCGGGCGCUCGCGGGGCACCGCCACCCAUACCGACCAAGCCAAUUGUACCGCCCAAGCGGGAGCCCUCUCUCUCCCGCCUGGGCUCCAUAACAAGCGGCAGUGCCA ((.(((((((.((.((((....))))))))))))).))..............(((..((((...(((((.(((((((........))))))).)))))......))))..)))....... ( -53.50) >consensus UGACGGUGCCACCAGCGGGCGCUCGCGGCGCACCGCCACCCAUACCGACCAAGCCAAUUGUACCGCCCAAGCGGGAGCCAUCGCUCUCCCGCCUGGGCUCCAUAACAAGCGGCAGUGCCA .................(((((..((((....))))................(((..((((...(((((.(((((((........))))))).)))))......))))..))).))))). (-33.29 = -34.02 + 0.72)
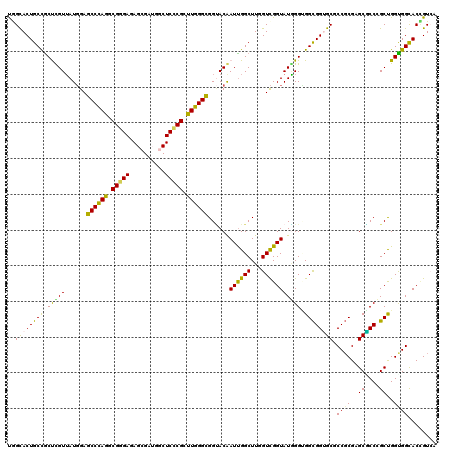
| Location | 13,678,215 – 13,678,335 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 83.33 |
| Mean single sequence MFE | -62.67 |
| Consensus MFE | -43.39 |
| Energy contribution | -41.95 |
| Covariance contribution | -1.44 |
| Combinations/Pair | 1.36 |
| Mean z-score | -2.65 |
| Structure conservation index | 0.69 |
| SVM decision value | 0.54 |
| SVM RNA-class probability | 0.775591 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
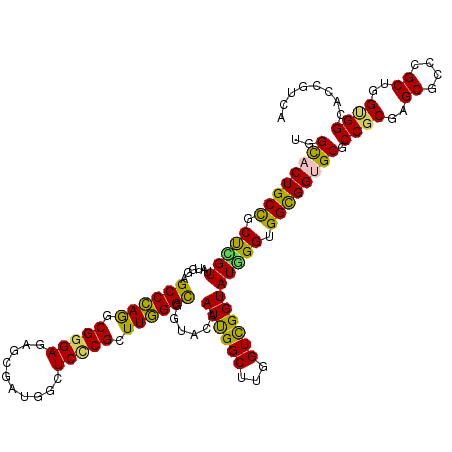
>2R_DroMel_CAF1 13678215 120 - 20766785 UGGCACUGCUGCCCGUUAUCGAGCCCAGCCGGGAGAGCGAUGGCUCCCGCUUGGGUGGCACGAUCGGCUUGGUUGGUAUGGGGGGCGGUGCUCCGCGAGCGCCCGCUGGCGGCACCGUAA .(((((((((.(((((...(.((((.(((((((.((((....))))))(((.....))).....))))).)))).).))))).)))))))))((((.(((....))).))))........ ( -64.90) >DroPse_CAF1 2310 120 - 1 UGGCACUGCCGCUUGUUAUGGAGCCCAGGCGGGAGAGAGAGGGCUCCCGCUUGGGCGGUACAAUUGGCUUGGUCGGUAUGGGUGGCGGUGCCCCGCGAGCGCCCGCUGGUGGCACCGUCA .((((((((((((..(.((.(((((((((((((((........)))))))))))))....(((.....))).)).)))..))))))))))))((((.(((....))).))))........ ( -68.30) >DroGri_CAF1 2483 120 - 1 CAGUACUGCCACUUGUUAUGGAGCCCAAACGGGACAGCGAUGGCUCCCGCUUGGGCGGUACAAUUGGCUUAGUCGGUAUAGGCGGUGGGGCGCCCCGUGCUCCAGCCGGUGGCACUGUCA ((((...((((((.((..((((((((((.(((((..((....))))))).)))))).((((.....(((((.......)))))...(((....))))))))))))).))))))))))... ( -53.10) >DroMoj_CAF1 2719 120 - 1 CAGCACUGCCGCUCGUUAUGGAGCCCAAGCGGGACAGCGAUGGCUCUCGCUUGGGUGGUACGAUUGGCUUCGUCGGUAUGGGUGGCGGUGCGCCGCGUGCACCGGCUGGUGGCACAGUCA ..((((((((((((((.((.((((((((((((((..((....))))))))))))))....(((......))))).))))))))))))))))(((((..((....))..)))))....... ( -63.10) >DroAna_CAF1 2409 120 - 1 UGGCACUGCUGCCCGUGAUGGAGCCAAGCCGUGAAAGCGAUGGCUCUCGUUUUGGCGGAACAAUUGGCUUGGUCGGUAUUGGAGGCGGAGCGCCGCGAGCUCCGGCCGGCGGUACUGUAA ..(((.(((((((...(((.(((((((.((((.(((((((......))))))).)))).....))))))).)))(((...((((((((....))))...)))).)))))))))).))).. ( -58.30) >DroPer_CAF1 2311 120 - 1 UGGCACUGCCGCUUGUUAUGGAGCCCAGGCGGGAGAGAGAGGGCUCCCGCUUGGGCGGUACAAUUGGCUUGGUCGGUAUGGGUGGCGGUGCCCCGCGAGCGCCCGCUGGUGGCACCGUCA .((((((((((((..(.((.(((((((((((((((........)))))))))))))....(((.....))).)).)))..))))))))))))((((.(((....))).))))........ ( -68.30) >consensus UGGCACUGCCGCUCGUUAUGGAGCCCAGGCGGGAGAGCGAUGGCUCCCGCUUGGGCGGUACAAUUGGCUUGGUCGGUAUGGGUGGCGGUGCGCCGCGAGCGCCCGCUGGUGGCACCGUCA ..((((((((.(((((......((((((.(((((..........))))).))))))......((((((...))))))))))).)))))))).((((..((....))..))))........ (-43.39 = -41.95 + -1.44)

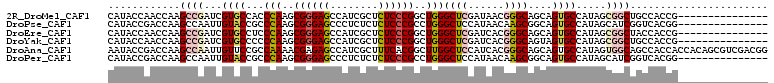
| Location | 13,678,255 – 13,678,350 |
|---|---|
| Length | 95 |
| Sequences | 6 |
| Columns | 110 |
| Reading direction | forward |
| Mean pairwise identity | 80.73 |
| Mean single sequence MFE | -37.80 |
| Consensus MFE | -21.14 |
| Energy contribution | -21.83 |
| Covariance contribution | 0.70 |
| Combinations/Pair | 1.24 |
| Mean z-score | -2.25 |
| Structure conservation index | 0.56 |
| SVM decision value | 0.44 |
| SVM RNA-class probability | 0.738840 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 13678255 95 + 20766785 CAUACCAACCAAGCCGAUCGUGCCACCCAAGCGGGAGCCAUCGCUCUCCCGGCUGGGCUCGAUAACGGGCAGCAGUGCCAUAGCGGCUGCCACCG--------------- ..............((((((.(((.....((((((((........))))).))).))).))))..))((((((.((......)).))))))....--------------- ( -34.70) >DroPse_CAF1 2350 95 + 1 CAUACCGACCAAGCCAAUUGUACCGCCCAAGCGGGAGCCCUCUCUCUCCCGCCUGGGCUCCAUAACAAGCGGCAGUGCCAUAGCAUCGGUCACGG--------------- ......((((..(((..((((...(((((.(((((((........))))))).)))))......))))..))).((((....)))).))))....--------------- ( -40.40) >DroEre_CAF1 2426 95 + 1 CAUACCAACCAAGCCGAUCGUGCCUCCCAAGCGGGAGCCAUCGCUCUCCCGGCUGGGCUCGAUCACGGGCAGCAGUGCCAUAGCGGCUACCACCG--------------- ...........(((((((((.((((....((((((((........))))).))))))).)))))...((((....)))).....)))).......--------------- ( -36.70) >DroYak_CAF1 2422 95 + 1 CAUACCAACCAAGCCGAUCGUGCCCCCCAAGCGGGAGCCAUCGCUCUCCCGGCUGGGCUCGAUCACGGGCAGUAGUGCCAUAGCGGCUGCCACCG--------------- ...............(((((.((((....((((((((........))))).))))))).)))))...((((((.((......)).))))))....--------------- ( -38.50) >DroAna_CAF1 2449 110 + 1 AAUACCGACCAAGCCAAUUGUUCCGCCAAAACGAGAGCCAUCGCUUUCACGGCUUGGCUCCAUCACGGGCAGCAGUGCCAUAGUGGCAGCCACCACCACAGCGUCGACGG .....((((((((((..(((......)))...((((((....))))))..)))))((((((((....((((....))))...)))).)))).........).)))).... ( -36.10) >DroPer_CAF1 2351 95 + 1 CAUACCGACCAAGCCAAUUGUACCGCCCAAGCGGGAGCCCUCUCUCUCCCGCCUGGGCUCCAUAACAAGCGGCAGUGCCAUAGCAUCGGUCACGG--------------- ......((((..(((..((((...(((((.(((((((........))))))).)))))......))))..))).((((....)))).))))....--------------- ( -40.40) >consensus CAUACCAACCAAGCCAAUCGUGCCGCCCAAGCGGGAGCCAUCGCUCUCCCGGCUGGGCUCCAUAACGGGCAGCAGUGCCAUAGCGGCUGCCACCG_______________ ............((((...((((...(((..((((((........))))))..)))((((......))))....)))).....))))....................... (-21.14 = -21.83 + 0.70)
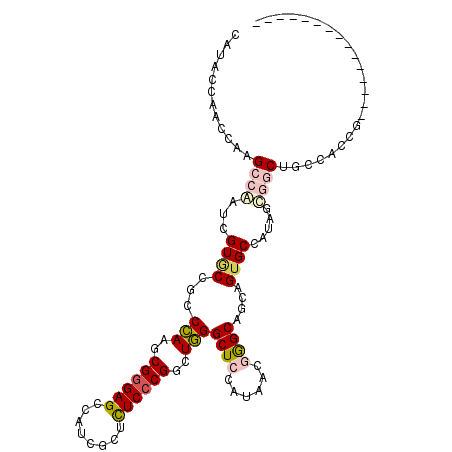
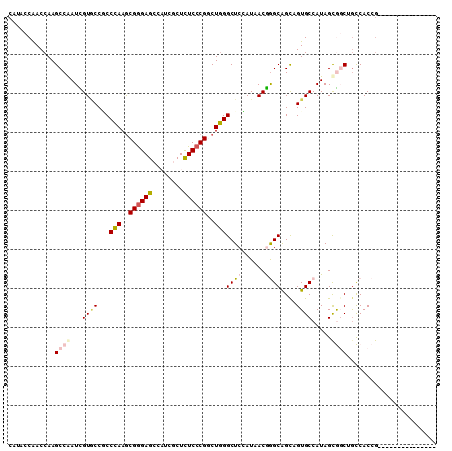
| Location | 13,678,255 – 13,678,350 |
|---|---|
| Length | 95 |
| Sequences | 6 |
| Columns | 110 |
| Reading direction | reverse |
| Mean pairwise identity | 80.73 |
| Mean single sequence MFE | -45.07 |
| Consensus MFE | -28.81 |
| Energy contribution | -28.03 |
| Covariance contribution | -0.77 |
| Combinations/Pair | 1.31 |
| Mean z-score | -1.83 |
| Structure conservation index | 0.64 |
| SVM decision value | 0.42 |
| SVM RNA-class probability | 0.730147 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 13678255 95 - 20766785 ---------------CGGUGGCAGCCGCUAUGGCACUGCUGCCCGUUAUCGAGCCCAGCCGGGAGAGCGAUGGCUCCCGCUUGGGUGGCACGAUCGGCUUGGUUGGUAUG ---------------.((..((((..((....)).))))..))...((((.((((.(((((((.((((....))))))(((.....))).....))))).)))))))).. ( -41.50) >DroPse_CAF1 2350 95 - 1 ---------------CCGUGACCGAUGCUAUGGCACUGCCGCUUGUUAUGGAGCCCAGGCGGGAGAGAGAGGGCUCCCGCUUGGGCGGUACAAUUGGCUUGGUCGGUAUG ---------------...((((((((((....)))..((((.((((......(((((((((((((........)))))))))))))...)))).)))))))))))..... ( -47.50) >DroEre_CAF1 2426 95 - 1 ---------------CGGUGGUAGCCGCUAUGGCACUGCUGCCCGUGAUCGAGCCCAGCCGGGAGAGCGAUGGCUCCCGCUUGGGAGGCACGAUCGGCUUGGUUGGUAUG ---------------.((..((((..((....)).))))..))...........(((((((((.((.((.((.(((((....))))).)))).))..))))))))).... ( -41.50) >DroYak_CAF1 2422 95 - 1 ---------------CGGUGGCAGCCGCUAUGGCACUACUGCCCGUGAUCGAGCCCAGCCGGGAGAGCGAUGGCUCCCGCUUGGGGGGCACGAUCGGCUUGGUUGGUAUG ---------------..(((.((((((((..((((....))))...(((((.((((..(((((.((((....))))...))))).)))).))))))))..))))).))). ( -43.70) >DroAna_CAF1 2449 110 - 1 CCGUCGACGCUGUGGUGGUGGCUGCCACUAUGGCACUGCUGCCCGUGAUGGAGCCAAGCCGUGAAAGCGAUGGCUCUCGUUUUGGCGGAACAAUUGGCUUGGUCGGUAUU ........(((((((((((....))))))))))).....((((...(((.(((((((.((((.(((((((......))))))).)))).....))))))).))))))).. ( -48.70) >DroPer_CAF1 2351 95 - 1 ---------------CCGUGACCGAUGCUAUGGCACUGCCGCUUGUUAUGGAGCCCAGGCGGGAGAGAGAGGGCUCCCGCUUGGGCGGUACAAUUGGCUUGGUCGGUAUG ---------------...((((((((((....)))..((((.((((......(((((((((((((........)))))))))))))...)))).)))))))))))..... ( -47.50) >consensus _______________CGGUGGCAGCCGCUAUGGCACUGCUGCCCGUGAUCGAGCCCAGCCGGGAGAGCGAUGGCUCCCGCUUGGGCGGCACAAUCGGCUUGGUCGGUAUG .................(((.....)))..((((...((((..(((......((((((.((((((........)))))).))))))...)))..))))...))))..... (-28.81 = -28.03 + -0.77)

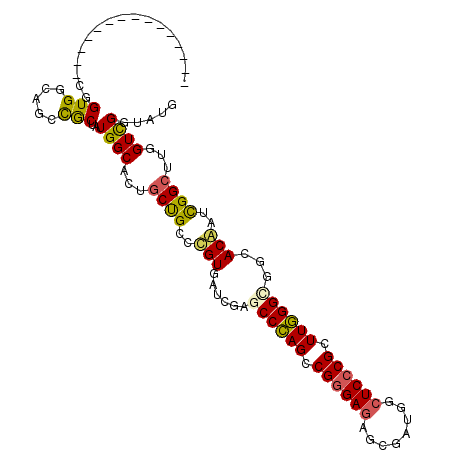
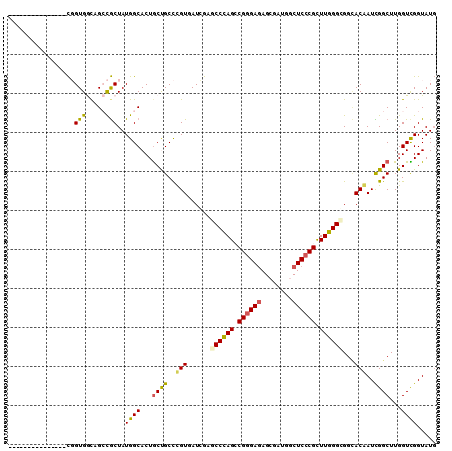
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:13:11 2006