
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 2,443,772 – 2,443,924 |
| Length | 152 |
| Max. P | 0.949212 |

| Location | 2,443,772 – 2,443,884 |
|---|---|
| Length | 112 |
| Sequences | 6 |
| Columns | 115 |
| Reading direction | forward |
| Mean pairwise identity | 78.43 |
| Mean single sequence MFE | -55.35 |
| Consensus MFE | -30.76 |
| Energy contribution | -34.23 |
| Covariance contribution | 3.48 |
| Combinations/Pair | 1.15 |
| Mean z-score | -2.50 |
| Structure conservation index | 0.56 |
| SVM decision value | 1.39 |
| SVM RNA-class probability | 0.949212 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
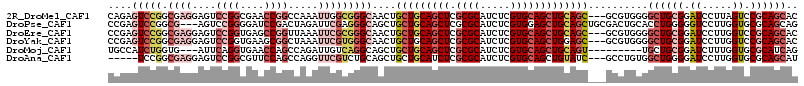
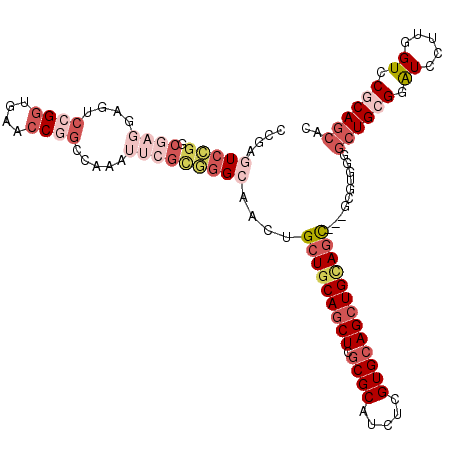
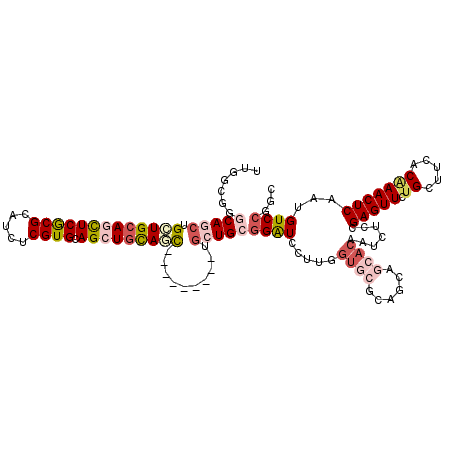
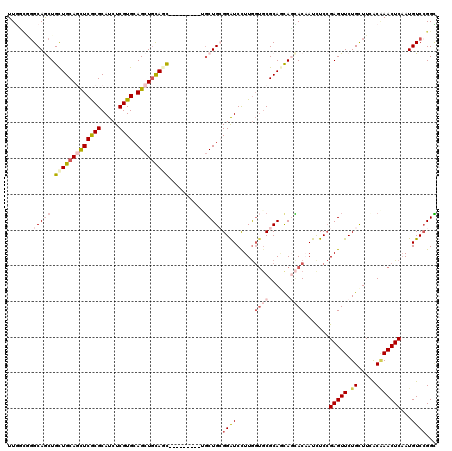
>2R_DroMel_CAF1 2443772 112 + 20766785 CAGAGUCCGGCGAGGAGUCCGGCGAACCGGCCAAAUUGGCGGGCAACUGCUGCAGCUCGCGCAUCUCGUGCAGCUGCAGC---GCGUGGGGCUGCGGAUCCUUAGUCCGCAGCAC ......((.(((((..((((((....)).(((.....)))))))..))(((((((((.((((.....)))))))))))))---.))).))(((((((((.....))))))))).. ( -61.10) >DroPse_CAF1 6075 112 + 1 CCGAGUCCGGCG---AGUCCGGGGAUCCGACUAGAUUCGAGGGCAGCUGCUGCAGCUCGCGCAUCUCGUGGAGCUGCAGCUGCGACUGCACCUGGGGGUCCUUGGUGCGCAGCAG ....((...(((---...((((((((((((......)).((((((((.((((((((((.(((.....)))))))))))))...).)))).)))..))))))))))..))).)).. ( -56.00) >DroEre_CAF1 5720 112 + 1 CCGAGUCCGGCGAGGAGUCCGGUGAGCCGGUUAAAUUCGCGGGCAACUGCUGCAGCUCGCGCAUCUCGUGCAGCUGCAGC---GCGUGGGGCUGCGGAUCCUUGGUCCGCAGCAC ((..(((((.((((....((((....)))).....))))))))).((((((((((((.((((.....)))))))))))))---).)))).(((((((((.....))))))))).. ( -65.00) >DroYak_CAF1 5693 112 + 1 CCGAGUCCGGCGAGGAGUCCGGUGAAGCGGCUAAAUUCGUGGGCAACUGCUGCAGCUCGCGCAUCUCGUGCAGCUGGAGC---GCGUGGGGCUGCGGAUCCUUGGUCCGCAGCAC ((..(((((.((((.((.(((......)))))...))))))))).((((((.(((((.((((.....))))))))).)))---).)))).(((((((((.....))))))))).. ( -56.30) >DroMoj_CAF1 6448 103 + 1 UGCCAUCUGGUG---AUUCAGGUGAACCAGCCAGAUUGUCAGGCAGCUGCUGCAGCUCGCGCAUCUCGUGCAGCUGCAGU---------UGCUGCGGAUCUUUGGUGCGCAUCAG ((((((((((..---..))))))).....((((((..(((..(((((.(((((((((.((((.....)))))))))))))---------.))))).))).))))))..))).... ( -50.50) >DroAna_CAF1 5607 107 + 1 -----UCCGGCGAGGAGUCCGGCGUUCCAGCCAGGUUCGUCUGCAGCUGCUGCAUCUCGCGCAUCUCGUGCAGCUGUAUC---GCCUGUGGCUGGGGAUCCUUGGUGCGCAGCAU -----...(((((.((..(.(((......))).)..))...(((((((((.((.....))((.....)))))))))))))---)))((((((..((....))..)).)))).... ( -43.20) >consensus CCGAGUCCGGCGAGGAGUCCGGUGAACCGGCCAAAUUCGCGGGCAACUGCUGCAGCUCGCGCAUCUCGUGCAGCUGCAGC___GCGUGGGGCUGCGGAUCCUUGGUCCGCAGCAC ....(((((.((((....((((....)))).....)))))))))....(((((((((.((((.....)))))))))))))..........((((((.((.....)).)))))).. (-30.76 = -34.23 + 3.48)
| Location | 2,443,807 – 2,443,924 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 80.14 |
| Mean single sequence MFE | -46.53 |
| Consensus MFE | -24.17 |
| Energy contribution | -25.90 |
| Covariance contribution | 1.73 |
| Combinations/Pair | 1.21 |
| Mean z-score | -2.64 |
| Structure conservation index | 0.52 |
| SVM decision value | 0.87 |
| SVM RNA-class probability | 0.871758 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
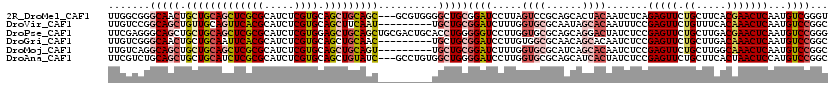
>2R_DroMel_CAF1 2443807 117 + 20766785 UUGGCGGGCAACUGCUGCAGCUCGCGCAUCUCGUGCAGCUGCAGC---GCGUGGGGCUGCGGAUCCUUAGUCCGCAGCACUACAAUCUCAGAGUUCUGCUUCACGAACUCAAUGUCGGGU ((((((((.....(((((((((.((((.....)))))))))))))---..((((.(((((((((.....))))))))).))))...))).((((((........))))))...))))).. ( -58.40) >DroVir_CAF1 6464 111 + 1 UUGUCCGGCAGCUGUUGCAGUUCACGCAUCUCGUGCAGCUUCAAU---------UGCUGCGGAUCUUUGGUGCGCAAUAGCACAAUUUCCGAGUUCUGUUUCACAAACUCAAUGUCCGGC ....((((((((((((((.(....)(((((((.((((((......---------.)))))))).....))))))))))))).........(((((.((.....)))))))..)).)))). ( -39.20) >DroPse_CAF1 6107 120 + 1 UUCGAGGGCAGCUGCUGCAGCUCGCGCAUCUCGUGGAGCUGCAGCUGCGACUGCACCUGGGGGUCCUUGGUGCGCAGCAGGACUAUCUCCGAGUUCUGCUUGACGAACUCAAUGUCCGGG .....(((((((.((((((((((.(((.....))))))))))))).))((..(((((.(((...))).)))))..(((((((((.......)))))))))........))..)))))... ( -57.20) >DroGri_CAF1 6502 111 + 1 UUGUCGGGCAACUGCUGCAAUUCACGCAUCUCGUGCAGCUGCAAC---------UGCUGCGGAUCCUUGUGGCGCAACAGCACAAUCUCCGAGUUCUGCUUGACAAACUCAAUGUCCGGC ..((((((((...((..(((.((.((((.(...(((....)))..---------.).))))))...)))..))((....)).........(((((.((.....)))))))..)))))))) ( -35.60) >DroMoj_CAF1 6480 111 + 1 UUGUCAGGCAGCUGCUGCAGCUCGCGCAUCUCGUGCAGCUGCAGU---------UGCUGCGGAUCUUUGGUGCGCAUCAGCACAAUCUCCGAGUUCUGCUUGGCAAACUCAAUGUCCGGC ..(((..(((((.(((((((((.((((.....)))))))))))))---------.)))))((((..(((((((......)))).....(((((.....)))))......))).))))))) ( -51.80) >DroAna_CAF1 5637 117 + 1 UUCGUCUGCAGCUGCUGCAUCUCGCGCAUCUCGUGCAGCUGUAUC---GCCUGUGGCUGGGGAUCCUUGGUGCGCAGCAUCACUAUCUCCGAGUUCUGCUUCACUAACUCCAUGUCCGGC ......(((((((((.((.....))((.....)))))))))))..---(((.(.((((((((....((((((.((((..((.........))...))))..))))))))))).))))))) ( -37.00) >consensus UUGGCGGGCAGCUGCUGCAGCUCGCGCAUCUCGUGCAGCUGCAGC_________UGCUGCGGAUCCUUGGUGCGCAGCAGCACAAUCUCCGAGUUCUGCUUCACAAACUCAAUGUCCGGC .......(((((.(((((((((((((.....)))).)))))))))..........)))))((((.....((((......)))).......(((((.((.....)))))))...))))... (-24.17 = -25.90 + 1.73)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:38:03 2006