
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 2,440,273 – 2,440,378 |
| Length | 105 |
| Max. P | 0.524181 |

| Location | 2,440,273 – 2,440,378 |
|---|---|
| Length | 105 |
| Sequences | 6 |
| Columns | 110 |
| Reading direction | forward |
| Mean pairwise identity | 81.52 |
| Mean single sequence MFE | -33.90 |
| Consensus MFE | -22.47 |
| Energy contribution | -22.08 |
| Covariance contribution | -0.38 |
| Combinations/Pair | 1.35 |
| Mean z-score | -1.42 |
| Structure conservation index | 0.66 |
| SVM decision value | -0.02 |
| SVM RNA-class probability | 0.524181 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 2440273 105 + 20766785 AAAUUCUAUGCUGUUGCUGGUGCUGCAGC---UGGAUUUGUUCAGAUUUACCGUCAAAGUGUUGCUGCUGUU--GCGGCUGCGACAUGGACAGGCCACUCCGCAUGUAGA ....((((((((((.((....)).)))))---(((.(((((((.(((.....)))...(((((((.((((..--.)))).)))))))))))))))))........))))) ( -35.50) >DroVir_CAF1 2954 99 + 1 AAAUUCUAUGUUGCUGCUGU-GU--CAGC---U---UAUGUUCAAAUUUACCGUCGUGGUGUUGCUGCUG--CUGCUGCUGUGUCAUGGACAAACCGCUGCGCAGGUAGA .............(((((((-(.--((((---.---..((((((....(((.((.(..((((....)).)--)..).)).)))...))))))....)))))))).)))). ( -27.20) >DroPse_CAF1 2476 108 + 1 AAAUUCUAUGCUGCUGCUGUUGUUGCAGUGGCUGUUGGUGCUCAGAUUUACCGUCAUAGUGUUGCUGCUGUU--GCGGCUGGGACAUGGACAGGCCGCUCCGCGUGUAGA ....(((((((.(((((.......)))(((((((((.((((((((.....((((.(((((......))))).--))))))))).))).)))).)))))...))))))))) ( -42.00) >DroEre_CAF1 2274 105 + 1 AAAUUCUAUGCUGUUGCUGGUGCUGCAGC---UGGAUUUGGUCAGAUUUACCGUCAUAGUGUUGCUGCUGUU--GCGGCUGCGACAUGGACAAGCCACUCCGCAUGUAGA ....((((((((((.((....)).)))))---.(((..((((..(......)(((...(((((((.((((..--.)))).))))))).)))..)))).)))....))))) ( -35.60) >DroYak_CAF1 2295 105 + 1 AAAUUCUAUGCUGUUGCUGGUGCUGCAGC---UGGAUUUGUUCAGAUUUACCGUCAUAGUGUUGCUGCUGUU--GCGGCUGCGACAUGGGCAAGCCACUCCGCAUGUAGA ....((((((((((.((....)).)))))---(((.(((((((.(((.....)))...(((((((.((((..--.)))).)))))))))))))))))........))))) ( -35.40) >DroAna_CAF1 2374 99 + 1 AAAUUCUAUGCUGUUGAUGUUGC---AUU---UG---CUGUUCAGAUUUACCGUCAAAGUGUUGCUGCUGAG--GCGUUUGCGACAUGGGCAAACCUCCUCGCAUAUAAA ......(((((((((.(((((((---(..---((---((..((((....(((......).)).....)))))--)))..)))))))).)))).........))))).... ( -27.70) >consensus AAAUUCUAUGCUGUUGCUGGUGCUGCAGC___UGGAUUUGUUCAGAUUUACCGUCAUAGUGUUGCUGCUGUU__GCGGCUGCGACAUGGACAAGCCACUCCGCAUGUAGA ......(((((....((((......)))).......................(((...(((((((.((((.....)))).))))))).)))..........))))).... (-22.47 = -22.08 + -0.38)

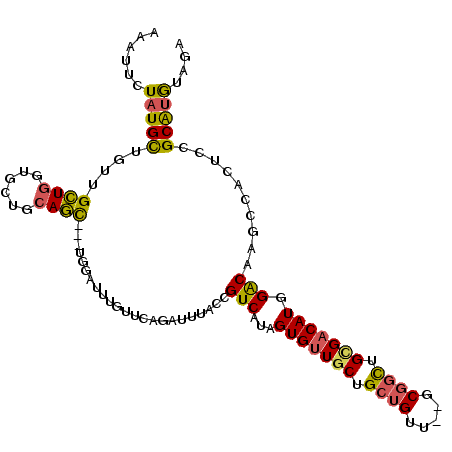
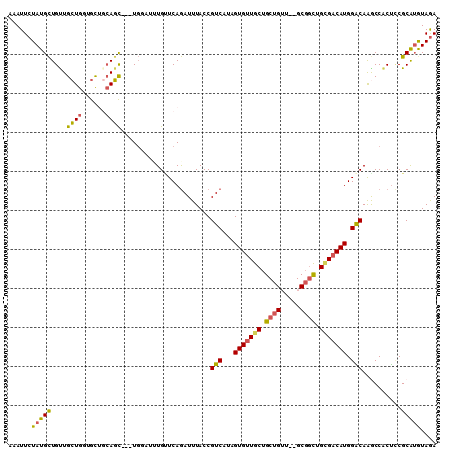
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:38:01 2006