
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 13,529,681 – 13,529,814 |
| Length | 133 |
| Max. P | 0.996631 |

| Location | 13,529,681 – 13,529,774 |
|---|---|
| Length | 93 |
| Sequences | 6 |
| Columns | 101 |
| Reading direction | reverse |
| Mean pairwise identity | 87.65 |
| Mean single sequence MFE | -31.87 |
| Consensus MFE | -28.33 |
| Energy contribution | -28.03 |
| Covariance contribution | -0.30 |
| Combinations/Pair | 1.21 |
| Mean z-score | -2.08 |
| Structure conservation index | 0.89 |
| SVM decision value | 1.32 |
| SVM RNA-class probability | 0.941833 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
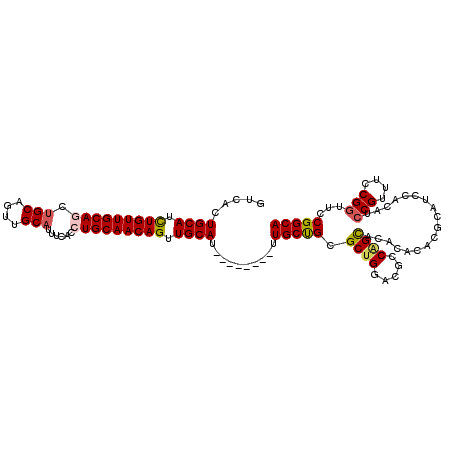
>2R_DroMel_CAF1 13529681 93 - 20766785 AACAGUUGCAU--------UUGCUGCGCUGGACGCCAGCACACACACGCAUCCACAUCCGUUUCCGGUUCCGGCAGCAGCCAAGCCACGAAUCGGAAGCGA ......(((..--------.((.((.(((((...))))).))))...)))........((((((((((((.(((.........)))..)))))))))))). ( -33.00) >DroSec_CAF1 78089 93 - 1 AACAGUUGCAU--------UUGCUGCGCUGGACGCCAGCACACACACGCAUCCACAUCCGUUUCCGGUUCCGGCAGCAGCCAAGCCACGAAUCGGAAGCGA ......(((..--------.((.((.(((((...))))).))))...)))........((((((((((((.(((.........)))..)))))))))))). ( -33.00) >DroSim_CAF1 80904 93 - 1 AACAGUUGCAU--------UUGCUGCGCUGGACGCCAGCACACACACGCAUCCACAUCCGUUUCCGGUUCCGGCAGCAGCCAAGCCACGAAUCGGAAGCGA ......(((..--------.((.((.(((((...))))).))))...)))........((((((((((((.(((.........)))..)))))))))))). ( -33.00) >DroEre_CAF1 78467 91 - 1 AACAGUUGCAU--------UUGCUGCGCUGGACCCCGGCACA--CACGCAUCCACAUCCGUUUCCGGUUCCGGCAGCAGCACAGCCACGAAUCGGAAGCGA ......(((..--------.((.((.(((((...))))).))--)).)))........((((((((((((.(((.........)))..)))))))))))). ( -31.60) >DroYak_CAF1 81172 91 - 1 AACAGUUGCAU--------UUGCUGCGCUGGACUCCAGCACA--CACGCAUCCACAUCCGUUUCCGGUUCCGGCAGCAGCGUAGCCACGAAUCGGAAGCGA ......(((..--------.((.((.(((((...))))).))--)).)))........((((((((((((.(((.((...)).)))..)))))))))))). ( -32.90) >DroAna_CAF1 78953 99 - 1 AACAGUUGCAGCACUAACCGUGCCGUGCAGA--GCCCGUACGCACACGCAUCCACAUCCGCUUCCGUUUCCGGCAACAGUCAAGCCACAAAACGGAAGAGA ......(((.((((.....)))).((((...--........))))..)))........(.(((((((((..(((.........)))...))))))))).). ( -27.70) >consensus AACAGUUGCAU________UUGCUGCGCUGGACGCCAGCACACACACGCAUCCACAUCCGUUUCCGGUUCCGGCAGCAGCCAAGCCACGAAUCGGAAGCGA ....((.(((..........))).))((((.....))))...................((((((((((((.(((.........)))..)))))))))))). (-28.33 = -28.03 + -0.30)

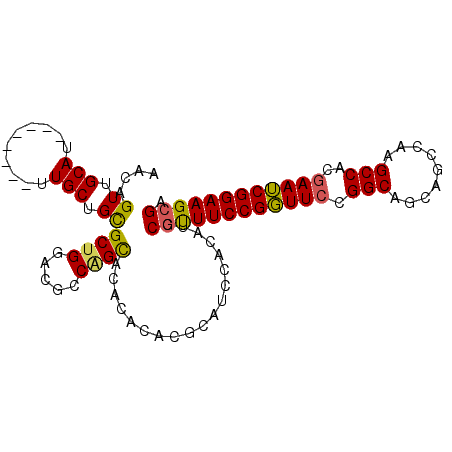
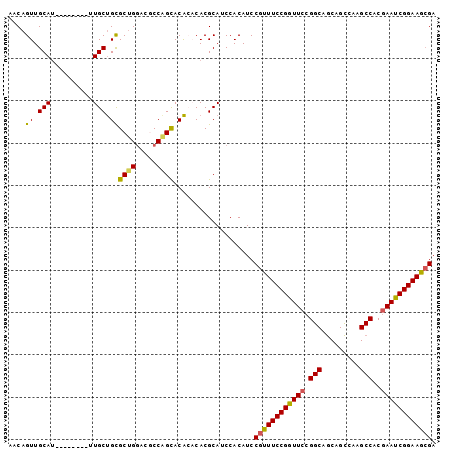
| Location | 13,529,707 – 13,529,814 |
|---|---|
| Length | 107 |
| Sequences | 6 |
| Columns | 115 |
| Reading direction | forward |
| Mean pairwise identity | 87.77 |
| Mean single sequence MFE | -45.53 |
| Consensus MFE | -40.20 |
| Energy contribution | -40.28 |
| Covariance contribution | 0.09 |
| Combinations/Pair | 1.15 |
| Mean z-score | -3.54 |
| Structure conservation index | 0.88 |
| SVM decision value | 2.72 |
| SVM RNA-class probability | 0.996631 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 13529707 107 + 20766785 UGCCGGAACCGGAAACGGAUGUGGAUGCGUGUGUGUGCUGGCGUCCAGCGCAGCAA--------AUGCAACUGUUGCAGGUAAAAUGCAACUGCAGCUGCAACAGAUGCAGUGAC ..(((.(.(((....))).).))).(((((.((((((((((...))))))).(((.--------.((((...((((((.......))))))))))..))).))).)))))..... ( -46.90) >DroSec_CAF1 78115 107 + 1 UGCCGGAACCGGAAACGGAUGUGGAUGCGUGUGUGUGCUGGCGUCCAGCGCAGCAA--------AUGCAACUGUUGCAGGUGAAAUGCAACUGCAGCUGCAACAGAUGCAGUGAC ..(((.(.(((....))).).))).(((((.((((((((((...))))))).(((.--------.((((...((((((.......))))))))))..))).))).)))))..... ( -46.90) >DroSim_CAF1 80930 107 + 1 UGCCGGAACCGGAAACGGAUGUGGAUGCGUGUGUGUGCUGGCGUCCAGCGCAGCAA--------AUGCAACUGUUGCAGGUGAAAUGCAACUGCAGCUGCAACAGAUGCAGUGAC ..(((.(.(((....))).).))).(((((.((((((((((...))))))).(((.--------.((((...((((((.......))))))))))..))).))).)))))..... ( -46.90) >DroEre_CAF1 78493 105 + 1 UGCCGGAACCGGAAACGGAUGUGGAUGCGUG--UGUGCCGGGGUCCAGCGCAGCAA--------AUGCAACUGUUGCAGGUGAAAUGCAACUGCGGGUGCAACAAAUGCAGCGAC ..(((.(.(((....))).).))).(((((.--(((((.((...)).)))))(((.--------.((((...((((((.......))))))))))..))).....)))))..... ( -38.90) >DroYak_CAF1 81198 105 + 1 UGCCGGAACCGGAAACGGAUGUGGAUGCGUG--UGUGCUGGAGUCCAGCGCAGCAA--------AUGCAACUGUUGCAGGUGAAAUGCAAGUGCAGCUGCAACAGAUGCAGUGAC ..(((.(.(((....))).).))).(((...--((((((((...))))))))))).--------.((((.(((((((((.((...........)).))))))))).))))..... ( -46.30) >DroAna_CAF1 78979 108 + 1 UGCCGGAAACGGAAGCGGAUGUGGAUGCGUGUGCGUACGGGC--UCUGCACGGCACGGUUAGUGCUGCAACUGUUGCA-----ACUGCAAGUGCAACUGCAACAGAUGCAGUGAC ..(((....)))..((.(.((..((.((.(((....))).))--))..))).)).........((((((.((((((((-----..(((....)))..)))))))).))))))... ( -47.30) >consensus UGCCGGAACCGGAAACGGAUGUGGAUGCGUGUGUGUGCUGGCGUCCAGCGCAGCAA________AUGCAACUGUUGCAGGUGAAAUGCAACUGCAGCUGCAACAGAUGCAGUGAC ..(((.(.(((....))).).))).(((.....((((((((...)))))))))))..........((((.(((((((((......(((....))).))))))))).))))..... (-40.20 = -40.28 + 0.09)

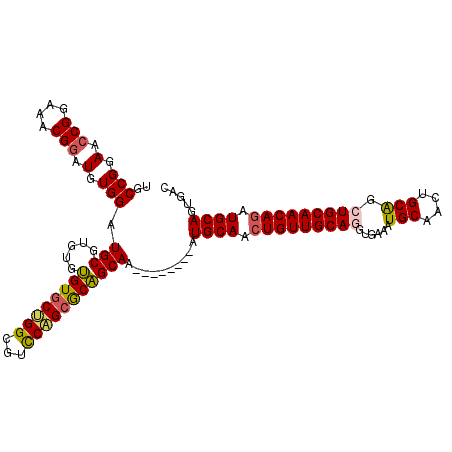
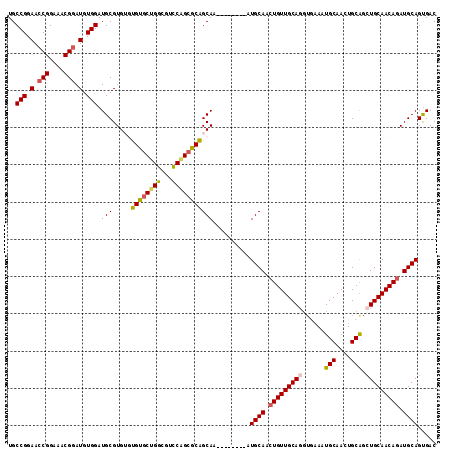
| Location | 13,529,707 – 13,529,814 |
|---|---|
| Length | 107 |
| Sequences | 6 |
| Columns | 115 |
| Reading direction | reverse |
| Mean pairwise identity | 87.77 |
| Mean single sequence MFE | -34.88 |
| Consensus MFE | -29.71 |
| Energy contribution | -30.02 |
| Covariance contribution | 0.31 |
| Combinations/Pair | 1.14 |
| Mean z-score | -2.29 |
| Structure conservation index | 0.85 |
| SVM decision value | 1.05 |
| SVM RNA-class probability | 0.906566 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 13529707 107 - 20766785 GUCACUGCAUCUGUUGCAGCUGCAGUUGCAUUUUACCUGCAACAGUUGCAU--------UUGCUGCGCUGGACGCCAGCACACACACGCAUCCACAUCCGUUUCCGGUUCCGGCA ((.(.((((.(((((((((.(((....)))......))))))))).)))).--------).))...((((((.(((.((........))....((....))....))))))))). ( -34.00) >DroSec_CAF1 78115 107 - 1 GUCACUGCAUCUGUUGCAGCUGCAGUUGCAUUUCACCUGCAACAGUUGCAU--------UUGCUGCGCUGGACGCCAGCACACACACGCAUCCACAUCCGUUUCCGGUUCCGGCA ((.(.((((.(((((((((.(((....)))......))))))))).)))).--------).))...((((((.(((.((........))....((....))....))))))))). ( -34.00) >DroSim_CAF1 80930 107 - 1 GUCACUGCAUCUGUUGCAGCUGCAGUUGCAUUUCACCUGCAACAGUUGCAU--------UUGCUGCGCUGGACGCCAGCACACACACGCAUCCACAUCCGUUUCCGGUUCCGGCA ((.(.((((.(((((((((.(((....)))......))))))))).)))).--------).))...((((((.(((.((........))....((....))....))))))))). ( -34.00) >DroEre_CAF1 78493 105 - 1 GUCGCUGCAUUUGUUGCACCCGCAGUUGCAUUUCACCUGCAACAGUUGCAU--------UUGCUGCGCUGGACCCCGGCACA--CACGCAUCCACAUCCGUUUCCGGUUCCGGCA ...((.(((..((((((....)))((((((.......))))))....))).--------.))).))((((((..((((....--.(((.((....)).)))..)))).)))))). ( -33.90) >DroYak_CAF1 81198 105 - 1 GUCACUGCAUCUGUUGCAGCUGCACUUGCAUUUCACCUGCAACAGUUGCAU--------UUGCUGCGCUGGACUCCAGCACA--CACGCAUCCACAUCCGUUUCCGGUUCCGGCA (((..((((.(((((((((.(((....)))......))))))))).)))).--------.(((((.(((((...))))).))--...))).......(((....)))....))). ( -34.40) >DroAna_CAF1 78979 108 - 1 GUCACUGCAUCUGUUGCAGUUGCACUUGCAGU-----UGCAACAGUUGCAGCACUAACCGUGCCGUGCAGA--GCCCGUACGCACACGCAUCCACAUCCGCUUCCGUUUCCGGCA (((.(((((.((((((((..(((....)))..-----)))))))).)))))........((((.((((...--....))))))))..........................))). ( -39.00) >consensus GUCACUGCAUCUGUUGCAGCUGCAGUUGCAUUUCACCUGCAACAGUUGCAU________UUGCUGCGCUGGACGCCAGCACACACACGCAUCCACAUCCGUUUCCGGUUCCGGCA .....((((.(((((((((.(((....)))......))))))))).))))..........(((((.((((.....))))..................(((....)))...))))) (-29.71 = -30.02 + 0.31)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:12:15 2006