
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 802,520 – 802,690 |
| Length | 170 |
| Max. P | 0.997378 |

| Location | 802,520 – 802,619 |
|---|---|
| Length | 99 |
| Sequences | 6 |
| Columns | 105 |
| Reading direction | forward |
| Mean pairwise identity | 74.98 |
| Mean single sequence MFE | -40.72 |
| Consensus MFE | -22.68 |
| Energy contribution | -22.35 |
| Covariance contribution | -0.33 |
| Combinations/Pair | 1.40 |
| Mean z-score | -2.03 |
| Structure conservation index | 0.56 |
| SVM decision value | 1.29 |
| SVM RNA-class probability | 0.938652 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
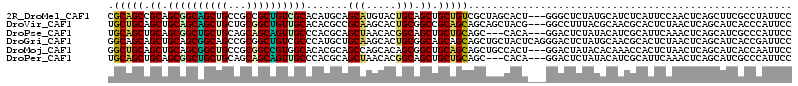
>2R_DroMel_CAF1 802520 99 + 20766785 GCCGUUGAGGGAGAAU---CUGAUCGAAAACGUAGAGCC---GGUGGAAAUAGUGCAAAUACAUCCGCAGCCGCAGCGGCAGCUGCCGCCGCUGUCGCACAUGCA ((.((((.(((.((..---....))......(((..(((---.((....)).).))...))).))).)))).)).((((((((.......))))))))....... ( -28.90) >DroVir_CAF1 1938 102 + 1 GCACCUGAAGGCGAUACUCCGGAUCGAAGAAGGCGCGCC---GCUGGCAAUAAUGCCAACACUUCUGCUGCAGCUGCAGCAGCUGCUGCGGCUGUUGCACACGCC .........((((............((((..((....))---(.(((((....))))).).))))(((.(((((((((((....))))))))))).)))..)))) ( -44.00) >DroPse_CAF1 2616 99 + 1 ACUGCUGAUGGGGAAA---CCGAUCGGAGACGUCGAGGC---GGUGGAAAUAGCGGAAACACAUCUGCAGCUGCAGCGGCUGCUGCAGCAGCAGUUGCCCACGCA .((.(((((.((....---)).))))))).(((....))---)((((.......(....).((.((((.((((((((....)))))))).)))).)).))))... ( -46.70) >DroYak_CAF1 335 99 + 1 GCUGUUGACGGAGAAU---CUGAUCGAAAACGUAGAGGU---GGGGGAAAUAGUGCAAACACAUCCGCAGCCGCAGCAGCAGCUGCAGCCGCUGUCGCACAUGCA ((((((((((((...(---((.(((...........)))---.)))......(((....))).)))((.((.(((((....))))).)).)).)))).))).)). ( -31.20) >DroMoj_CAF1 2029 105 + 1 GCAGCUGAAGGCGAUACACCGGAUCGAAGGAGGCGCGGCGGUGCUGGCAAUAAUGCAAAUACUUCGGCUGCAGCUGCAGCGGCUGCCGCGGCCGUGGCACACGCA (((((((((((((...(.((((.((....))..).))).).)))..(((....))).....)))))))))).......(((..((((((....))))))..))). ( -46.80) >DroPer_CAF1 2728 99 + 1 ACUGCUGAUGGGGAAA---CCGAUCGGAGACGUCGAGGC---GGUGGAAAUAGCGGAAACACAUCUGCAGCUGCAGCGGCUGCUGCAGCAGCAGUUGCCCACGCA .((.(((((.((....---)).))))))).(((....))---)((((.......(....).((.((((.((((((((....)))))))).)))).)).))))... ( -46.70) >consensus GCUGCUGAAGGAGAAA___CCGAUCGAAGACGUCGAGGC___GGUGGAAAUAGUGCAAACACAUCUGCAGCAGCAGCAGCAGCUGCAGCAGCUGUUGCACACGCA (((((.((((.((......))...((....)).............................)))).))))).((.((((((((((...))))))))))....)). (-22.68 = -22.35 + -0.33)

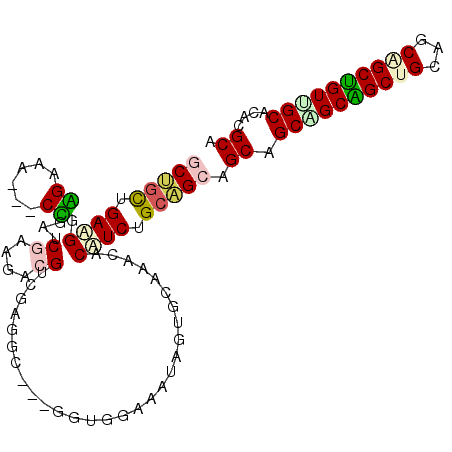
| Location | 802,520 – 802,619 |
|---|---|
| Length | 99 |
| Sequences | 6 |
| Columns | 105 |
| Reading direction | reverse |
| Mean pairwise identity | 74.98 |
| Mean single sequence MFE | -38.61 |
| Consensus MFE | -19.85 |
| Energy contribution | -20.72 |
| Covariance contribution | 0.86 |
| Combinations/Pair | 1.32 |
| Mean z-score | -1.71 |
| Structure conservation index | 0.51 |
| SVM decision value | 0.20 |
| SVM RNA-class probability | 0.631361 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 802520 99 - 20766785 UGCAUGUGCGACAGCGGCGGCAGCUGCCGCUGCGGCUGCGGAUGUAUUUGCACUAUUUCCACC---GGCUCUACGUUUUCGAUCAG---AUUCUCCCUCAACGGC ((((.(((((.(.(((((.(((((....))))).))))).).))))).)))).........((---(...........((.....)---)...........))). ( -32.55) >DroVir_CAF1 1938 102 - 1 GGCGUGUGCAACAGCCGCAGCAGCUGCUGCAGCUGCAGCAGAAGUGUUGGCAUUAUUGCCAGC---GGCGCGCCUUCUUCGAUCCGGAGUAUCGCCUUCAGGUGC (((((((((..((((.(((((....))))).))))..)).....((((((((....)))))))---))))))))..(((((...)))))...((((....)))). ( -48.60) >DroPse_CAF1 2616 99 - 1 UGCGUGGGCAACUGCUGCUGCAGCAGCCGCUGCAGCUGCAGAUGUGUUUCCGCUAUUUCCACC---GCCUCGACGUCUCCGAUCGG---UUUCCCCAUCAGCAGU ((((((((((.((((.((((((((....)))))))).)))).)))....)))).......(((---(..(((.......))).)))---)..........))).. ( -38.10) >DroYak_CAF1 335 99 - 1 UGCAUGUGCGACAGCGGCUGCAGCUGCUGCUGCGGCUGCGGAUGUGUUUGCACUAUUUCCCCC---ACCUCUACGUUUUCGAUCAG---AUUCUCCGUCAACAGC .((.(((..(((.(((((((((((....)))))))))))(((.(((....)))..........---.......((....)).....---....)))))).))))) ( -35.00) >DroMoj_CAF1 2029 105 - 1 UGCGUGUGCCACGGCCGCGGCAGCCGCUGCAGCUGCAGCCGAAGUAUUUGCAUUAUUGCCAGCACCGCCGCGCCUCCUUCGAUCCGGUGUAUCGCCUUCAGCUGC .(((..((((.(....).))))..))).(((((((.((.(((.......(((....)))..((((((...((.......))...)))))).))).)).))))))) ( -39.30) >DroPer_CAF1 2728 99 - 1 UGCGUGGGCAACUGCUGCUGCAGCAGCCGCUGCAGCUGCAGAUGUGUUUCCGCUAUUUCCACC---GCCUCGACGUCUCCGAUCGG---UUUCCCCAUCAGCAGU ((((((((((.((((.((((((((....)))))))).)))).)))....)))).......(((---(..(((.......))).)))---)..........))).. ( -38.10) >consensus UGCGUGUGCAACAGCCGCUGCAGCUGCCGCUGCAGCUGCAGAUGUGUUUGCACUAUUUCCACC___GCCUCGACGUCUUCGAUCAG___UUUCCCCAUCAGCAGC .(((.(((((.(.((.((((((((....)))))))).)).).))))).)))...................................................... (-19.85 = -20.72 + 0.86)

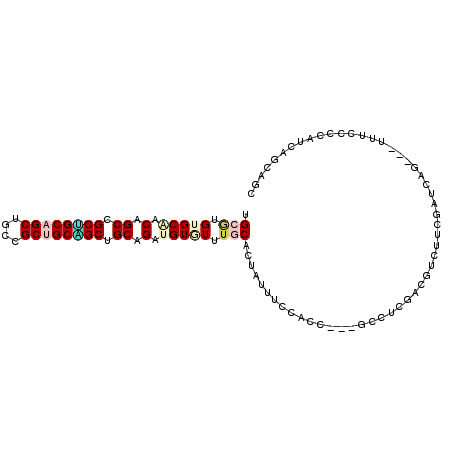
| Location | 802,554 – 802,650 |
|---|---|
| Length | 96 |
| Sequences | 6 |
| Columns | 99 |
| Reading direction | forward |
| Mean pairwise identity | 76.70 |
| Mean single sequence MFE | -47.25 |
| Consensus MFE | -27.38 |
| Energy contribution | -26.60 |
| Covariance contribution | -0.77 |
| Combinations/Pair | 1.39 |
| Mean z-score | -3.77 |
| Structure conservation index | 0.58 |
| SVM decision value | 2.77 |
| SVM RNA-class probability | 0.996931 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 802554 96 + 20766785 CC---GGUGGAAAUAGUGCAAAUACAUCCGCAGCCGCAGCGGCAGCUGCCGCCGCUGUCGCACAUGCAGCAUGUACUGCAGCUGCUGUCGCUAGCACUG .(---((((....(((((((............((.((((((((.......)))))))).))....(((((.((.....))))))))).))))).))))) ( -38.20) >DroVir_CAF1 1975 96 + 1 CC---GCUGGCAAUAAUGCCAACACUUCUGCUGCAGCUGCAGCAGCUGCUGCGGCUGUUGCACACGCCGCAAGCACUGCGGCCGCAGCAGCAGCUACGG ((---(.(((((....)))))........((((((((((((((....)))))))))(((((....((((((.....)))))).))))).)))))..))) ( -50.70) >DroEre_CAF1 16791 96 + 1 GC---GGUGGAAAUAGUGCAAACACAUCAGCAGCCGCAGCGGCAGCUGCUGCCGCUGUCGCACAUGCAGCAUGUACUGCAGCUGCUGCCGCCACCACAG (.---(((((.....(((....)))..((((((((((((((((((...))))))))).))....(((((......))))))))))))...))))).).. ( -47.40) >DroYak_CAF1 369 96 + 1 GU---GGGGGAAAUAGUGCAAACACAUCCGCAGCCGCAGCAGCAGCUGCAGCCGCUGUCGCACAUGCAGCGUGUACAGCGGCUGCUGCCGCCACCACUG ((---((((......(((....)))....(((((.(((((....)))))(((((((((.((((.......))))))))))))))))))..)).)))).. ( -48.50) >DroMoj_CAF1 2066 99 + 1 GCGGUGCUGGCAAUAAUGCAAAUACUUCGGCUGCAGCUGCAGCGGCUGCCGCGGCCGUGGCACACGCAGCCAGCACAGCGGCUGCAGCAGCUGCCACUG (.((((...(((....)))...)))).)(((.((.((((((((.((((..(((((.(((.....))).))).)).)))).)))))))).)).))).... ( -50.00) >DroPer_CAF1 2762 93 + 1 GC---GGUGGAAAUAGCGGAAACACAUCUGCAGCUGCAGCGGCUGCUGCAGCAGCAGUUGCCCACGCAGCUAACACGGCAGCUGCUGCAGC---CACAG ..---.(((......(((((......))))).(((((((((((((((((....))((((((....)))))).....)))))))))))))))---))).. ( -48.70) >consensus GC___GGUGGAAAUAGUGCAAACACAUCCGCAGCCGCAGCAGCAGCUGCAGCCGCUGUCGCACACGCAGCAAGCACUGCAGCUGCUGCAGCCACCACUG .............................(((((.((.((((((((.......)))))))).......(((.....))).)).)))))........... (-27.38 = -26.60 + -0.77)

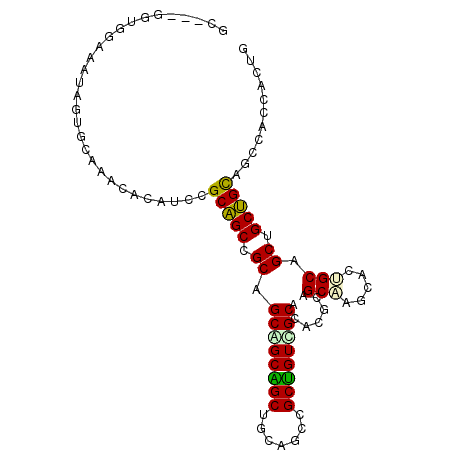
| Location | 802,554 – 802,650 |
|---|---|
| Length | 96 |
| Sequences | 6 |
| Columns | 99 |
| Reading direction | reverse |
| Mean pairwise identity | 76.70 |
| Mean single sequence MFE | -51.08 |
| Consensus MFE | -28.58 |
| Energy contribution | -30.05 |
| Covariance contribution | 1.47 |
| Combinations/Pair | 1.38 |
| Mean z-score | -3.95 |
| Structure conservation index | 0.56 |
| SVM decision value | 2.85 |
| SVM RNA-class probability | 0.997378 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 802554 96 - 20766785 CAGUGCUAGCGACAGCAGCUGCAGUACAUGCUGCAUGUGCGACAGCGGCGGCAGCUGCCGCUGCGGCUGCGGAUGUAUUUGCACUAUUUCCACC---GG .(((((..(((.(.((((((((.(((((((...)))))))...((((((((...)))))))))))))))).).)))....))))).........---.. ( -44.80) >DroVir_CAF1 1975 96 - 1 CCGUAGCUGCUGCUGCGGCCGCAGUGCUUGCGGCGUGUGCAACAGCCGCAGCAGCUGCUGCAGCUGCAGCAGAAGUGUUGGCAUUAUUGCCAGC---GG ((...(((.((((((((((.((((((((((((((.((.....))))))))..))).))))).)))))))))).)))(((((((....)))))))---)) ( -58.30) >DroEre_CAF1 16791 96 - 1 CUGUGGUGGCGGCAGCAGCUGCAGUACAUGCUGCAUGUGCGACAGCGGCAGCAGCUGCCGCUGCGGCUGCUGAUGUGUUUGCACUAUUUCCACC---GC ..(((((((...((((((((((.(((((((...)))))))...((((((((...))))))))))))))))))..(((....))).....)))))---)) ( -54.10) >DroYak_CAF1 369 96 - 1 CAGUGGUGGCGGCAGCAGCCGCUGUACACGCUGCAUGUGCGACAGCGGCUGCAGCUGCUGCUGCGGCUGCGGAUGUGUUUGCACUAUUUCCCCC---AC ((((.(..(((((((((((((((((.(.(((.....))).))))))))))...))))))))..).)))).(((.(((....)))....)))...---.. ( -52.10) >DroMoj_CAF1 2066 99 - 1 CAGUGGCAGCUGCUGCAGCCGCUGUGCUGGCUGCGUGUGCCACGGCCGCGGCAGCCGCUGCAGCUGCAGCCGAAGUAUUUGCAUUAUUGCCAGCACCGC .((((((.((....)).))))))((((((((((((.((((..((((.(((((.((....)).))))).))))..)))).)))).....))))))))... ( -52.10) >DroPer_CAF1 2762 93 - 1 CUGUG---GCUGCAGCAGCUGCCGUGUUAGCUGCGUGGGCAACUGCUGCUGCAGCAGCCGCUGCAGCUGCAGAUGUGUUUCCGCUAUUUCCACC---GC (((..---((((((((.(((((..(((.(((...(..(....)..).))))))))))).))))))))..)))..(((....)))..........---.. ( -45.10) >consensus CAGUGGUAGCGGCAGCAGCCGCAGUACAUGCUGCAUGUGCGACAGCGGCAGCAGCUGCCGCUGCGGCUGCAGAUGUGUUUGCACUAUUUCCACC___GC ..(((((.((....)).)))))..........(((..((((.((((.((((((((....)))))))).)))).))))..)))................. (-28.58 = -30.05 + 1.47)


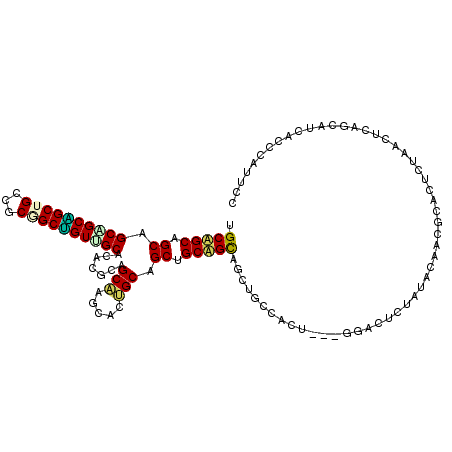
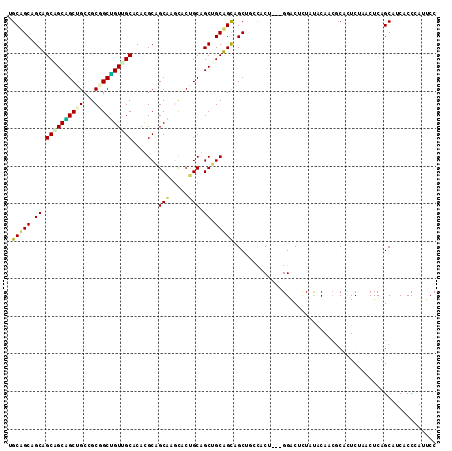
| Location | 802,579 – 802,690 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 77.16 |
| Mean single sequence MFE | -43.61 |
| Consensus MFE | -27.44 |
| Energy contribution | -26.72 |
| Covariance contribution | -0.72 |
| Combinations/Pair | 1.45 |
| Mean z-score | -3.05 |
| Structure conservation index | 0.63 |
| SVM decision value | 2.32 |
| SVM RNA-class probability | 0.992342 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 802579 111 + 20766785 CGCAGCCGCAGCGGCAGCUGCCGCCGCUGUCGCACAUGCAGCAUGUACUGCAGCUGCUGUCGCUAGCACU---GGGCUCUAUGCAUCUCAUUCCAACUCAGCUUCGCCUAUUCC .(((((.(((((((((((.......))))))))....(((((.((.....)))))))))).))).))..(---((((.....((................))...))))).... ( -37.79) >DroVir_CAF1 2000 111 + 1 UGCUGCAGCUGCAGCAGCUGCUGCGGCUGUUGCACACGCCGCAAGCACUGCGGCCGCAGCAGCAGCUACG---GGCCUUUACGCAACGCACUCUAACUCAGCAUCACCCAUUCC (((((.(((((.(((.(((((((((((((((((...........)))..)))))))))))))).))).))---)((......))............)))))))........... ( -46.00) >DroPse_CAF1 2675 108 + 1 UGCAGCUGCAGCGGCUGCUGCAGCAGCAGUUGCCCACGCAGCUAACACGGCAGCUGCUGCAGC---CACA---GGACUCUAUACAUCGCAUUCAAACUCAGCAUCGCCCAUUCC .((.(((((((((((((((((....))((((((....)))))).....)))))))))))))))---....---..............((...........))...))....... ( -41.50) >DroGri_CAF1 2512 114 + 1 GGCAGCAGCUGCAGCGGCAGCCGCGGCUGUCGCCCAUGCUGCAAGCACUGCGGCAGCAGCAGCUGCUACUCAGGGACUCUAUGCAACGCACUCUAACUCAGCAUCACCGAUUCC ((((((.(((((.((((((((....))))))))...(((((((.....)))))))))))).))))))......(((.((.((((................))))....)).))) ( -49.69) >DroMoj_CAF1 2094 111 + 1 GGCUGCAGCUGCAGCGGCUGCCGCGGCCGUGGCACACGCAGCCAGCACAGCGGCUGCAGCAGCUGCCACU---GGACUAUACACAAACCACUCUAACUCAGCAUCACCAAUUCC (((.((.((((((((.((((..(((((.(((.....))).))).)).)))).)))))))).)).)))...---......................................... ( -45.20) >DroPer_CAF1 2787 108 + 1 UGCAGCUGCAGCGGCUGCUGCAGCAGCAGUUGCCCACGCAGCUAACACGGCAGCUGCUGCAGC---CACA---GGACUCUAUACAUCGCAUUCAAACUCAGCAUCGCCCAUUCC .((.(((((((((((((((((....))((((((....)))))).....)))))))))))))))---....---..............((...........))...))....... ( -41.50) >consensus UGCAGCAGCAGCAGCAGCUGCCGCGGCUGUUGCACACGCAGCAAGCACUGCAGCUGCAGCAGCUGCCACU___GGACUCUAUACAACGCACUCUAACUCAGCAUCACCCAUUCC .(((((.((.((((((((((...)))))))))).......(((.....))).)).)))))...................................................... (-27.44 = -26.72 + -0.72)
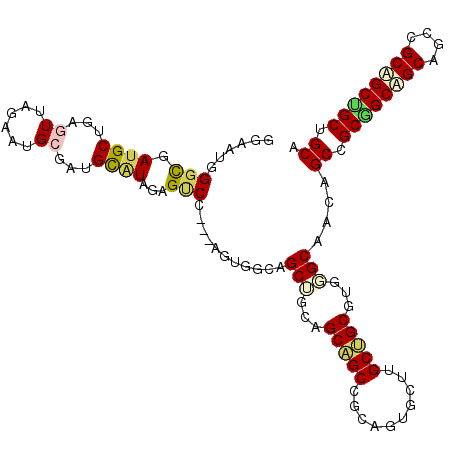
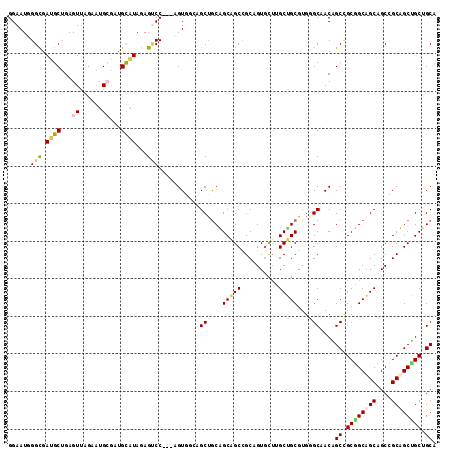
| Location | 802,579 – 802,690 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 77.16 |
| Mean single sequence MFE | -51.63 |
| Consensus MFE | -29.19 |
| Energy contribution | -29.13 |
| Covariance contribution | -0.05 |
| Combinations/Pair | 1.33 |
| Mean z-score | -2.62 |
| Structure conservation index | 0.57 |
| SVM decision value | 1.91 |
| SVM RNA-class probability | 0.982247 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 802579 111 - 20766785 GGAAUAGGCGAAGCUGAGUUGGAAUGAGAUGCAUAGAGCCC---AGUGCUAGCGACAGCAGCUGCAGUACAUGCUGCAUGUGCGACAGCGGCGGCAGCUGCCGCUGCGGCUGCG .......((...((((.(((...(((.....)))..))).)---)))....))....((((((((.(((((((...)))))))...((((((((...)))))))))))))))). ( -45.60) >DroVir_CAF1 2000 111 - 1 GGAAUGGGUGAUGCUGAGUUAGAGUGCGUUGCGUAAAGGCC---CGUAGCUGCUGCUGCGGCCGCAGUGCUUGCGGCGUGUGCAACAGCCGCAGCAGCUGCUGCAGCUGCAGCA ...((((((.((((...((......))...))))....)))---))).(((((.((((((((.((..((((.(((((.((.....))))))))))))).)))))))).))))). ( -57.90) >DroPse_CAF1 2675 108 - 1 GGAAUGGGCGAUGCUGAGUUUGAAUGCGAUGUAUAGAGUCC---UGUG---GCUGCAGCAGCUGCCGUGUUAGCUGCGUGGGCAACUGCUGCUGCAGCAGCCGCUGCAGCUGCA .....((((.((((...((......))...))))...))))---.(..---((((((((.(((((..(((.(((...(..(....)..).))))))))))).))))))))..). ( -50.60) >DroGri_CAF1 2512 114 - 1 GGAAUCGGUGAUGCUGAGUUAGAGUGCGUUGCAUAGAGUCCCUGAGUAGCAGCUGCUGCUGCCGCAGUGCUUGCAGCAUGGGCGACAGCCGCGGCUGCCGCUGCAGCUGCUGCC (((.((.(..((((...........))))..)...)).)))....(((((((((((.((.(((((.((((.....)))).(((....))))))))....)).))))))))))). ( -56.20) >DroMoj_CAF1 2094 111 - 1 GGAAUUGGUGAUGCUGAGUUAGAGUGGUUUGUGUAUAGUCC---AGUGGCAGCUGCUGCAGCCGCUGUGCUGGCUGCGUGUGCCACGGCCGCGGCAGCCGCUGCAGCUGCAGCC ...(((((..((((.((.(......).))...))))...))---)))(((.((.((((((((.(((((((.(((((.(.....).))))))).))))).)))))))).)).))) ( -48.90) >DroPer_CAF1 2787 108 - 1 GGAAUGGGCGAUGCUGAGUUUGAAUGCGAUGUAUAGAGUCC---UGUG---GCUGCAGCAGCUGCCGUGUUAGCUGCGUGGGCAACUGCUGCUGCAGCAGCCGCUGCAGCUGCA .....((((.((((...((......))...))))...))))---.(..---((((((((.(((((..(((.(((...(..(....)..).))))))))))).))))))))..). ( -50.60) >consensus GGAAUGGGCGAUGCUGAGUUAGAAUGCGAUGCAUAGAGUCC___AGUGGCAGCUGCAGCAGCCGCAGUGCUUGCUGCGUGGGCAACAGCCGCGGCAGCAGCCGCAGCUGCUGCA ......(((.((((...((......))...))))...)))...........(((...(((((..........)))))...)))....((.((((((((....)))))))).)). (-29.19 = -29.13 + -0.05)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:23:09 2006