
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 13,055,056 – 13,055,176 |
| Length | 120 |
| Max. P | 0.779563 |

| Location | 13,055,056 – 13,055,176 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 82.39 |
| Mean single sequence MFE | -47.28 |
| Consensus MFE | -34.54 |
| Energy contribution | -34.52 |
| Covariance contribution | -0.02 |
| Combinations/Pair | 1.27 |
| Mean z-score | -2.05 |
| Structure conservation index | 0.73 |
| SVM decision value | 0.55 |
| SVM RNA-class probability | 0.779563 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
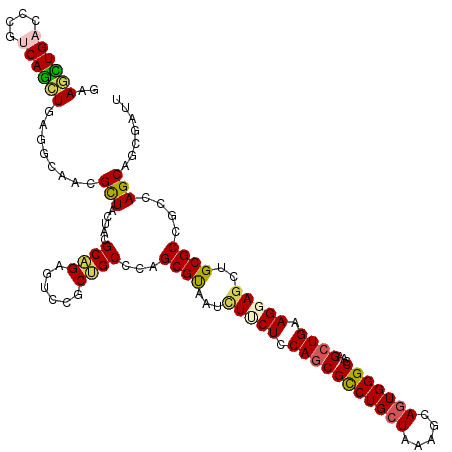
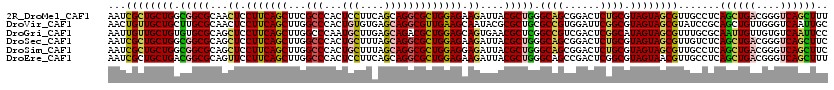
>2R_DroMel_CAF1 13055056 120 + 20766785 AAAGCUGACCCGUCAGCUGAGGCAACGCUACUACGCAGAGUCCGCUGCCCAGCGUAAUCUUCUCCAGCGCCUGCUGAAGGAGUGGGCGAAGCUGAAGGAGUUGCGCCGCCAGCAGCGAUU ..((((((....))))))..(....)((......))......((((((.(.((((((.(((((.(((((((..((.....))..)))...)))).))))))))))).)...))))))... ( -50.00) >DroVir_CAF1 5451 120 + 1 GCAAUUGACCCAACAGCUGCGGAUACGCUACUACGCCGAAUCCACGGCGCAGCGCGUAUUGCUUCAACGCCUGCUCACACAGUGGGCCAAGCUGAAGGAGUUGCGCAAGCAGCAACAGUU ..(((((..((..((((((((.((((((..((.(((((......))))).)).)))))))))......(((..((.....))..)))..)))))..)).(((((....)))))..))))) ( -50.10) >DroGri_CAF1 5014 120 + 1 GGAAUUGACACAACAAUUGCGCAAACGCUACUAUGCCGAGUCGACGGCCGAGCGUUCACUGCUCCAGCGUCUGCUCAAGCAUUGGGCCAAGCUGAAGGAGCUGCGCACACAGCAACAAUU ..(((((......)))))(((((((((((.....((((......))))..))))))....((((((((....(((((.....)))))...)))...)))))))))).............. ( -38.90) >DroSec_CAF1 3804 120 + 1 GAAGCUGACCCGUCAGCUGAGACAACGCUACUACGCAGAGUCCGCUGCCCAGCGUAAUCUUCUCCAGCGCCUGCUAAAGCAGUGGGCCAAGCUGAAGGAGCUGCGCCGCCAGCAGCGAUU ..((((((....))))))..(((...((......))...)))((((((.(.(((((..(((((.(((((((..((.....))..)))...)))).))))).))))).)...))))))... ( -47.70) >DroSim_CAF1 3756 120 + 1 GAAGCUGACCCGUCAGCUGAGGCAACGCUACUACGCAGAGUCCGCUGCCCAGCGUAAUCUCCUCCAGCGCCUGCUAAAGCAGUGGGCCAAGCUGAAGGAGCUGCGCCGCCAGCAGCGAUU ..((((((....))))))..(....)((......))......((((((.(.(((((..(((((.(((((((..((.....))..)))...)))).))))).))))).)...))))))... ( -50.20) >DroEre_CAF1 3746 120 + 1 AAAGCUGACCCGUCAGCUGAGGCAACGUUACUACGCCGAGUCGGCUGCCCAGCGUAAUCUUCUCCAGCGCCUGCUGAAGGAGUGGGCCAAGCUGAAGGAACUGCGCCGUCAGCAGCGAUU ...((((((......((((.((((..((....))((((...))))))))))))(((...((((.(((((((..((.....))..)))...)))).))))..)))...))))))....... ( -46.80) >consensus GAAGCUGACCCGUCAGCUGAGGCAACGCUACUACGCAGAGUCCGCUGCCCAGCGUAAUCUUCUCCAGCGCCUGCUAAAGCAGUGGGCCAAGCUGAAGGAGCUGCGCCGCCAGCAGCGAUU ..((((((....))))))........(((.....((((......))))...((((...(((((.(((((((((((.....)))))))...)))).)))))..))))....)))....... (-34.54 = -34.52 + -0.02)

| Location | 13,055,056 – 13,055,176 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 82.39 |
| Mean single sequence MFE | -50.03 |
| Consensus MFE | -36.35 |
| Energy contribution | -35.97 |
| Covariance contribution | -0.38 |
| Combinations/Pair | 1.32 |
| Mean z-score | -1.69 |
| Structure conservation index | 0.73 |
| SVM decision value | 0.27 |
| SVM RNA-class probability | 0.660979 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
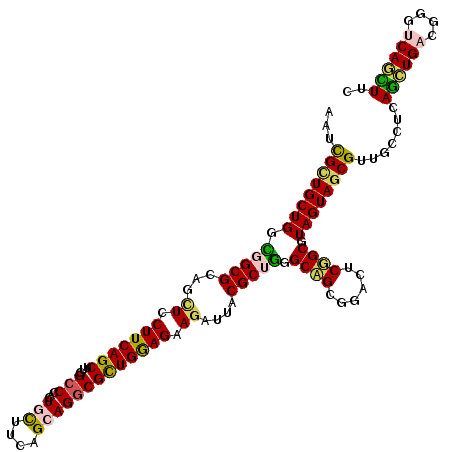
>2R_DroMel_CAF1 13055056 120 - 20766785 AAUCGCUGCUGGCGGCGCAACUCCUUCAGCUUCGCCCACUCCUUCAGCAGGCGCUGGAGAAGAUUACGCUGGGCAGCGGACUCUGCGUAGUAGCGUUGCCUCAGCUGACGGGUCAGCUUU ...(((((....)))))..........((((..((((.((.(((((((....))))))).)).....(((((((((((.(((......)))..))))))).))))....)))).)))).. ( -48.40) >DroVir_CAF1 5451 120 - 1 AACUGUUGCUGCUUGCGCAACUCCUUCAGCUUGGCCCACUGUGUGAGCAGGCGUUGAAGCAAUACGCGCUGCGCCGUGGAUUCGGCGUAGUAGCGUAUCCGCAGCUGUUGGGUCAAUUGC ....(((((.......))))).....(((.(((((((((((((.((....((......))..((((((((((((((......))))))))).))))))))))))....))))))))))). ( -48.60) >DroGri_CAF1 5014 120 - 1 AAUUGUUGCUGUGUGCGCAGCUCCUUCAGCUUGGCCCAAUGCUUGAGCAGACGCUGGAGCAGUGAACGCUCGGCCGUCGACUCGGCAUAGUAGCGUUUGCGCAAUUGUUGUGUCAAUUCC .......(((((....))))).........(((((.((((..(((.((((((((((((((.(....))))).((((......))))....)))))))))).)))..)))).))))).... ( -46.50) >DroSec_CAF1 3804 120 - 1 AAUCGCUGCUGGCGGCGCAGCUCCUUCAGCUUGGCCCACUGCUUUAGCAGGCGCUGGAGAAGAUUACGCUGGGCAGCGGACUCUGCGUAGUAGCGUUGUCUCAGCUGACGGGUCAGCUUC ...(((((((..(((((...((.(((((((...(((...(((....))))))))))))).))....))))))))))))(((..(((......)))..)))..((((((....)))))).. ( -50.60) >DroSim_CAF1 3756 120 - 1 AAUCGCUGCUGGCGGCGCAGCUCCUUCAGCUUGGCCCACUGCUUUAGCAGGCGCUGGAGGAGAUUACGCUGGGCAGCGGACUCUGCGUAGUAGCGUUGCCUCAGCUGACGGGUCAGCUUC ..........((((((((..((((((((((...(((...(((....))))))))))))))))(((((((.((((....).))).))))))).))))))))..((((((....)))))).. ( -56.80) >DroEre_CAF1 3746 120 - 1 AAUCGCUGCUGACGGCGCAGUUCCUUCAGCUUGGCCCACUCCUUCAGCAGGCGCUGGAGAAGAUUACGCUGGGCAGCCGACUCGGCGUAGUAACGUUGCCUCAGCUGACGGGUCAGCUUU .......(((((((((...)))((.((((((.(((...((.(((((((....))))))).))((((((((((((....).)))))))))))......)))..)))))).))))))))... ( -49.30) >consensus AAUCGCUGCUGGCGGCGCAGCUCCUUCAGCUUGGCCCACUGCUUCAGCAGGCGCUGGAGAAGAUUACGCUGGGCAGCGGACUCGGCGUAGUAGCGUUGCCUCAGCUGACGGGUCAGCUUC ...((((((((.(((((...((.(((((((...(((...(((....))))))))))))).))....))))).((((......)))).)))))))).......((((((....)))))).. (-36.35 = -35.97 + -0.38)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:08:21 2006