
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 13,041,695 – 13,041,850 |
| Length | 155 |
| Max. P | 0.581042 |

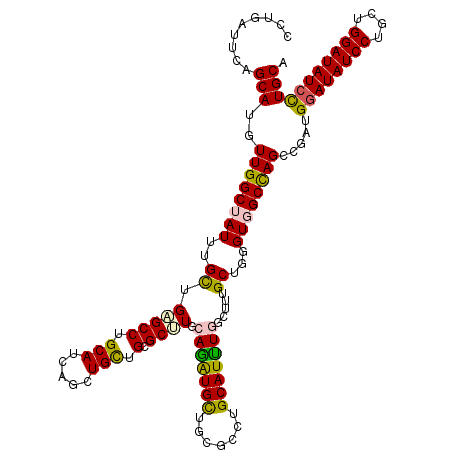
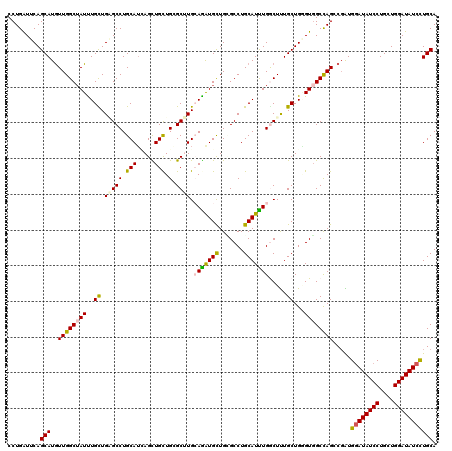
| Location | 13,041,695 – 13,041,815 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 88.44 |
| Mean single sequence MFE | -51.32 |
| Consensus MFE | -37.21 |
| Energy contribution | -36.77 |
| Covariance contribution | -0.44 |
| Combinations/Pair | 1.25 |
| Mean z-score | -3.24 |
| Structure conservation index | 0.73 |
| SVM decision value | 0.09 |
| SVM RNA-class probability | 0.576737 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 13041695 120 + 20766785 CCUGGUUCAGCAUGUUGGCUAUUUGCUGAGCCUGCAUGAGUUGUUGGGCUUGGAGGUGCUGCGCCUGCAUUUGGCUUUGCUGGGUGGCCAGGCGGUGGAUAUCCUGCUGGAUAUCCUGCA ((((((((((((....(((((..(((.(((((((((.....)).)))))))..(((((...))))))))..))))).))))))...))))))..((((((((((....)))))))).)). ( -53.40) >DroVir_CAF1 16356 120 + 1 CCUGAUUCAGCAUGUUGGCUAUUUGUUGUGCCUGCAUCAGCUGCUGCGCUUGCAGAUGCUGUGCCUGCAUUUGGCUUUGCUGCGUGGCCAGCCGAUGGAUAUCCUGUUGGAUAUCCUGCA .........(((.(((((((....((((((....)).)))).((..(((..(((((.((((((....)))..)))))))).)))..)).)))))))((((((((....))))))))))). ( -49.30) >DroGri_CAF1 24001 120 + 1 CCUGAUUCAGCAUGUUGGCGAUUUGCUGAGCCUGCAUUAGUUGCUGGGCUUGCAAGUGCUGCGCCUGCAUUUGACUUUGCUGGGUGGCCAGGCGGUGGAUAUCCUGCUGGAUAUCCUGCA (((((((((((((((.((((((((((.(((((((((.....))).))))))))))))....)))).)))........))))))))...))))..((((((((((....)))))))).)). ( -50.00) >DroWil_CAF1 15017 120 + 1 CCUGAUUCAGCAUGUUGGCUAUCUGUUGGGCCUGCAUCAGCUGCUGGGCCUGAAGAUGUUGGGCCUGCAUUUGACUUUGCUGAGUUGCUAGCCUAUGGAUAUCCUGCUGGAUAUCUUGCA ...((((((((((((.((((........)))).)))((((.(((.(((((..(.....)..)))))))).))))...)))))))))....((....((((((((....)))))))).)). ( -48.40) >DroMoj_CAF1 28878 120 + 1 CCUGAUUCAGCAUGUUGGCUAUUUGCUGUGCCUGCAUCAGCUGCUGCGCUUGCAAAUGCUGCGCCUGCAUUUGGCUUUGCUGGGUGGCCAGCCGAUGAAUAUCCUGCUGGAUAUCCUGCA .........(((.(((((((((((((.((((..(((.....))).))))..))))))((..(.((.(((........))).)))..)).)))))))(.((((((....)))))).)))). ( -43.70) >DroPer_CAF1 15896 120 + 1 CCUGGUUCAGCAUAUUGGCUAUUUGCUGGGCCUGCAGGAGCUGCUGCGCCUGCAGGUGCUGGGCCUGCAUCUGGCUCUGCUGCGUGGCCAGGCGGUGGAUAUCCUGCUGGAUAUCCUGCA ...((((((((...(..((.....))..)(((((((((.((....)).))))))))))))))))).((((((((((.((...)).)))))))....((((((((....))))))))))). ( -63.10) >consensus CCUGAUUCAGCAUGUUGGCUAUUUGCUGAGCCUGCAUCAGCUGCUGCGCUUGCAGAUGCUGCGCCUGCAUUUGGCUUUGCUGGGUGGCCAGCCGAUGGAUAUCCUGCUGGAUAUCCUGCA .........(((..((((((((..((.(((((.(((.....))).).)))).(((((((.......))))))).....))...)))))))).....((((((((....))))))))))). (-37.21 = -36.77 + -0.44)

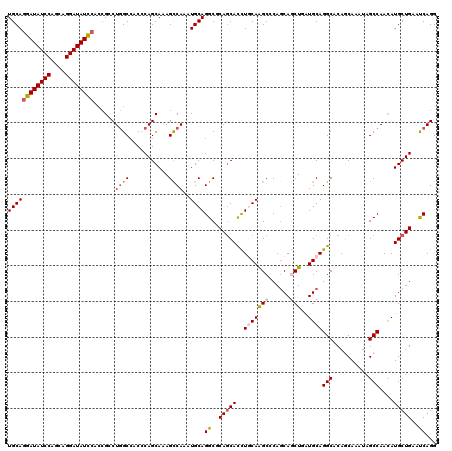
| Location | 13,041,695 – 13,041,815 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 88.44 |
| Mean single sequence MFE | -41.57 |
| Consensus MFE | -30.72 |
| Energy contribution | -31.08 |
| Covariance contribution | 0.36 |
| Combinations/Pair | 1.12 |
| Mean z-score | -3.05 |
| Structure conservation index | 0.74 |
| SVM decision value | 0.09 |
| SVM RNA-class probability | 0.581042 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 13041695 120 - 20766785 UGCAGGAUAUCCAGCAGGAUAUCCACCGCCUGGCCACCCAGCAAAGCCAAAUGCAGGCGCAGCACCUCCAAGCCCAACAACUCAUGCAGGCUCAGCAAAUAGCCAACAUGCUGAACCAGG ((((((((((((....))))))))..(((((((....)).(((........)))))))).........................))))((.((((((...........)))))).))... ( -36.50) >DroVir_CAF1 16356 120 - 1 UGCAGGAUAUCCAACAGGAUAUCCAUCGGCUGGCCACGCAGCAAAGCCAAAUGCAGGCACAGCAUCUGCAAGCGCAGCAGCUGAUGCAGGCACAACAAAUAGCCAACAUGCUGAAUCAGG .(((((((((((....))))))))...(((((((...........)))...(((..(((((((..((((....))))..)))).)))..)))........))))....)))......... ( -44.50) >DroGri_CAF1 24001 120 - 1 UGCAGGAUAUCCAGCAGGAUAUCCACCGCCUGGCCACCCAGCAAAGUCAAAUGCAGGCGCAGCACUUGCAAGCCCAGCAACUAAUGCAGGCUCAGCAAAUCGCCAACAUGCUGAAUCAGG (((.((((((((....))))))))..(((((((....)).(((........))))))))..)))((((...(((..(((.....))).)))((((((...........))))))..)))) ( -39.10) >DroWil_CAF1 15017 120 - 1 UGCAAGAUAUCCAGCAGGAUAUCCAUAGGCUAGCAACUCAGCAAAGUCAAAUGCAGGCCCAACAUCUUCAGGCCCAGCAGCUGAUGCAGGCCCAACAGAUAGCCAACAUGCUGAAUCAGG .(((.(((((((....)))))))....(((((........((...((((..(((.((((...........))))..)))..))))....))........)))))....)))......... ( -35.09) >DroMoj_CAF1 28878 120 - 1 UGCAGGAUAUCCAGCAGGAUAUUCAUCGGCUGGCCACCCAGCAAAGCCAAAUGCAGGCGCAGCAUUUGCAAGCGCAGCAGCUGAUGCAGGCACAGCAAAUAGCCAACAUGCUGAAUCAGG .((.((((((((....))))))))....(((((....)))))...))....(((..(((((((..((((....))))..)))).)))..)))(((((...........)))))....... ( -41.30) >DroPer_CAF1 15896 120 - 1 UGCAGGAUAUCCAGCAGGAUAUCCACCGCCUGGCCACGCAGCAGAGCCAGAUGCAGGCCCAGCACCUGCAGGCGCAGCAGCUCCUGCAGGCCCAGCAAAUAGCCAAUAUGCUGAACCAGG ((((((((((((....)))))))).....((((((........).))))).))))((..(((((((((((((.((....)).))))))))....((.....)).....)))))..))... ( -52.90) >consensus UGCAGGAUAUCCAGCAGGAUAUCCACCGCCUGGCCACCCAGCAAAGCCAAAUGCAGGCGCAGCACCUGCAAGCCCAGCAGCUGAUGCAGGCACAGCAAAUAGCCAACAUGCUGAAUCAGG ((((((((((((....))))))))......((((...........))))..))))((..(((((..(((((((......)))..))))(((..........)))....)))))..))... (-30.72 = -31.08 + 0.36)


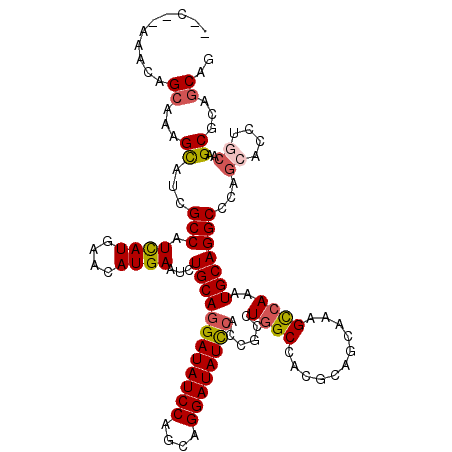
| Location | 13,041,735 – 13,041,850 |
|---|---|
| Length | 115 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | reverse |
| Mean pairwise identity | 86.70 |
| Mean single sequence MFE | -36.32 |
| Consensus MFE | -27.00 |
| Energy contribution | -27.37 |
| Covariance contribution | 0.36 |
| Combinations/Pair | 1.14 |
| Mean z-score | -2.60 |
| Structure conservation index | 0.74 |
| SVM decision value | 0.06 |
| SVM RNA-class probability | 0.564687 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 13041735 115 - 20766785 --C--AAGCAGCAAAGUAUCGCCAUUAUGAACAUGAAUCUGCAGGAUAUCCAGCAGGAUAUCCACCGCCUGGCCACCCAGCAAAGCCAAAUGCAGGCGCAGCACCUCCAAGCCCAACAA --.--..((.((........))...........((..((((((((((((((....))))))))...((.(((....))))).........))))))..))))................. ( -29.20) >DroVir_CAF1 16396 119 - 1 AACGAAAACAGCAAAGCAUCGCCAUUAUGAACAUGAAUCUGCAGGAUAUCCAACAGGAUAUCCAUCGGCUGGCCACGCAGCAAAGCCAAAUGCAGGCACAGCAUCUGCAAGCGCAGCAG ..........((...((...(((.((((....))))...((((((((((((....))))))))...((((.((......))..))))...)))))))...((....))..))...)).. ( -36.20) >DroPse_CAF1 17410 115 - 1 --C--GUGCAGCAAAGCAUCGCCAUCAUGAACAUGAAUCUGCAGGAUAUCCAGCAGGAUAUCCACCGCCUGGCCACGCAGCAGAGCCAGAUGCAGGCCCAGCACCUGCAGGCGCAGCAG --.--.(((.((...(((((....((((....)))).(((((.((((((((....))))))))...((........)).)))))....)))))..(((..((....)).))))).))). ( -42.90) >DroWil_CAF1 15057 117 - 1 --CGUAAACAGCAAAGCAUCGCCAUCAUGAACAUGAAUCUGCAAGAUAUCCAGCAGGAUAUCCAUAGGCUAGCAACUCAGCAAAGUCAAAUGCAGGCCCAACAUCUUCAGGCCCAGCAG --.............((...(((.((((....)))).((((((.(((((((....)))))))....((((.((......))..))))...)))))).............)))...)).. ( -29.40) >DroMoj_CAF1 28918 119 - 1 AAAAACAACAGCAAAGCAUUGCCAUCAUGAACAUGAAUCUGCAGGAUAUCCAGCAGGAUAUUCAUCGGCUGGCCACCCAGCAAAGCCAAAUGCAGGCGCAGCAUUUGCAAGCGCAGCAG ..........(((((((..((((.((((....))))...((((((((((((....))))))))....(((((....))))).........))))))))..)).)))))........... ( -37.30) >DroPer_CAF1 15936 115 - 1 --C--GUGCAGCAAAGCAUCGCCAUCAUGAACAUGAAUCUGCAGGAUAUCCAGCAGGAUAUCCACCGCCUGGCCACGCAGCAGAGCCAGAUGCAGGCCCAGCACCUGCAGGCGCAGCAG --.--.(((.((...(((((....((((....)))).(((((.((((((((....))))))))...((........)).)))))....)))))..(((..((....)).))))).))). ( -42.90) >consensus __C__AAACAGCAAAGCAUCGCCAUCAUGAACAUGAAUCUGCAGGAUAUCCAGCAGGAUAUCCACCGCCUGGCCACGCAGCAAAGCCAAAUGCAGGCCCAGCACCUGCAAGCGCAGCAG ..........((...((...(((.((((....))))...((((((((((((....))))))))......((((...........))))..)))))))...((....))..))...)).. (-27.00 = -27.37 + 0.36)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:08:09 2006