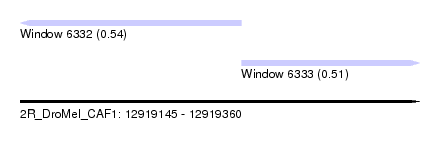
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 12,919,145 – 12,919,360 |
| Length | 215 |
| Max. P | 0.542184 |

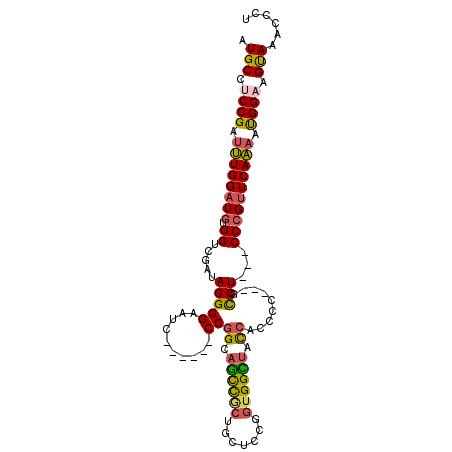
| Location | 12,919,145 – 12,919,264 |
|---|---|
| Length | 119 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | reverse |
| Mean pairwise identity | 81.57 |
| Mean single sequence MFE | -42.25 |
| Consensus MFE | -26.92 |
| Energy contribution | -26.23 |
| Covariance contribution | -0.69 |
| Combinations/Pair | 1.45 |
| Mean z-score | -1.59 |
| Structure conservation index | 0.64 |
| SVM decision value | 0.02 |
| SVM RNA-class probability | 0.542184 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
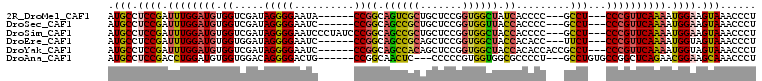
>2R_DroMel_CAF1 12919145 119 - 20766785 UCAUGGUACUGCCGCUCUGAAUGCUGCUGGCAUUUGAGGAUCGCUUGGUGCUCACCAUACUGAUGCUACUGAUAUUGGCACUUGCUCCGGGCAUCUCCUGUUUGGUGCCCUCGUAUCCA ....(((((((((.(((.((((((.....))))))))).((((..((((....))))...))))............))))........(((((((........)))))))..))))).. ( -37.30) >DroVir_CAF1 45322 119 - 1 UCAUUGUGCUGCCACUUUGAAUGCUGCUCGCAUUCGAGAUGCGCUUCGUGCUGGUCACGCUGAUGCUGCUGAUGUUGGCGCUGGCGCCGGGUAUCUCCUGCUUGGUGCCCUCAUAGCUG (((.((((..((((.(((((((((.....)))))))))..((((...)))))))))))).))).((((.(((....((((....))))(((((((........)))))))))))))).. ( -44.80) >DroGri_CAF1 41554 119 - 1 UCAUUGUGCUGCCACUUUGAAUGCUGCUCGCAUUCGAAGUCCGCUUUGUGCUGGUCACACUGAUGCUACUGAUAUUCGCACUGGCACCGGGCAACUCCUGCUUGGUGCCCUCAUAGCUC (((.((((..((((((((((((((.....)))))))))))..((.....)).))))))).))).((((.(((..........(((((((((((.....))))))))))).))))))).. ( -45.90) >DroWil_CAF1 74539 118 - 1 UCAUAGUGCUGCCACUUUGAAUGCUACUGGCAUUCGAUGAUCGUUUGGUACUGACCAUGCUAAUGCUACUUAUAUUGGCACUGGCACCGGGCAUCUCUUGUUUGGUACCCUCAUAGGC- ...(((((((((....((((((((.....)))))))).....))..))))))).((..((((.(((((.......))))).))))((((((((.....)))))))).........)).- ( -35.50) >DroMoj_CAF1 64671 119 - 1 UCAUUGUGCUGCCGCUCUGAGUGCUGCUCGCAUUGGAGACGCGCUUCAUGCUGGUCACACUGAUGCUGCUGAUGUUGGCGCUGGCGCCGGGCAGCUCCUGCUUGGUACCCUCAUAGCUG (((.((((..(((((...((((((..(((......)))..))))))...)).))))))).))).((((((......)))...((..((((((((...))))))))..)).....))).. ( -42.90) >DroAna_CAF1 42545 119 - 1 UCACGGUGCUGCCACUCUGGAUGCUGCUGGCGUUUGAGGAUCGCUUGGUGCUCACCAUGCUGAUGCUGCUUAUGUUGGCACUCGCUCCGGGCAUCUCCUGCUUGGUGCCCUCGUAUCCG ....((.....)).....(((((((((..((((..(((.(((((.((((....)))).)).))).)).)..))))..)))........(((((((........)))))))..)))))). ( -47.10) >consensus UCAUUGUGCUGCCACUCUGAAUGCUGCUCGCAUUCGAGGAUCGCUUGGUGCUGACCACACUGAUGCUACUGAUAUUGGCACUGGCACCGGGCAUCUCCUGCUUGGUGCCCUCAUAGCCG .........((((((((.((((((.....)))))))))....(((..(((.......)))..).)).........)))))..(((((((((((.....))))))))))).......... (-26.92 = -26.23 + -0.69)

| Location | 12,919,264 – 12,919,360 |
|---|---|
| Length | 96 |
| Sequences | 6 |
| Columns | 108 |
| Reading direction | forward |
| Mean pairwise identity | 83.93 |
| Mean single sequence MFE | -37.07 |
| Consensus MFE | -25.96 |
| Energy contribution | -25.88 |
| Covariance contribution | -0.08 |
| Combinations/Pair | 1.33 |
| Mean z-score | -1.82 |
| Structure conservation index | 0.70 |
| SVM decision value | -0.05 |
| SVM RNA-class probability | 0.508700 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 12919264 96 + 20766785 AUGCCUCCGAUUUGGAUGUGGUCGAUAGGGGAAUA------CCGGCAGUCGCUGCUCCGGUGGCUAUCACCCC---GCCU---CCCGUUCAAAAUGGAAGUAAACCCU .(((.((((.((((((((.((..(...((((..((------((((.(((....))))))))).......))))---.)..---)))))))))).)))).)))...... ( -35.50) >DroSec_CAF1 42597 96 + 1 AUGCCUCCGAUUUGGAUGUGGUCGAUAGGGGAAUC------CCGGCAGCCGCUGCUCCGGUGGUUACCACCCC---GCCU---CCCGUUCAAAAUGGAAGUAAACCCU .(((.((((.((((((((.((..(...((((((((------((((.(((....))))))).))))....))))---.)..---)))))))))).)))).)))...... ( -37.70) >DroSim_CAF1 30810 102 + 1 AUGCCUCCGAUUUGGAUGUGGUCGAUAGGGGAAUCCCUAUCCCGGCAGCCGCUGCUCCGGUGGCUACCACCCC---GCCU---CCCGUUCAAAAUGGAAGUAAACCCU .(((.((((.((((((((.(((.(((((((....)))))))..((.((((((((...)))))))).)).....---))).---..)))))))).)))).)))...... ( -41.90) >DroEre_CAF1 45348 96 + 1 AUGCCUCCGAUUUGGAUGUGGUGGAUAGGGGAAUC------CCGGCAGCCGCAGCUCCGGUGGCUACCACACC---UUCU---CCCGUUCAAAAUGGUAGUAAACCCU .(((..(((.((((((((.((.(((.(((((....------))((.((((((.......)))))).))...))---))))---)))))))))).)))..)))...... ( -34.80) >DroYak_CAF1 45083 99 + 1 AUGCCUCCGAUUUGGAUGUGGUCGAUAGGGGAAUC------CCGGCAGCCACAGCUCCGGUGGCUACCACACCACCGCCU---CCCGUUCAAAAUGGUAGUAAACCCU .(((..(((.((((((((.((......(((....)------))((((((....)))..(((((........)))))))).---)))))))))).)))..)))...... ( -34.60) >DroAna_CAF1 42664 96 + 1 AUGCCUCCGACCUGGAUGUGGUGGACAGGGGACUG------CCGGCAACUC---CCCCCGUGGUGGCGCCCCU---GCCUGUGCCGGCUCAGAACGGAAGCAAACCCU .(((.((((..((((...((((((.((((((...(------(((.((.(..---.....))).)))).)))))---)))...))))..))))..)))).)))...... ( -37.90) >consensus AUGCCUCCGAUUUGGAUGUGGUCGAUAGGGGAAUC______CCGGCAGCCGCUGCUCCGGUGGCUACCACCCC___GCCU___CCCGUUCAAAAUGGAAGUAAACCCU .(((.((((.((((((((.((.....(((((..........))((.((((((.......)))))).)).........)))...)))))))))).)))).)))...... (-25.96 = -25.88 + -0.08)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:07:23 2006