
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
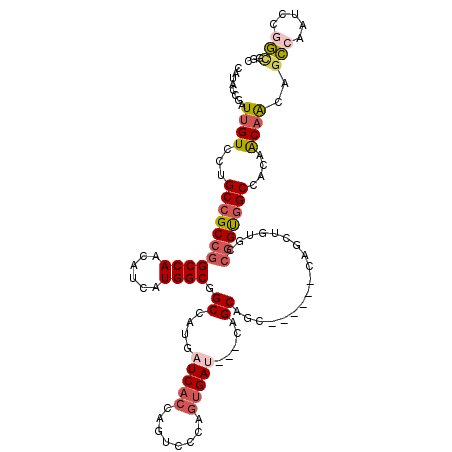
| Location | 12,888,771 – 12,888,879 |
| Length | 108 |
| Max. P | 0.736424 |

| Location | 12,888,771 – 12,888,879 |
|---|---|
| Length | 108 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 82.82 |
| Mean single sequence MFE | -41.58 |
| Consensus MFE | -22.45 |
| Energy contribution | -23.40 |
| Covariance contribution | 0.95 |
| Combinations/Pair | 1.20 |
| Mean z-score | -2.74 |
| Structure conservation index | 0.54 |
| SVM decision value | 0.13 |
| SVM RNA-class probability | 0.597030 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 12888771 108 + 20766785 CAUACCGAUUGUCCUGCCGCCGGCCAACAUCAUGGCGGCCAUGAUCACCAGUCCCAGUGAU---CAGCAGC------CAGCUGUGCCGGUGGCCACAACAACAGCCAAUCCGGCGGC ....(((.((((...(((((((((..(((...((((.((..(((((((........)))))---)))).))------))..)))))))))))).)))))....(((.....))))). ( -45.90) >DroPse_CAF1 11597 113 + 1 CAUACCCAUUGUCCUGCCGCCGGCCAACAUCAUGGCUGCCAUGAUCACAAAUCCAAACGAA---CAGCAACAGCAGGCCACUGCUGCAGCAGCUGCAGCUGCUGUCGGUCCCAAUU- .......((((....((((..((((...(((((((...)))))))................---..((....)).))))......((((((((....))))))))))))..)))).- ( -34.80) >DroSec_CAF1 12053 108 + 1 CAUACCGAUUGUCCUGCCGCCGGCCAACAUCAUGGCGGCCAUGAUCACCAGUCCCAGUGAU---CAGCAGC------CAGCUGUGCCGGUGGCCACAACAACUGCCAAUCCGGCGGC ......(.((((...(((((((((..(((...((((.((..(((((((........)))))---)))).))------))..)))))))))))).)))))..(((((.....))))). ( -46.80) >DroEre_CAF1 11895 108 + 1 CAUACCGAUUGUCCUGCCGCCGGCCAACAUCAUGGCGGCCAUGAUCACCAGUCCCAGUGAU---CAGCAGC------CAGCUGUGCCGGUGGCCACAACAACAGCCAAUCCGGUAGC ..(((((((((..(((((((((((..(((...((((.((..(((((((........)))))---)))).))------))..)))))))))))).........)).)))).))))).. ( -45.00) >DroYak_CAF1 13202 108 + 1 CAUACCGAUUGUCCUGCCGCCGGCCAACAUCAUGGCGGCCAUGAUCACCAGUCCCAGUGAU---CAGCAGG------CAGCUGUCCCGGUGGCCACAACAACAGCCAAUCCGGCGGC ...............(((((((((((......))))((((((((((((........)))))---).((((.------...))))....))))))................))))))) ( -41.80) >DroAna_CAF1 12155 105 + 1 UAUACCGAUUGUCCUGCCGCCAGCCAACAUCAUGGCGGCCAUGAUCACCAGUCCCAGUGACGCCCAGCAGC------CGG------CGGUGGCCACCACAGCCGCCAAUCCUGCAGC ......(((..(...(((((((..........))))))).)..)))....((..(((.((.(((.......------.))------)((((((.......))))))..)))))..)) ( -35.20) >consensus CAUACCGAUUGUCCUGCCGCCGGCCAACAUCAUGGCGGCCAUGAUCACCAGUCCCAGUGAU___CAGCAGC______CAGCUGUGCCGGUGGCCACAACAACAGCCAAUCCGGCGGC ........((((...(((((((((((......)))).((....(((((........))))).....))..................)))))))....))))..(((.....)))... (-22.45 = -23.40 + 0.95)

| Location | 12,888,771 – 12,888,879 |
|---|---|
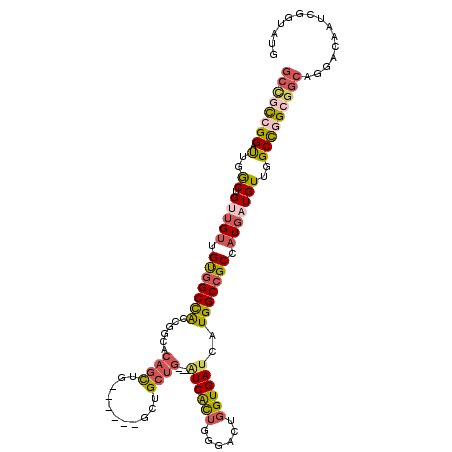
| Length | 108 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 82.82 |
| Mean single sequence MFE | -50.57 |
| Consensus MFE | -33.99 |
| Energy contribution | -35.17 |
| Covariance contribution | 1.17 |
| Combinations/Pair | 1.36 |
| Mean z-score | -2.35 |
| Structure conservation index | 0.67 |
| SVM decision value | 0.44 |
| SVM RNA-class probability | 0.736424 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
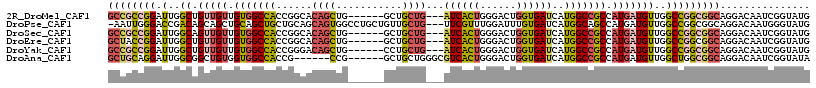
>2R_DroMel_CAF1 12888771 108 - 20766785 GCCGCCGGAUUGGCUGUUGUUGUGGCCACCGGCACAGCUG------GCUGCUG---AUCACUGGGACUGGUGAUCAUGGCCGCCAUGAUGUUGGCCGGCGGCAGGACAAUCGGUAUG ((((((((.(..((.((..(.((((((((((((...))))------)....((---((((((......))))))))))))))).)..))))..)))))))))............... ( -54.70) >DroPse_CAF1 11597 113 - 1 -AAUUGGGACCGACAGCAGCUGCAGCUGCUGCAGCAGUGGCCUGCUGUUGCUG---UUCGUUUGGAUUUGUGAUCAUGGCAGCCAUGAUGUUGGCCGGCGGCAGGACAAUGGGUAUG -((((.((((.(((((((((.(((((..(((...)))..).)))).)))))))---)).)))).))))....(((((((...)))))))(((.(((...)))..))).......... ( -43.80) >DroSec_CAF1 12053 108 - 1 GCCGCCGGAUUGGCAGUUGUUGUGGCCACCGGCACAGCUG------GCUGCUG---AUCACUGGGACUGGUGAUCAUGGCCGCCAUGAUGUUGGCCGGCGGCAGGACAAUCGGUAUG ((((((((.(..(((.(..(.((((((((((((...))))------)....((---((((((......))))))))))))))).)..))))..)))))))))............... ( -55.10) >DroEre_CAF1 11895 108 - 1 GCUACCGGAUUGGCUGUUGUUGUGGCCACCGGCACAGCUG------GCUGCUG---AUCACUGGGACUGGUGAUCAUGGCCGCCAUGAUGUUGGCCGGCGGCAGGACAAUCGGUAUG ..(((((.((((.(((((((((.((((((((((...))))------)....((---((((((......)))))))))))))((((......)))))))))))))..))))))))).. ( -51.00) >DroYak_CAF1 13202 108 - 1 GCCGCCGGAUUGGCUGUUGUUGUGGCCACCGGGACAGCUG------CCUGCUG---AUCACUGGGACUGGUGAUCAUGGCCGCCAUGAUGUUGGCCGGCGGCAGGACAAUCGGUAUG ((((((((.(..((.((..(.(((((((.((((.(....)------)))).((---((((((......))))))))))))))).)..))))..)))))))))............... ( -52.60) >DroAna_CAF1 12155 105 - 1 GCUGCAGGAUUGGCGGCUGUGGUGGCCACCG------CCG------GCUGCUGGGCGUCACUGGGACUGGUGAUCAUGGCCGCCAUGAUGUUGGCUGGCGGCAGGACAAUCGGUAUA (((((.((.(..(((..(((((((((((.((------(((------(.(.(..(......)..).)))))))....))))))))))).)))..))).)))))............... ( -46.20) >consensus GCCGCCGGAUUGGCUGUUGUUGUGGCCACCGGCACAGCUG______GCUGCUG___AUCACUGGGACUGGUGAUCAUGGCCGCCAUGAUGUUGGCCGGCGGCAGGACAAUCGGUAUG ((((((((.(..((.(((((.(((((((......((((...........))))...((((((......))))))..))))))).)))))))..)))))))))............... (-33.99 = -35.17 + 1.17)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:07:07 2006